Introduction
- 그동안 객체 인식 분야에서 컨볼루션 네트워크를 활용한 수많은 모델들이 있었지만 train dataset과 네트워크의 크기에 영향을 받아 실질적인 사용이 제한적이었다.
- 따라서, 본 논문에서는 적은 데이터로도 학습이 가능한 여러가지 기법들을 소개한다.
- 본 논문은 biomedical 이미지에 대해 중점적으로 다루며 픽셀 단위의 분류가 필요하므로 segmentation으로 연결된다.
- FCN의 구조를 수정, 확장하여 더 적은 데이터로부터 더 정확한 예측을 가능하게 하였다.
-> contracting path에서의 feature map을 upsampling에서의 feature map에 더해주어 정보를 전달한다.
<기존 연구의 단점>
- sliding window방식으로 patch를 뽑아 각 patch별로 네트워크를 거치며, 중복되는 부분들이 많아 느리다.
- 큰 patch를 사용할 경우 max pooling이 늘어나 localiztion의 성능이 떨어지고, 작은 patch를 사용할 경우 context를 많이 담을 수 없다는 trade-off가 있다.
Architecture
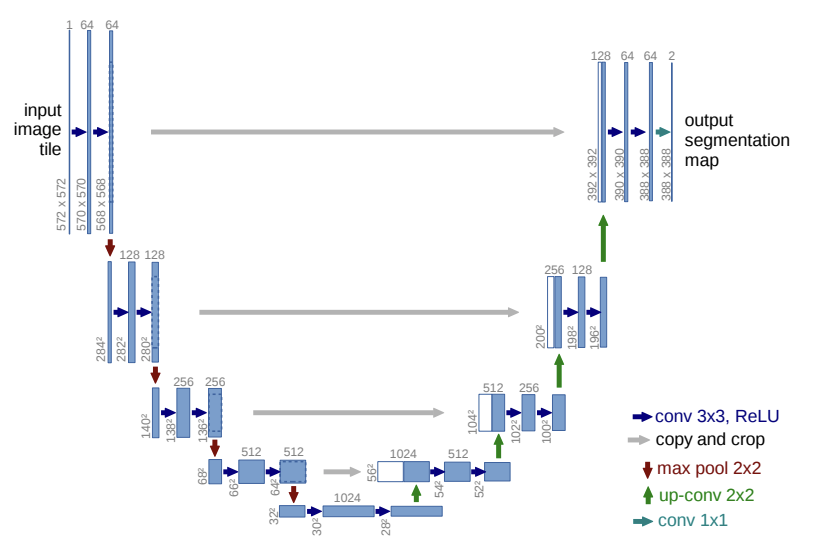
Contracting path
- 그림의 좌측에 해당하는 부분으로 일반적인 컨볼루션 네트워크를 활용한 DownSampling 과정이다. 의미정보(Context information)를 추출한다.
- Unpadding 3x3 필터를 활용하여 feature map의 크기가 감소하며 max pooling도 함께 활용한다.
- DownSampling을 할 때마다 채널의 수를 2배 증가시킨다.
Expansive path
- 그림의 우측에 해당하는 부분으로 Upsampling 과정이다. 위치정보(Localization)를 추출한다.
- 2x2 Deconvolution 레이어를 활용하며 채널수를 반씩 줄여나간다.
- contracting path에서의 feature map과 concatenation을 하여 expansive path의 의미정보를 전달한다.
- 최종적으로 로 출력한다.
Methods
Upsampling larger channels
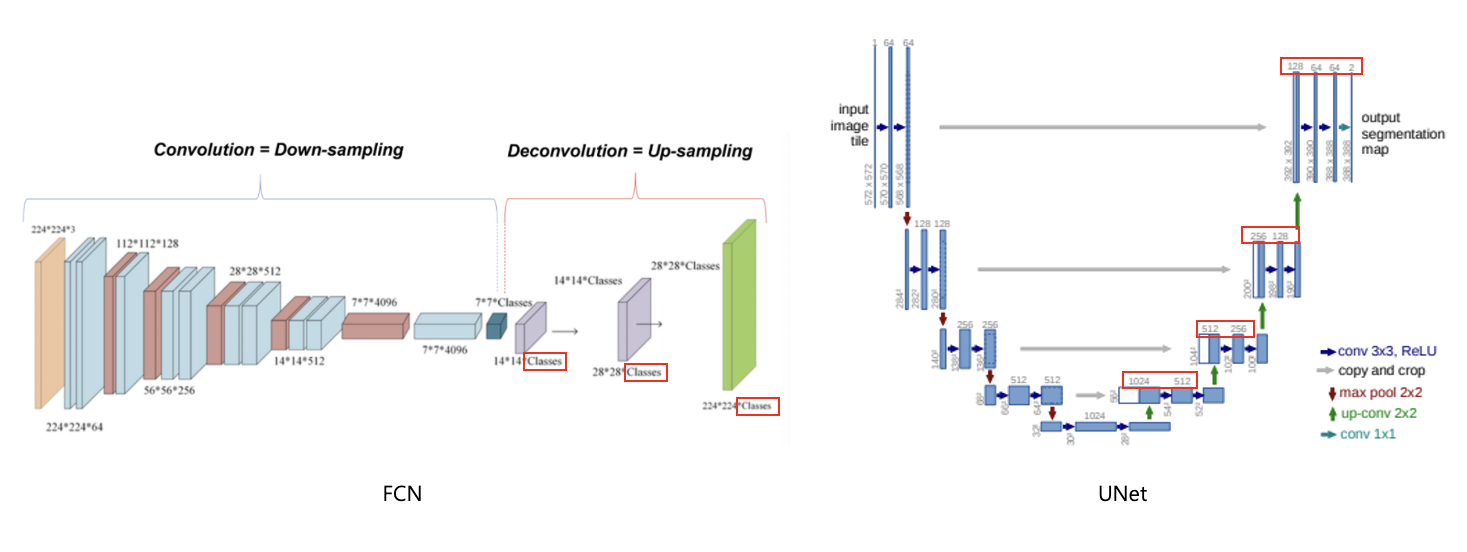
UNet에서는 upsampling 시에도 높은 채널수를 유지하였다. 이를 통해 context information을 높은 해상도에서도 이어 받을 수 있다.
Mirroring extrapolate
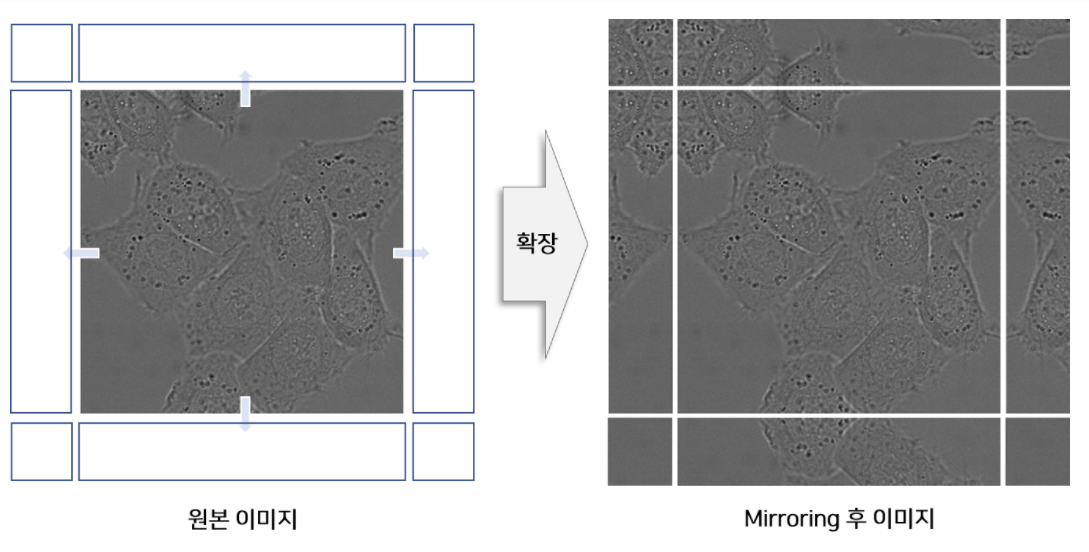
UNet에서 사용하는 Biomedical 이미지들의 특성상 테두리에 있는 부분도 동일하게 중요하다. 따라서 본 논문에서는 테두리의 정보도 활용하기 위해 미러링을 통해 패딩을 해주었다. 미러링 패딩을 쓰면 테두리의 픽셀들을 반사시킨 형태로 채워주게 되는데 biomedical 분야에서 주로 등장하는 세포가 대칭구도를 이루는 경우가 많기 때문으로 추측된다고 한다.
Overlap-tile strategy
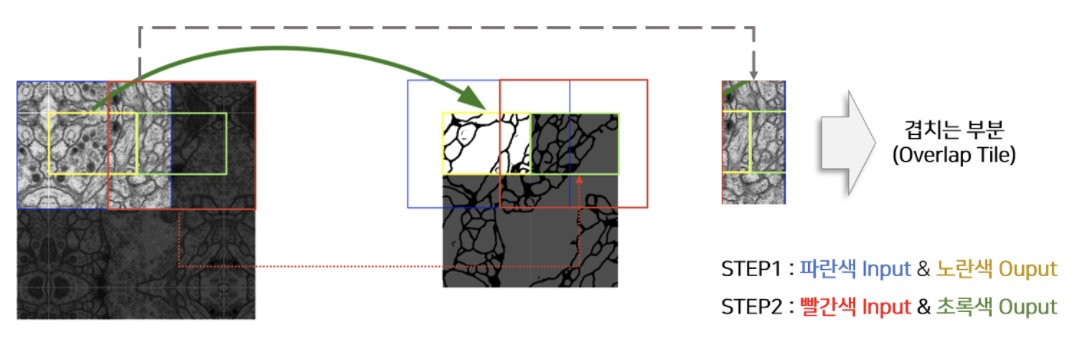
패딩을 이용하며 output 사이즈가 input 사이즈에 비해 작기 때문에 겹치는 부분이 존재하도록 patch를 구성해야 원본 크기의 output을 얻을 수 있다. 위 그림에서 파란색 patch를 input으로 넣으면 노란색 output이, 빨간색 patch를 input으로 넣으면 초록색 output이 나와 연결되는 것을 알 수 있다.
Weight Loss
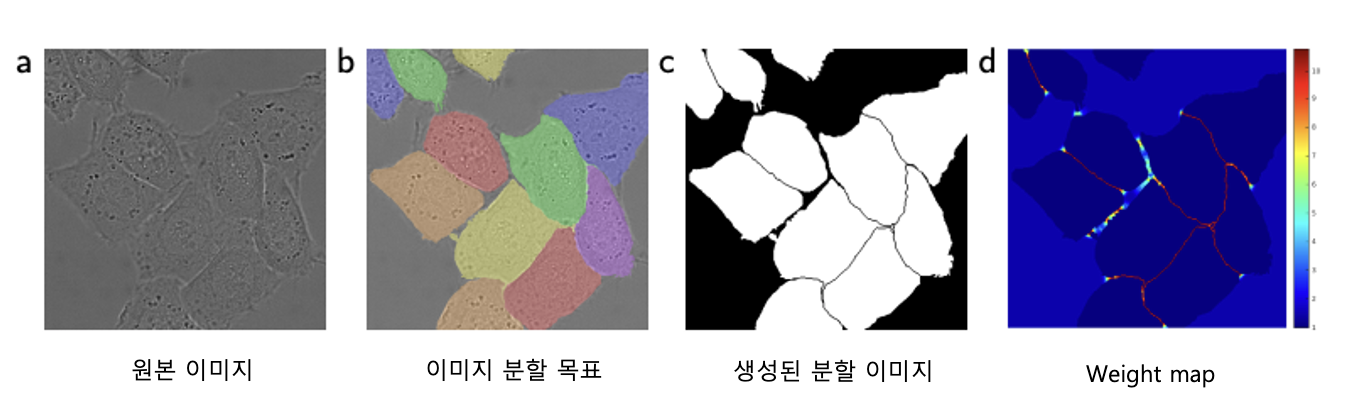
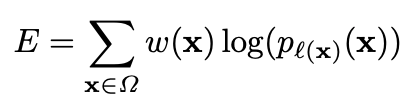
Segmentation에서는 동일 클래스의 객체를 구분하기 어렵다는 문제가 있는데 본 논문에서는 weighted loss를 통해 해결하였다. UNet의 loss function은 위와 같이 pixel-wise cross entropy loss로 계산하며 경계와 가까운 픽셀들에는 높은 가중치를 부여하여 학습을 세밀하게 할 수 있도록 만들어주었다.
: 클래스의 수
: , 실제 라벨 집합
: 픽셀 가 k클래스일 확률(softmax)
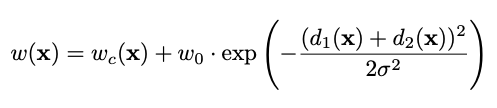
가중치()는 위와 같이 계산된다. 그림(d)에서 볼 수 있듯이 경계에 있을수록 높은 값을 가진다.
: weight map to balance the class frequencies.
: 하이퍼파라미터로 논문에서는 10으로 설정
: 하이퍼파라미터로 논문에서는 5로 설정
: 픽셀 에서 가장 가까운 셀과의 거리
: 픽셀 에서 두번째로 가까운 셀과의 거리
Data Augmentation

본 논문에서는 Data augmentation을 적절히 활용하여 객체의 불변하는 고유한 특성들을 잘 뽑아낼 수 있게 만들었다. Rotation, Shift, Elastic distortion 등을 활용하였으며 특히 Elastic distortion은 픽셀별로 이미지가 다른 방향으로 뒤틀리게 만드는 방법으로 성능 향상에 효과적이다. Biomedical data의 경우 살아있는 것을 관찰한 데이터이기 때문에 순간순간의 미세한 변형을 주어 자연스러운 변화에 강건한 모델을 만들 수 있도록 한다.
Experiments
EM Segmentation challenge
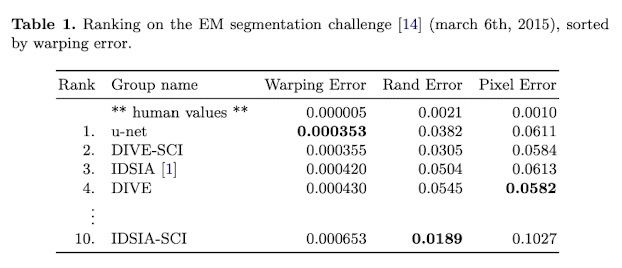
30개의 세포 구조 Training 데이터셋과 Ground truth segmentation map(객체와 배경을 0,1로 분류)이 제공된다. 해당 데이터셋에서 가장 낮은 warping error를 보여 적은 데이터셋으로 좋은 성능을 보이고 있음을 증명한다.
ISBI cell tracking challening
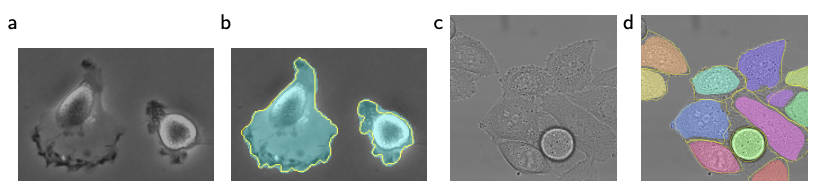
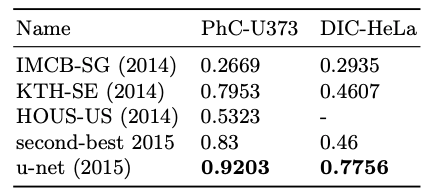
세포 분류 대회에서 제공되는 데이터로 (a),(b)가 'PhC-U373" (c),(d)가 'DIC-HeLa'이다. 두 데이터셋에서 모두 UNet이 가장 높은 IOU score를 기록하였으며 2등과의 격차가 굉장히 큰 것을 볼 수 있다.
