머신러닝으로 AI 모델링하기
선형회귀
선형회귀 알고리즘
= 독립변수
= 가중치
= 종속변수
데이터는 이고, 에 입력할 데이터는 1 ~ 10이다. 가 로 출력된 결과이기에 해당 데이터를 학습한 모델이 핵심 숫자 4와 7를 기울기 및 절편으로 지나는 모델이라면 학습이 잘 된 모델이라고 할 수 있다.
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LinearRegression
# 랜덤 시드 고정하기
np.random.seed(2023)
# x 는 1 ~10, y는 4x+7인 학습 데이터 생성
x = []
y = []
for i in range(1,11):
x.append(i)
y.append(4*i + 7)
# 학습시키기
# 선형회귀 생성
reg = LinearRegression()
#학습을 위해 1행 10열 구성을 10행 1열 구성으로 형태 변환
x = np.array(x).reshape(-1,1)
#학습
reg.fit(x,y)
# 산식 추정을 위한 기울기(coef)와 절편(intercept) 확인하기
print(f'''기울기 및 절편 확인 coef = {reg.coef_}, intercept = {reg.intercept_}''')
# 절편과 기울기로 결과를 수동 계산하여 비교
coef_intercept = x * reg.coef_ + reg.intercept_
print(coef_intercept)
#scatter plot 그래프 그리기
plt.scatter(x,y, color = 'r', s = 20)
#선 그래프 그리기
plt.plot(x, coef_intercept, color = 'blue')
#coef, intercept 값 그래프 내에 텍스트 삽입
plt.text(8,20, 'coef = %.0f'%reg.coef_, size=12)
plt.text(8,18, 'intercept = %.0f'%reg.intercept_, size=12)
#라벨설정
plt.xlabel('x')
plt.ylabel('y')
plt.show()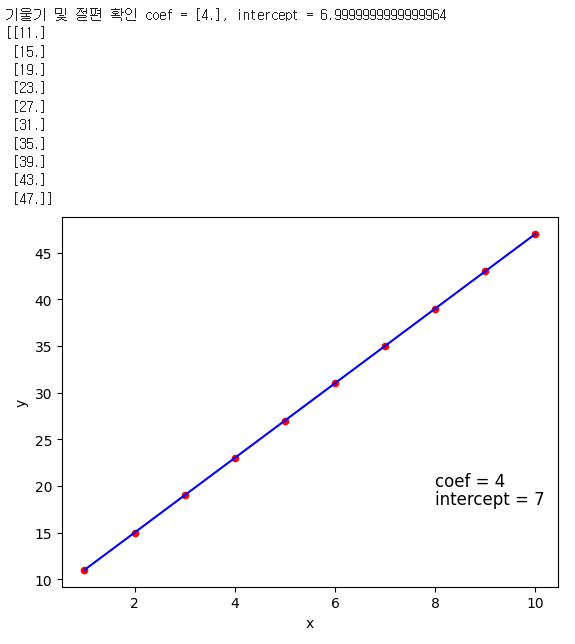
예제
데이터 전처리
데이터의 분석 목적에 맞게끔 필요하지 않는 데이터를 제외시키고 이상치와 결측지를 처리한다음
학습 데이터와 정답데이터를 만들어야한다.
데이터분석하고자 하는 모델은 선형회귀 모델로 지도학습에 해당됨으로 정답데이터를 만들어야한다.
import pandas as pd
from sklearn.model_selection import train_test_split
df = pd.read_csv('/content/drive/MyDrive/Colab Notebooks/국민건강보험공단_건강검진정보_20211229.CSV', encoding='cp949')
# display 옵션을 통한 전체 열 확장
pd.set_option('display.max_columns', None)
# 콜레스테롤 수치는 치아, 시력, 청력과 관계가 없다는 가설로 데이터 일부 삭제
df.drop(['치아우식증유무','치석','구강검진 수검여부','시력(좌)','시력(우)','청력(좌)','청력(우)','기준년도'],inplace=True, axis=1)
# 불필요한 데이터 추가 삭제
df.drop(['가입자 일련번호','데이터 공개일자','성별코드','시도코드'], axis = 1, inplace=True)
#테스트 데이터 추출
test = df[df['LDL 콜레스테롤'].isnull()]
#NaN 데이터 행 단위로 삭제
train = df.dropna(axis=0)
# 정답 데이터 생성하기
y = train['LDL 콜레스테롤']
#학습 데이터 생성하기
x = train.drop('LDL 콜레스테롤', axis = 1)
#사이킷런 예시 코드 비율대로 불러오기
x_train, x_test, y_train, y_test = train_test_split(x,y,test_size=0.33, random_state=42)
print('학습 / 검증 데이터 확인')
print(x_train.shape, y_train.shape, x_test.shape, y_test.shape)
선형 회귀 학습 및 추론
사이킷 라이브러리를 통해서 데이터를 학습을 시킨다.
from sklearn.linear_model import LinearRegression
# 모델 생성하기
reg = LinearRegression()
#학습하기
reg.fit(x_train, y_train)
# 각 데이터에 대해 가중치 확인
print('전체에 대해 가중치(기울기) 확인')
for index, columns in enumerate(x_train.columns) :
print(f'{columns} = {reg.coef_[index]}')
예측을 통한 최종 검증
결과 검증을 위해 MSE 라이브러리를 사용한다.
#예측하기
y_pred = reg.predict(x_test)
#결과 검증을 위해 MSE 라이브러리 불러오기
#최종적으로는 RMSE를 사용하기
from sklearn.metrics import mean_squared_error
# MSE 라이브러리에서 RMSE는 squared 옵션을 False로 설정하기
rmse = mean_squared_error(y_test, y_pred, squared = False)
print(f'주요 Feature 삭제 전 rmse = {round(rmse,3)}')
앞선 결과를 보게되면 음주여부와 HDL콜레스테롤, 총 콜레스테롤이 가장 많이 영향을 주는 것을 알 수 있다.
가중치의 의미 확인
가중치가 큰 콜레스테롤 관련 Feature를 삭제 한 후 독립변수의 가중치가 어떻게 변할까?
# 주요 Feature 삭제
x = x.drop(['총 콜레스테롤', 'HDL 콜레스테롤', '음주여부'], axis = 1)
#사이킷런 예시 코드 비율대로 불러오기
x_train, x_test, y_train, y_test = train_test_split(x,y,test_size=0.33, random_state=42)선형 회귀 모델 재학습하기
# 선형 회귀 재생성 및 학습하기
lr = LinearRegression()
lr.fit(x_train, y_train)
# 각각 데이터에 대해 가중치 확인
print(f'''
coef
{lr.coef_}
intercept
{lr.intercept_}
''')
가중치 비교하기
# 검증 데이터로 예측하기
y_pred = lr.predict(x_test)
print('가중치 확인')
for index, columns in enumerate(x_train.columns) :
print(f'{columns} = {reg.coef_[index]}')
# 주요 Feature 삭제 후 RMSE 확인하기
rmse_2 = mean_squared_error(y_test, y_pred, squared = False)
# 가중치가 큰 특성을 삭제 했을 때 영향을 많이 받는지 확인
print(f'''주요 Feature 삭제 후 rmse = {round(rmse_2,3)}''')
