4장. 좋은 훈련 데이터셋 만들기: 데이터 전처리

💡 좋은 머신 러닝 모델을 구축하는 데 도움이 되는 핵심적인 전처리 기법 다루기
- 데이터셋에서 누락된 값을 제거하거나 대체하기
- 머신 러닝 알고리즘을 위해 범주형 데이터 변환하기
- 모델과 관련이 높은 특성 선택하기
누락된 데이터 다루기
-
데이터 수집 과정에 오류가 있거나 어떤 측정 방법은 적용이 불가능 할 수 있다
-
설문에서 특정 필드가 그냥 비워져 있을 수도 있다
-
일반적으로 누락된 갓은 데이터 테이블에 빈 공간이나 예약된 문자열로 채워진다
NaN 이나 Null
-
대부분의 수치 계산 라이브러리는 누락된 값을 다룰 수 없거나 단순히 이를 무시했을 때 예상치 못한 결과를 만든다
📍 테이블 형태 데이터에서 누락된 값 식별
import pandas as pd
from io import StringIO💡 StringIO: 하드 디스크에 있는 일반 CSV파일처럼 csv_data에 저장된 문자열을 읽어 판다스 DataFrame으로 변환할 수 있다
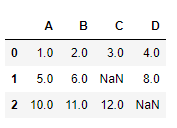
csv_data = '''
A,B,C,D
1.0, 2.0, 3.0, 4.0
5.0, 6.0,, 8.0
10.0, 11.0, 12.0,
'''
df = pd.read_csv(StringIO(csv_data))
df
~~>
📍 isnull 메소드를 사용해 셀에 Null 값이 있는지 확인
df.isnull().sum()
~~>A 0
B 0
C 1
D 1
dtype: int64
📍 누락된 값이 있는 훈련 샘플이나 특성 제외
- 데이터셋에서 해당 훈련 샘플( 행 )이나 특성( 열 )을 완전히 삭제하는 것
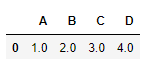
# 누락된 값이 있는 행 제거
df.dropna(axis=0)
~~>
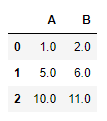
# 누락된 값이 있는 열 제거(axis = 1)
df.dropna(axis=1)
~~>
# 모든 열이 NaN일 때만 행을 제거
df.dropna(how='all')
~~>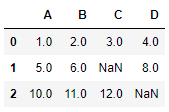
👉 모든 값이 NaN인 행이 없기 때문에 전체 배열이 반환
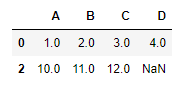
# 특정 열에 NaN이 있는 행만 삭제
df.dropna(subset=['C']) # 'C' 열에 NaN이 있으면 제거
~~>
💡 누락된 데이터를 제거하는 것이 간단해 보이지만 단점이 존재한다
- 너무 많은 데이터를 제거하면 안정된 분석이 불가능 할 수 있다
- 너무 많은 특성 열을 제거하면 분류기가 클래스를 구분하는 데 필요한 중요한 정보를 잃을 위험이 있다
📍 누락된 값 대체
- 보간 기법을 사용하여 다른 훈련 샘플로부터 누락된 값을 추정
- 가장 흔히 사용하는 보간 기법은 평균으로 대체하는 것
- 사이킷런의 SimpleImputer 클래스를 사용하면 간단하게 처리 가능
from sklearn.impute import SimpleImputer
import numpy as npimr = SimpleImputer(missing_values=np.nan, strategy='mean')
imr = imr.fit(df.values)
imputed_data = imr.transform(df.values)
imputed_data
~~>
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 7.5, 8. ],
[10. , 11. , 12. , 6. ]])매개변수 strategy 설정 값
- mean: 평균
- median: 중간값
- most_frequent: 가장 많이 나타난 값
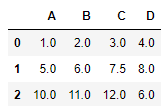
# fillna 메서드를 사용해 누락된 값 채우기
df.fillna(df.mean())
~~>
fillna 매서드
- bfill( = backfill ) : 누락된 값을 다음 행의 값으로 채움
- ffill( = pad ) : 이전 행의 값으로 채움
- 위의 두 매개변수에 axis = 1로 설정하면 열을 기준으로 바뀐다
범주형 데이터 다루기
- 범주형 데이터에 관해 이야기할 때 순서가 있는 것과 없는 것을 구분해야 한다
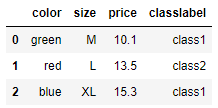
df = pd.DataFrame([
['green', 'M', 10.1, 'class1'],
['red', 'L', 13.5, 'class2'],
['blue', 'XL', 15.3, 'class1']
])
df.columns = ['color', 'size', 'price', 'classlabel']
df
~~>
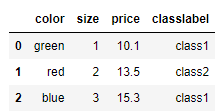
📍 순서가 있는 특성 매핑
- 범주형의 문자열 값을 정수로 바꿔주어야 한다
size_mapping = {
'XL' : 3,
'L' : 2,
'M' : 1
}
df['size'] = df['size'].map(size_mapping)
df
~~>
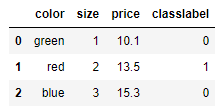
📍 클래스 레이블 인코딩
class_mapping = {label:idx for idx, label in enumerate(np.unique(df['classlabel']))}
class_mapping
~~>
{'class1': 0, 'class2': 1}df['classlabel'] = df['classlabel'].map(class_mapping)
df
~~>
# 다시 원본 문자열로 바꿀경우
# 다시 원본 문자열로 바꿀경우
inv_class_mapping = {v: k for k, v in class_mapping.items()}
df['classlabel'] = df['classlabel'].map(inv_class_mapping)
df
~~>
# 사이킷런에 구현된 LabelEncoder 클래스를 사용해도 된다
from sklearn.preprocessing import LabelEncoder
class_le = LabelEncoder()
y = class_le.fit_transform(df['classlabel'].values)
y
~~>
array([0, 1, 0])# 정수 클래스 레이블을 원본 문자열 형태로 되돌릴때
class_le.inverse_transform(y)
~~>
array(['class1', 'class2', 'class1'], dtype=object)📍 순서가 없는 특성에 원-핫 인코딩 적용
x = df[['color', 'size', 'price']].values
color_le = LabelEncoder()
x[:, 0] = color_le.fit_transform(x[:, 0])
x
~~>
array([[1, 1, 10.1],
[2, 2, 13.5],
[0, 3, 15.3]], dtype=object)
- blue : 0
- green : 1
- red : 2
- 이런 형식으로 데이터를 정리하면
blue > green > red의 형식이 되어 버린다 - 이러한 문제를 해결하기 위해 사용하는 방법이 원-핫 인코딩
from sklearn.preprocessing import OneHotEncoder
x = df[['color', 'size', 'price']].values
color_ohe = OneHotEncoder()
color_ohe.fit_transform(x[:, 0].reshape(-1, 1)).toarray()
~~>
array([[0., 1., 0.],
[0., 0., 1.],
[1., 0., 0.]])- 특정 열만 변환하려면 ColumnTransformer를 사용
from sklearn.compose import ColumnTransformer
x = df[['color', 'size', 'price']].values
c_transf = ColumnTransformer([
('onehot', OneHotEncoder(), [0]),
('nothing', 'passthrough', [1, 2])
])
c_transf.fit_transform(x)
~~>
array([[0.0, 1.0, 0.0, 1, 10.1],
[0.0, 0.0, 1.0, 2, 13.5],
[1.0, 0.0, 0.0, 3, 15.3]], dtype=object)데이터셋을 훈련 데이터셋과 테스트 데이터셋으로 나누기
UCI 머신 러닝 저장소에 있는 Wine 공개 데이터셋을 사용
df_wine = pd.read_csv('https://archive.ics.uci.edu/ml/'
'machine-learning-databases/wine/wine.data',
header=None)
df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue', 'OD280/OD315 OF diluted wines',
'Proline']
print('클래스 레이블', np.unique(df_wine['Class label']))
df_wine.head()
~~>
클래스 레이블 [1 2 3]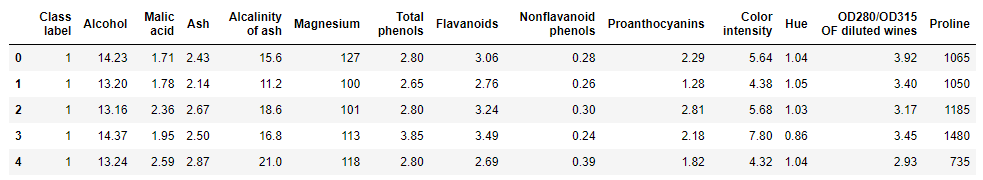
📍 랜덤한 훈련 데이터셋과 테스트 데이터셋으로 나누기
- 사이킷런의 model_selection 모듈에 있는 train_test_split 함수를 이용
from sklearn.model_selection import train_test_split
x, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.3,
random_state=0, stratify=y)특성 스케일 맞추기
정규화
- 대부분의 정규화는 특성의 스케일을
[0, 1]범위에 맞추는 것을 의미
📍 최소-최대 스케일 변환( Min-Max Scaling )
from sklearn.preprocessing import MinMaxScaler
mms = MinMaxScaler()
x_train_norm = mms.fit_transform(x_train)
x_test_norm = mms.transform(x_test)표준화
- : 어떤 특성의 샘플 평균
- : 그에 해당하는 표준 편차
📍 표준화 클래스
from sklearn.preprocessing import StandardScaler
stdsc = StandardScaler()
x_train_std = stdsc.fit_transform(x_train)
x_test_std = stdsc.transform(x_test)📍 표준화와 정규화
ex = np.array([0, 1, 2, 3, 4, 5])
print('표준화: ', (ex-ex.mean()) / ex.std())
print('정규화: ', (ex-ex.min()) / (ex.max()) - ex.min())
~~>
표준화: [-1.46385011 -0.87831007 -0.29277002 0.29277002 0.87831007 1.46385011]
정규화: [0. 0.2 0.4 0.6 0.8 1. ]💡 사이킷런에서 특성 스케일을 조정하는 다른 좋은 방법은 RobustScaler
- 이상치가 많이 포함된 작은 데이터셋을 다룰 때 특히 도움이 된다
- 데이터셋에 적용된 머신 러닝 알고리즘이 과대적합되기 쉽다면 좋은 선택
- 특성 열 마다 독립적으로 작용하며 중간 값을 뺀 다음 데이터셋의 1사분위수와 3사분위수를 사용해서 데이터셋의 스케일을 조정
- 극단적인 값과 이상치에 영향을 덜 받는다
유용한 특성 선택
✨ 모델이 테스트 데이터셋보다 훈련 데이터셋에서 성능이 훨씬 높다면 과대적합( Overfitting )에 대한 신호
과대적합의 이유
- 주어진 훈련 데이터에 비해 모델이 너무 복잡하기 때문
과대적합을 해결하기 위한 방법
- 더 많은 훈련 데이터를 모은다
- 규제를 통해 복잡도를 제한한다
- 파라미터 개수가 적은 간단한 모델을 선택한다
- 데이터 차원을 줄인다
모델 복잡도 제한을 위한 L1 규제와 L2 규제
L2 규제 : 개별 가중치 값을 제한하여 모델 복잡도를 줄이는 한 방법
- 비용 함수에 페널티 항을 추가해 규제가 없는 비용 함수로 훈련한 모델에 비해 가중치 값을 아주 작게 만드는 효과를 만든다
- 원 모양으로 제한 범위를 그릴 수 있다
- 모델을 학습할 만한 충분한 훈련 데이터가 없을 때 편향을 추가하여 모델을 간단하게 만듦으로써 분산을 줄이는 것
L1 규제 : L2 규제와 비슷하지만 가중치 절댓값의 합을 제한
- 다이아몬드 모양의 제한 범위를 그릴 수 있다
from sklearn.linear_model import LogisticRegression
LogisticRegression(solver='liblinear', penalty='l1')
~~>
LogisticRegression(penalty='l1', solver='liblinear')💡 LogisticRegrssion 클래스의 solver 중 1bfgs, newton-cg, sag는 L2 규제만 지원
saga, liblinear는 L1, L2 규제를 모두 지원
lr = LogisticRegression(solver='liblinear', penalty='l1', C=1.0, random_state=1)
# 규제 효과를 높이거나 낮추려면 C값을 증가시키거나 감소시킨다( C = 1.0 이 기본 값)
lr.fit(x_train_std, y_train)
print('훈련 정확도: ', lr.score(x_train_std, y_train))
print('테스트 정확도: ', lr.score(x_test_std, y_test))
~~>
훈련 정확도: 1.0
테스트 정확도: 1.0
lr.intercept_
~~>
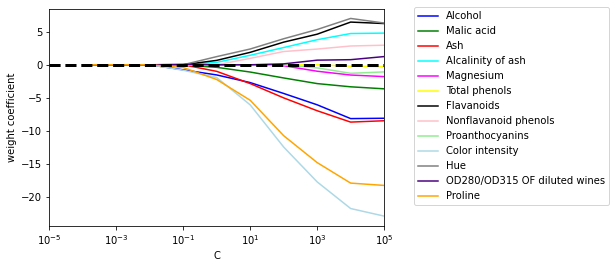
array([-1.26392152, -1.21596534, -2.37040177])📍 규제 강도를 달리하여 특성의 가중치 변화를 그래프로 표현
import matplotlib.pyplot as plt
fig = plt.figure()
ax = plt.subplot(111)
colors = ['blue', 'green', 'red', 'cyan', 'magenta',
'yellow', 'black', 'pink', 'lightgreen',
'lightblue', 'gray', 'indigo', 'orange']
weights, params = [], []
for c in np.arange(-4., 6.):
lr = LogisticRegression(solver='liblinear', penalty='l1',
C=10.**c, random_state=0)
lr.fit(x_train_std, y_train)
weights.append(lr.coef_[1])
params.append(10**c)
weights = np.array(weights)
for col, color in zip(range(weights.shape[1]), colors):
plt.plot(params, weights[:, col],
label=df_wine.columns[col + 1],
color = color)
plt.axhline(0, color='black', linestyle='--', linewidth=3)
plt.xlim([10**(-5), 10**5])
plt.ylabel('weight coefficient')
plt.xlabel('C')
plt.xscale('log')
plt.legend(loc='upper left')
ax.legend(loc='upper center', bbox_to_anchor=(1.38, 1.03),
ncol=1, fancybox=True)
plt.show()
~~>
순차 특성 선택 알고리즘
✨ 모델 복잡도를 줄이고 과대적합을 피하는 다른 방법은 특성 선택을 통한 차원 축소
- 규제가 없는 모델에서 특히 유용
차원 축소 기법
특성 선택( feature selection )
- 원본 특성에서 일부를 선택
순차 특성 선택 알고리즘
- 탐욕적 탐색 알고리즘
- 초기 차원의 특성 공간을 인 차원의 특성 부분 공간으로 축소
- 주어진 문제에 가장 관련이 높은 특성 부분 집합을 자동으로 선택하는 것이 목적
- 관계없는 특성이나 잡음을 제거하여 계산 효율성을 높이고 모델의 일반화 오차를 줄인다
- 전통적인 순차 특성 선택 알고리즘은 순차 후진 선택( Sequential Backwoad Selection, SBS )
- 새로운 특성의 부분 공간이 목표하는 특성 개수가 될 때까지 전체 특성에서 순차적으로 특성을 제거
- 각 단계에서 어떤 특성을 제거할지 판단하기 위해 최대화할 기준 함수를 정의
👉 각 단계에서 제거했을 때 성능 손실이 최소가 되는 특성을 제거
특성 추출( feature extraction )
- 일련의 특성에서 얻은 정보로 새로운 특성을 만든다
📍 순차 후진 선( SBS )택 알고리즘 구현
from sklearn.base import clone
from itertools import combinations
import numpy as np
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
class SBS():
def __init__(self, estimator, k_features, scoring=accuracy_score,
test_size=0.25, random_state=1):
self.scoring = scoring
self.estimator = clone(estimator)
self.k_features = k_features
self.test_size = test_size
self.random_state = random_state
def fit(self, x, y):
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size = self.test_size,
random_state = self.random_state)
dim = x_train.shape[1]
self.indices_ = tuple(range(dim))
self.subsets_ = [self.indices_]
score = self._calc_score(x_train, y_train, x_test, y_test, self.indices_)
self.scores_ = [score]
while dim > self.k_features:
scores = []
subsets = []
for p in combinations(self.indices_, r=dim - 1):
score = self._calc_score(x_train, y_train, x_test, y_test, p)
scores.append(score)
subsets.append(p)
best = np.argmax(scores)
self.indices_ = subsets[best]
self.subsets_.append(self.indices_)
dim -= 1
self.scores_.append(scores[best])
self.k_socre_ = self.scores_[-1]
return self
def transform(self, x):
return x[:, self.indices_]
def _calc_score(self, x_train, y_train, x_test, y_test, indices):
self.estimator.fit(x_train[:, indices], y_train)
y_pred = self.estimator.predict(x_test[:, indices])
score = self.scoring(y_test, y_pred)
return score📍 KNN 분류기를 사용하여 SBS 구현이 잘 동작하는지 확인
import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors=5)
sbs = SBS(knn, k_features=1)
sbs.fit(x_train_std, y_train)📍 KNN 분류기의 정확도 시각화
k_feat = [len(k) for k in sbs.subsets_]
plt.plot(k_feat, sbs.scores_, marker='o')
plt.ylim([0.7, 1.02])
plt.ylabel('Accuracy')
plt.xlabel('Number of features')
plt.grid()
plt.tight_layout()
plt.show()
~~>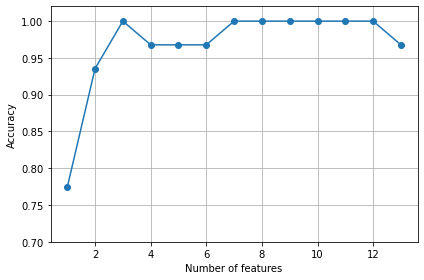
📍 가장 작은 개수의 조합( k = 3 )에서 높은 검증 데이터셋 성능을 내는 특성 확인
k3 = list(sbs.subsets_[10])
print(df_wine.columns[1:][k3])
~~>
Index(['Alcohol', 'Malic acid', 'OD280/OD315 OF diluted wines'], dtype='object')knn.fit(x_train_std, y_train)
print('훈련 정확도: ', knn.score(x_train_std, y_train))
print('테스트 정확도: ', knn.score(x_test_std, y_test))
~~>
훈련 정확도: 0.967741935483871
테스트 정확도: 0.9629629629629629knn.fit(x_train_std[:, k3], y_train)
print('훈련 정확도: ', knn.score(x_train_std[:, k3], y_train))
print('테스트 정확도: ', knn.score(x_test_std[:, k3], y_test))
~~>
훈련 정확도: 0.9516129032258065
테스트 정확도: 0.9259259259259259랜덤 포레스트의 특성 중요도 사용
✨ 랜덤 포레스트를 사용하면 앙상블에 참여한 모든 결정 트리에서 계산한 평균적인 불순도 감소로 특성 주용도를 측정할 수 있다
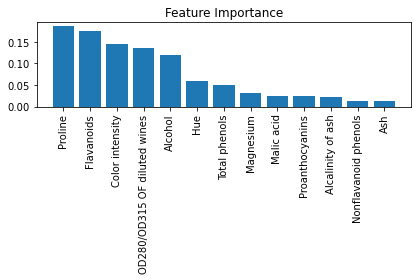
📍 랜덤 포레스트 모델의 특성 중요도 시각화
from sklearn.ensemble import RandomForestClassifier
feat_labels = df_wine.columns[1:]
forest = RandomForestClassifier(n_estimators=500, random_state=1)
forest.fit(x_train, y_train)
importances = forest.feature_importances_
indices = np.argsort(importances)[::-1]
for f in range(x_train.shape[1]):
print('%2d) %-*s %f' %(f+1, 30, feat_labels[indices[f]], importances[indices[f]]))
plt.title('Feature Importance')
plt.bar(range(x_train.shape[1]),
importances[indices],
align='center')
plt.xticks(range(x_train.shape[1]),
feat_labels[indices], rotation=90)
plt.xlim([-1, x_train.shape[1]])
plt.tight_layout()
plt.show()
~~>
1) Proline 0.185453
2) Flavanoids 0.174751
3) Color intensity 0.143920
4) OD280/OD315 OF diluted wines 0.136162
5) Alcohol 0.118529
6) Hue 0.058739
7) Total phenols 0.050872
8) Magnesium 0.031357
9) Malic acid 0.025648
10) Proanthocyanins 0.025570
11) Alcalinity of ash 0.022366
12) Nonflavanoid phenols 0.013354
13) Ash 0.013279