5장. 차원 축소를 사용한 데이터 압축 -1

💡 이 장에서 다룰 주제
- 주성분 분석( Principal Component Analysis, PCA )을 사용한 비지도 데이터 압축하기
- 지도 방식의 차원 축소 기법인 선형 판별 분석( Linear Disriminant Analysis, LDA )을 이용하여 클래스 구별 능력 최대화하기
- 커널 PCA( Kernel Principal Component Analysis, KPCA )를 사용한 비선형 차원 축소하기
주성분 분석을 통한 비지도 차원 축소
- 특성 선택과 마찬가지로 여러 가지 특성 추출 기법을 사용하여 데이터셋의 특성 개수를 줄일 수 있다
- 특성 선택과 특성 추출의 차이는 원본 특성을 유지하느냐의 차이
- 순차 후진 선택 같은 특성 선택 알고리즘을 사용할 때는 원본 특성을 유지하지만 특성 추출은 새로운 특성 공간으로 데이터를 변환하거나 투영
- 특성 추출은 대부분의 관련 있는 정보를 유지하면서 데이터를 압축하는 방법
- 저장 공간을 절약
- 학습 알고리즘의 계산 효율성을 향상
- 차원의 저주 문제를 감소시켜 예측 성능을 향상
PCA
- 주로 특성 추출과 차원 축소 용도로 많은 분야에서 널리 사용
- PCA를 많이 사용하는 애플리케이션에는 탐색적 데이터 분석과 주식 거래 시장의 잡음 제거, 생물정보학 문야에서 게놈 데이터나 유전자 발현 분석 등이 있다
- 특성 사이의 상관관계를 기반으로 하여 데이터에 있는 특성을 잡아낼 수 있다
- PCA는 고차원 데이터에서 분산이 가장 큰 방향을 찾고 좀 더 작거나 같은 수의 차원을 갖는 새로운 부분 공간으로 이를 투영
- 새로운 부분 공간의 직교 좌표는 주어진 조건하에서 분산이 최대인 방향으로 해석할 수 있다
- 새로운 특성 축은 서로 직각을 이룬다
- 특성의 스케일이 다르고 모든 특성의 중요도를 동일하게 취급하려면 PCA를 적용하기 전에 특성을 표준화 전처리해야 한다
💡 차원 축소를 위한 PCA 알고리즘을 자세히 알아보기 전에 사용할 방법
1. 차원 데이터셋을 표준화 전처리 한다
2. 공분산 행렬( covariance matrix )을 만든다
3. 공분산 행렬을 고유 벡터와 고윳값으로 분해
4. 고윳값을 내림차순으로 정렬하고 그에 해당하는 고유 벡터의 순위를 매긴다
5. 고윳값이 가장 큰 개의 고유 벡터를 선택 ( 는 새로운 특성 부분 공간의 차원 )
6. 최상위 개의 고유 벡터로 투영 행렬 를 만든다
7. 투영 행렬 를 사용해서 차원 입력 데이터셋 를 새로운 차원의 특성 부분 공간으로 변환
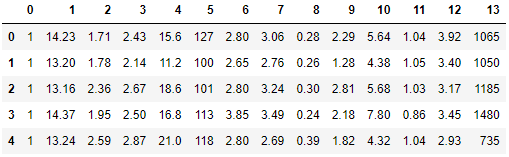
📍 Wine 데이터셋 불러오기
import pandas as pd
df_wine = pd.read_csv('http://archive.ics.uci.edu/ml/'
'machine-learning-databases/wine/wine.data',
header=None)
df_wine.head()
~~>
📍 train, test 데이터셋 나누기
from sklearn.model_selection import train_test_split
x, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.3,
stratify=y, random_state=0)📍 특성 표준화
from sklearn.preprocessing import StandardScaler
sc = StandardScaler()
x_train_std = sc.fit_trainsform(x_train)
x_test_std = sc.transform(x_test)📍 공분산 행렬에 대한 고유 벡터와 고윳값 쌍 계산
import numpy as np
cov_mat = np.cov(x_train_std.T)
eigen_vals, eigen_vecs = np.linalg.eig(cov_mat)
print('고윳값 \n%s' % eigen_vals)
~~>
고윳값
[4.84274532 2.41602459 1.54845825 0.96120438 0.84166161 0.6620634
0.51828472 0.34650377 0.3131368 0.10754642 0.21357215 0.15362835
0.1808613 ]📍 고유 벡터와 고윳값의 쌍을 정렬
# ( 고윳값, 고유 벡터 ) 튜플의 리스트 만들기
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i])
for i in range(len(eigen_vals))]
# 높은 값에서 낮은 값으로 ( 고윳값, 고유 벡터 ) 튜플 정렬
eigen_pairs.sort(key=lambda k: k[0], reverse=True)w = np.hstack((eigen_pairs[0][1][:, np.newaxis],
eigen_pairs[1][1][:, np.newaxis]))
print('투영 행렬 W: \n', w)
~~>
투영 행렬 W:
[[-0.13724218 -0.50303478]
[ 0.24724326 -0.16487119]
[-0.02545159 -0.24456476]
[ 0.20694508 0.11352904]
[-0.15436582 -0.28974518]
[-0.39376952 -0.05080104]
[-0.41735106 0.02287338]
[ 0.30572896 -0.09048885]
[-0.30668347 -0.00835233]
[ 0.07554066 -0.54977581]
[-0.32613263 0.20716433]
[-0.36861022 0.24902536]
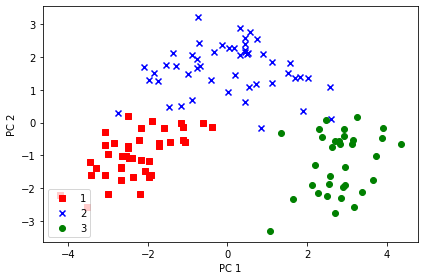
[-0.29669651 -0.38022942]]📍 훈련 데이터셋 시각화
x_train_std[0].dot(w)
~~>
array([ 2.38299011, -0.45458499])import matplotlib.pyplot as plt
colors = ['r', 'b', 'g']
markers = ['s', 'x', 'o']
x_train_pca = x_train_std.dot(w)
for l, c, m in zip(np.unique(y_train), colors, markers):
plt.scatter(x_train_pca[y_train==l, 0],
x_train_pca[y_train==l, 1],
c=c, label=l, marker=m)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
~~>
사이킷런의 주성분 분석
- 사이킷런의 PCA클래스를 Wine 데이터셋의 훈련 데이터셋에 적용
- 로지스틱 회귀로 변환된 샘플 데이터를 분류
from matplotlib.colors import ListedColormap
def plot_decision_regions(x, y, classifier, resolution=0.02):
# 마커와 컬러맵 준비
markers = ('s', 'x', 'o', '^', 'v')
colors = ('r', 'b', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
# 결정 경계 그리기
x1_min, x1_max = x[:, 0].min() - 1, x[:, 0].max() + 1
x2_min, x2_max = x[:, 1].min() - 1, x[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T)
z = z.reshape(xx1.shape)
plt.contourf(xx1, xx2, z, alpha=0.4, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
# 클래스 샘플을 표시
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=x[y==cl, 0], y=x[y==cl, 1],
alpha=0.6, c=cmap.colors[idx],
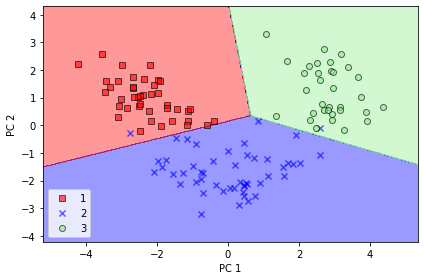
edgecolor='black', marker=markers[idx], label=cl)📍 훈련 데이터에서 만든 결정 경계
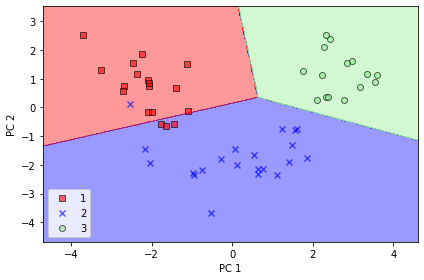
from sklearn.linear_model import LogisticRegression
from sklearn.decomposition import PCA
# PCA 변환기와 로지스틱 회귀 추정기를 초기화
pca = PCA(n_components=2)
lr = LogisticRegression(random_state=1)
# 차원 축소
x_train_pca = pca.fit_transform(x_train_std)
x_test_pca = pca.transform(x_test_std)
# 축소된 데이터셋으로 로지스틱 회귀 모델 훈력
lr.fit(x_train_pca, y_train)
plot_decision_regions(x_train_pca, y_train, classifier=lr)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
~~>
📍 테스트 데이터 결정 경계
plot_decision_regions(x_test_pca, y_test, classifier=lr)
plt.xlabel('PC 1')
plt.ylabel('PC 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
~~>
💡 전체 주성분의 설명된 분산 비율
- n_components 매개변수를 None으로 지정하고 PCA 클래스의 객체를 만들면 된다
- explainedvariance_ratio 속성에서 모든 주성분의 설명된 분산 비율을 확인 가능
pca = PCA(n_components=None)
x_train_pca = pca.fit_transform(x_train_std)
pca.explained_variance_ratio_
~~>
array([0.36951469, 0.18434927, 0.11815159, 0.07334252, 0.06422108,
0.05051724, 0.03954654, 0.02643918, 0.02389319, 0.01629614,
0.01380021, 0.01172226, 0.00820609])from sklearn.decomposition import IncrementalPCA
ipca = IncrementalPCA(n_components=9)
for batch in range(len(x_train_std)//25+1):
x_batch = x_train_std[batch*25:(batch+1)*25]
ipca.partial_fit(x_batch)
print('주성분 개수: ', ipca.n_components_)
print('설명된 분산 비율: ', np.sum(ipca.explained_variance_ratio_))
~~>
주성분 개수: 9
설명된 분산 비율: 0.9478392700446665선형 판별 분석을 통한 지도 방식의 데이터 압축
선형 판별 분석( Linear Discriminant Analysis, LDA )
- 규제가 없는 모델에서 차원의 저주로 인해 과대적합 정도를 줄이고 계산 효율성을 높이기 위한 특성 추출의 기법을 사용할 수 있다
- PCA와 비슷하지만 PCA는 데이터셋에서 있는 분산이 최대인 직교 성분 축을 찾으려 하지만,
LDA는 클래스를 최적으로 구분할 수 있는 특성 부분 공간을 찾는 것
주성분 분석( PCA ) VS 선형 판별 분석( LDA )
- PCA와 LDA 모두 데이터셋의 차원 개수를 줄일 수 있는 선형 변환 기법
- PCA는 비지도 학습 알고리즘
- LDA는 지도 학습 알고리즘
- 따라서 LDA가 PCA보다 분류 작업에서 더 뛰어난 특성 추출 기법이라고 생각할 수 있다
💡 각 클래스에 속한 샘플이 몇 개 되지 않을 때는 PCA를 통한 전처리가 특정 이미지 인식 작업에 더 뛰어난 분류 결과를 내는 경향이 있기도 하다
📍 이진 분류 문제를 위한 LDA 개념 요약
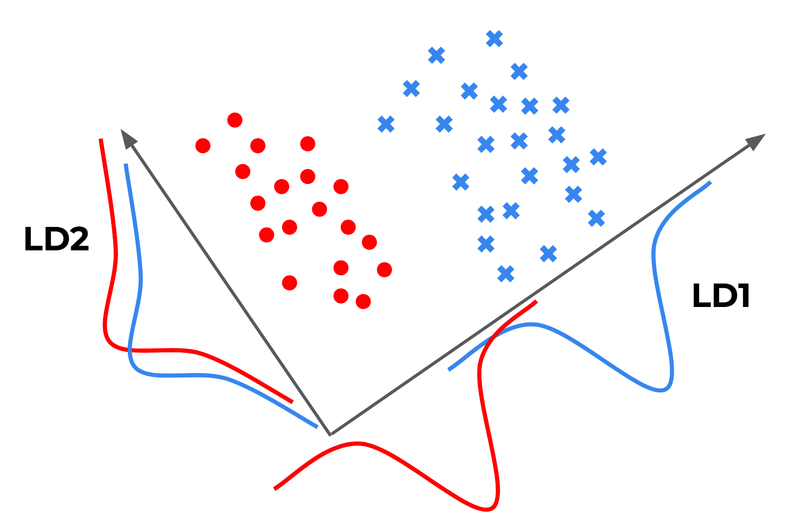
- X축으로 투영하는 선형 판별 벡터는 두 개의 정규 분포 클래스를 잘 구분한다
- Y축으로 투영하는 선형 판별 벡터는 데이터셋에 있는 분산을 많이 잡아 내지만 클래스 판별 정보가 없기 때문에 좋은 선형 판별 벡터가 되지 못 한다
선형 판별 분석의 내부 동작 방식
- 차원의 데이터셋을 표준화 전처리한다( 는 특성 개수 )
- 각 클래스에 대해 차원의 평균 벡터를 계산
- 클래스 간의 산포 행렬 와 클래스 내 산포 행렬 를 구성
- 행렬의 고유 벡터와 고윳값을 계산
- 고윳값을 내림차순으로 정렬하여 고유 벡터의 순서를 매긴다
- 고윳값이 가장 큰 개의 고유 벡터를 선택하여 차원의 변환 행렬 를 구성한다( 이 행렬의 열이 고유 벡터 )
- 변환 행렬 를 사용하여 샘플을 새로운 특성 부분 공간으로 투영
산포 행렬 계산
💡 PCA 절에서 Wine 데이터셋의 특성을 이미 표준화했기 때문에 단계 1 은 건너뛰고 바로 평균 벡터 계산을 진행
-
평균 벡터를 사용하여 클래스 간의 산포 행렬과 클래스 내 산포 행렬을 구성
- 평균 벡터 는 클래스 의 샘플에 대한 특성의 평균값 을 저장
- 평균 벡터 는 클래스 의 샘플에 대한 특성의 평균값 을 저장
-
세 개의 평균 벡터가 만들어진다
📍 평균 벡터를 사용하여 클래스 내 산포 행렬 계산
np.set_printoptions(precision=4)
mean_vecs = []
for label in range(1, 4):
mean_vecs.append(np.mean(x_train_std[y_train==label], axis=0))
print('MV %s: %s\n' %(label, mean_vecs[label-1]))
~~>
MV 1: [ 0.9066 -0.3497 0.3201 -0.7189 0.5056 0.8807 0.9589 -0.5516 0.5416
0.2338 0.5897 0.6563 1.2075]
MV 2: [-0.8749 -0.2848 -0.3735 0.3157 -0.3848 -0.0433 0.0635 -0.0946 0.0703
-0.8286 0.3144 0.3608 -0.7253]
MV 3: [ 0.1992 0.866 0.1682 0.4148 -0.0451 -1.0286 -1.2876 0.8287 -0.7795
0.9649 -1.209 -1.3622 -0.4013]📍 개별 클래스 의 산포 행렬 를 더해 행렬 구하기
d = 13 # 특성 개수
S_W = np.zeros((d, d))
for label, mv in zip(range(1, 4), mean_vecs):
class_scatter = np.zeros((d, d))
for row in x_train_std[y_train==label]:
row, mv = row.reshape(d, 1), mv.reshape(d, 1)
class_scatter += (row - mv).dot((row-mv).T)
S_W += class_scatter
print('클래스 내의 산포 행렬: %s x %s' %(S_W.shape[0], S_W.shape[1]))
~~>
클래스 내의 산포 행렬: 13 x 13💡 산포 행렬을 계산할 때 훈련 데이터셋의 클래스 레이블이 균등하게 분포되어 있다고 가정한다
print('클래스 레이블 분포: %s' %(np.bincount(y_train)[1:]))
~~>
클래스 레이블 분포: [41 50 33]👉 하지만 클래스 레이블의 개수를 출력해 보면 가정이 틀린 것을 알 수 있다
✨✨✨ 개별 산포 행렬 를 산포 행렬 로 모두 더하기 전에 스케일을 조정해야 한다
- 산포 행렬을 클래스 샘플 개수 로 나누면 산포 행렬을 계산하는 것이 공분산 행렬를 계산하는 것과 같다
- 즉 공분산 행렬은 산포 행렬의 정규화 버전이다
d = 13 # 특성 개수
S_W = np.zeros((d, d))
for label, mv in zip(range(1, 4), mean_vecs):
class_scatter = np.cov(x_train_std[y_train==label].T)
S_W += class_scatter
print('스케일 조정된 클래스 내의 산포 행렬: %s x %s' %(S_W.shape[0], S_W.shape[1]))
~~>
스케일 조정된 클래스 내의 산포 행렬: 13 x 13📍 클래스 간의 산포 행렬 계산
mean_overall = np.mean(x_train_std, axis=0)
mean_overall = mean_overall.reshape(d, 1) # 열 벡터로 만들기
d = 13 # 특성 개수
S_B = np.zeros((d, d))
for i, mean_vec in enumerate(mean_vecs):
n = x_train[y_train == i + 1, :].shape[0]
mean_vec = mean_vec.reshape(d, 1) # 열 벡터로 만들기
S_B += n * (mean_vec - mean_overall).dot((mean_vec - mean_overall).T)
print('클래스 간의 산포 행렬: %s x %s' %(S_B.shape[0], S_B.shape[1]))
~~>
클래스 간의 산포 행렬: 13 x 13새로운 특성 부분 공간을 위해 선형 판별 벡터 선택
📍 행렬 의 고윳값 계산
eigen_vals, eigen_vecs = np.linalg.eig(np.linalg.inv(S_W).dot(S_B))📍 고유 벡터와 고윳값 쌍을 계산 후 내림차순으로 고윳값 정렬
eigen_pairs = [(np.abs(eigen_vals[i]), eigen_vecs[:, i])
for i in range(len(eigen_vals))]
eigen_pairs = sorted(eigen_pairs, key=lambda k: k[0], reverse=True)
print('내림차순의 고윳값 \n')
for eigen_val in eigen_pairs:
print(eigen_val[0])
~~>
내림차순의 고윳값
349.61780890599397
172.76152218979388
2.500628935846022e-14
2.500628935846022e-14
1.7655500524988485e-14
1.7655500524988485e-14
1.5928250906297086e-14
1.2137014082220281e-14
1.2137014082220281e-14
8.535519974411294e-15
4.951574641747866e-15
4.951574641747866e-15
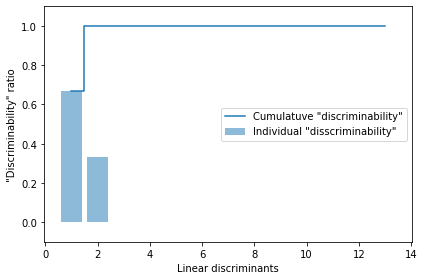
0.0📍 선형 판별 벡터 시각화
tot = sum(eigen_vals.real)
discr = [(i / tot) for i in sorted(eigen_vals.real, reverse=True)]
cum_discr = np.cumsum(discr)
plt.bar(range(1, 14), discr, alpha=0.5,
align='center', label='Individual "disscriminability"')
plt.step(range(1, 14), cum_discr, where='mid',
label='Cumulatuve "discriminability"')
plt.ylabel('"Discriminability" ratio')
plt.xlabel('Linear discriminants')
plt.ylim([-0.1, 1.1])
plt.legend(loc='best')
plt.tight_layout()
plt.show()
~~>
w = np.hstack((eigen_pairs[0][1][:, np.newaxis].real,
eigen_pairs[1][1][:, np.newaxis].real))
print('행렬 W\n', w)
~~>
행렬 W
[[-0.1481 -0.4092]
[ 0.0908 -0.1577]
[-0.0168 -0.3537]
[ 0.1484 0.3223]
[-0.0163 -0.0817]
[ 0.1913 0.0842]
[-0.7338 0.2823]
[-0.075 -0.0102]
[ 0.0018 0.0907]
[ 0.294 -0.2152]
[-0.0328 0.2747]
[-0.3547 -0.0124]
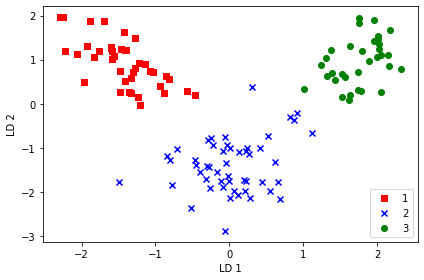
[-0.3915 -0.5958]]📍 와인 클래스 특성 구분
- 변환 행렬 를 훈련 데이터셋에 곱해 데이터를 변환할 수 있다
x_train_ida = x_train_std.dot(w)
colors = ['r', 'b', 'g']
markers = ['s', 'x', 'o']
for l, c, m in zip(np.unique(y_train), colors, markers):
plt.scatter(x_train_ida[y_train==l, 0],
x_train_ida[y_train==l, 1] * (-1),
c=c, label=l, marker=m)
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower right')
plt.tight_layout()
plt.show()
~~>
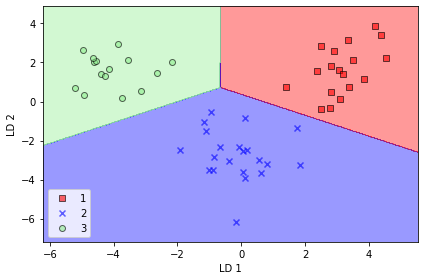
📍 사이킷런의 LDA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
lda = LDA(n_components=2)
x_train_lda = lda.fit_transform(x_train_std, y_train)lr = LogisticRegression(random_state=1)
lr = lr.fit(x_train_lda, y_train)
plot_decision_regions(x_train_lda, y_train, classifier=lr)
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
~~>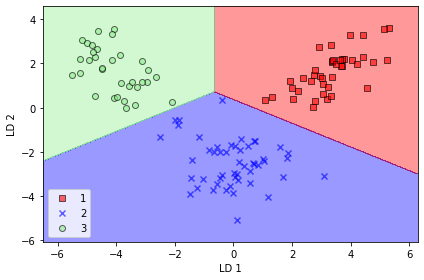
- 로지스틱 회귀 모델이 클래스 2의 샘플 하나를 제대로 분류하지 못 했다
- 규제 강도를 낮추어 로지스틱 회귀 모델이 훈련 데이터셋의 모든 샘플을 정확하게 분류하도록 결정 경계를 옮길 수 있다
📍 test 결과
x_test_lda = lda.transform(x_test_std)
plot_decision_regions(x_test_lda, y_test, classifier=lr)
plt.xlabel('LD 1')
plt.ylabel('LD 2')
plt.legend(loc='lower left')
plt.tight_layout()
plt.show()
~~>