
본 포스팅은 제가 읽었던 논문을 간단하게 정리하는 글입니다. 논문의 모든 내용을 작성하는 것이 아닌, 일부분만 담겨 있으므로 자세한 내용은 원본 논문을 확인해 주시기를 바랍니다. 또한, 논문을 잘못 이해한 부분이 있을 수 있으므로, 양해 바랍니다.
Overview
Camelyon16 competition 우승자의 논문으로, 저자들은 WSI(whole slide image) classification과 tumor localization 모두 우승하였음.
Our team won both competitions in the grand challenge, obtaining an area under the receiver operating curve (AUC) of 0.925 for the task of whole slide image classification and a score of 0.7051 for the tumor localization task.
본 포스팅에서는 논문의 내용 중 모델링 일부만을 다루므로, 자세한 내용은 원본 논문을 참고.
Method
Image Pre-processing
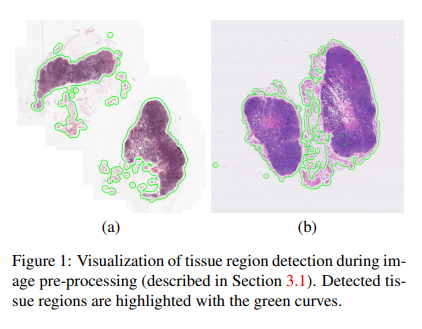
모델에 WSI를 입력하기 전, 전체 WSI 중 tissue(조직)의 영역을 segmentation 하여 조직이 포함된 영역만을 모델에 입력하였음. 위의 이미지에서는 초록색으로 조직의 영역을 표시하였음.
RGB 이미지를 HSV 색 공간으로 변환한 후, Otsu thresholding 알고리즘을 각 채널에 적용함. 최종 mask는 H 채널과 S 채널의 마스크를 조합하여 생성하였음. 평균적으로 WSI의 82%가 background임을 확인하였음.
Detection Framework

Camelyon16 데이터셋의 경우 tumor 위치가 ROI polygon으로 제공되므로, WSI 내 어떤 영역이 양성인지 확인할 수 있음. 양성인 영역과 음성인 영역에서 패치(patch)를 각각 추출한 뒤, patch-level의 분류기(CNN)를 훈련하였음.
- semi-supervised, weakly-supervised 등 추가적인 학습 방법론을 사용하지 않고 단순한 fully-supervised learning 적용
- 분류기의 경우 GoogLeNet, AlexNet, VGG16, FaceNet을 사용하여 실험하였으며 최종적으로 GoogLeNet 사용
데이터셋이 다양한 배율(40x, 20x, 10x)의 이미지를 제공하나, 40배율에서 가장 좋은 성능을 확인하여 40배율만 사용한 예측 결과를 제출하였음.
Hard Negative Mining
전체 데이터셋을 훈련하여 성능을 확인해 보니, 전체 오류 중 상당한 오류가 암의 조직학적 모방(histologic mimics of cancer)에 의한 false positive error임을 확인하였음.
이러한 영역에서의 모델 성능을 향상시키고자, hard negative 영역에서의 patch(hard negative pathces)를 추가로 추출하여 모델을 재훈련하였음.
Post-processing
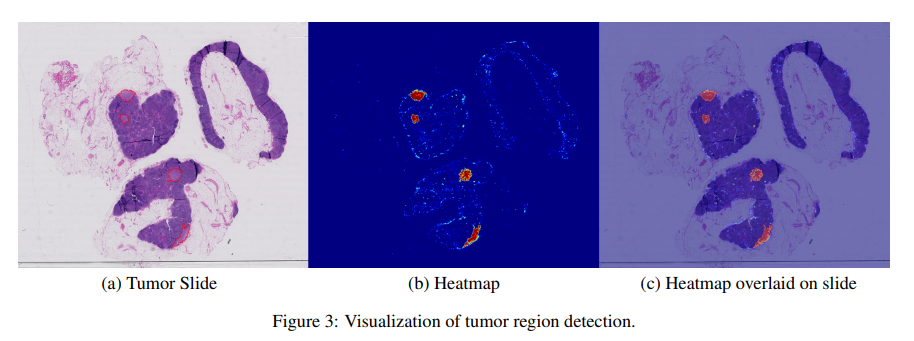
Patch-level의 classification을 모두 수행하면, 각 WSI 마다 tumor probability heatmap을 생성함. Heatmap의 각 픽셀은 0에서 1 사이의 값을 가짐.
각 heatmap을 기반으로 slide-based score와 lesion-based score를 계산함.
Slide-based Classification
Heatmap에 post-processing을 적용하여 최종 slide-level 예측 결과를 계산함.
Heatmap 기반의 28개의 feature를 추출하며, 각 feature는 전체 조직 영역 대비 tumor 영역의 비율, patch 평균 예측 값, tumor 영역의 longest axis 등을 포함함.
이러한 feature를 RandomForest에 입력하여 주어진 WSI가 양성인지 음성인지 분류함.
Lesion-based Detection
Lesion detection의 경우 false positive가 적은 모델을 목표로 하였으며, 이를 위해 2개의 딥러닝 모델을 훈련하였음.
첫 번째 모델(D-I)의 경우 위의 Detection Framework대로 모델을 훈련하며, 두 번째 모델(D-II)의 경우 tumor에 인접한 음성 샘플이 많은 훈련 세트로 훈련함.
두 모델을 모두 훈련한 후, 다음의 과정을 진행하였음.
- D-I 모델에서 생성된 heatmap을 기반으로, 픽셀 값이 0.9(임계값) 이상이면 1, 아니면 0인 binary heatmap을 생성
- Tumor binary mask에서 연결된 component를 식별한 후, 그 중심점을 tumor의 위치로 식별
- D-II 모델 또한 tumor 위치를 식별하며, 두 모델의 평균값으로 최종 위치를 예측
