Joint Semi-supervised and Active Learning for Segmentation of Gigapixel Pathology Images with Cost-Effective Labeling
논문 정리

본 포스팅은 제가 읽었던 논문을 간단하게 정리하는 글입니다. 논문의 모든 내용을 작성하는 것이 아닌, 일부분만 담겨 있으므로 자세한 내용은 원본 논문을 확인해 주시기를 바랍니다. 또한, 논문을 잘못 이해한 부분이 있을 수 있으므로, 양해 바랍니다.
Overview
WSI에서 딥러닝의 적용은 많은 성과를 이루었지만, supervised learning을 위한 labeling 작업은 여전히 많은 시간과 비용이 필요함. WSI의 경우 하나의 슬라이드에서 수만 개의 patch가 추출되며, 각 patch마다 label을 달아주는 작업은 매우 고난도의 작업임.
최근, 모델 학습을 위한 large labeled dataset을 직접 구하기 보다는 semi-supervised learning (SSL) 등의 방법이 많은 주목을 받고 있음. SSL은 컴퓨터 비전 분야에서 많은 성과를 이루었지만, 병리 AI에서는 아직 추가적인 연구가 필요함.
Labeling 작업의 어려움을 줄이기 위한 다른 방법으로는 active learning이 있음. Active learning은 최소한의 labeling 작업만으로 최대한의 모델 성능을 얻고자 하는 학습 방법임. 모델이 잘 맞추는 쉬운 샘플보다는 모델이 잘 맞추지 못하는 어려운 샘플의 레이블을 제공한다면 모델은 보다 빠르고 정확하게 학습할 수 있음.
Active learning 적용 시 주의해야 하는 것은 cold-start problem임. 충분하지 않은 labeled data에서 모델을 학습한다면 모델은 high-biased가 되어 query를 위한 selection의 결과가 좋지 않아짐(poor selection). Selection의 결과가 좋지 않으므로 query에 대한 응답 또한 좋지 않을 것이며, 결과적으로 좋은 labeled dataset을 얻을 수 없으므로 악순환이 반복됨.
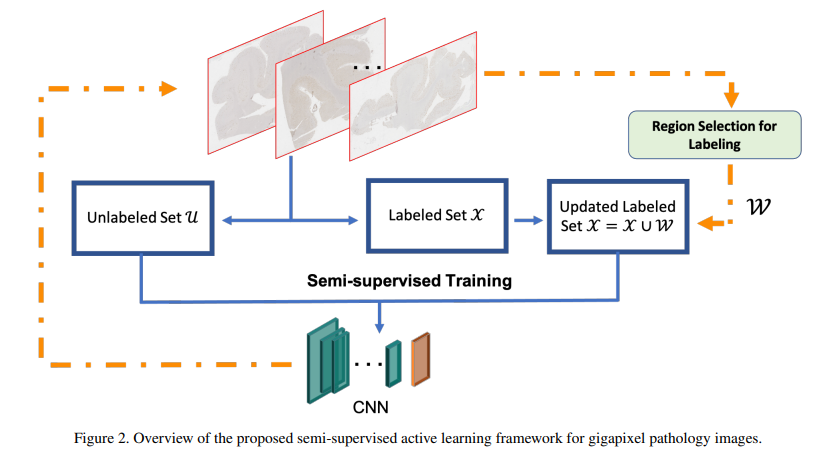
이러한 문제를 완화하기 위해, 본 논문에서는 SSL과 active learning을 결합(joint)한 새로운 framework를 제안함. Lack of labeled data 상황에서 SSL 기반의 학습은 단순한 supervised learning 보다 cold-start problem을 완화시킬 것이며, 결과적으로 active learning의 query를 통해 보다 가치 있는 labeling 작업을 수행할 수 있을 것임.
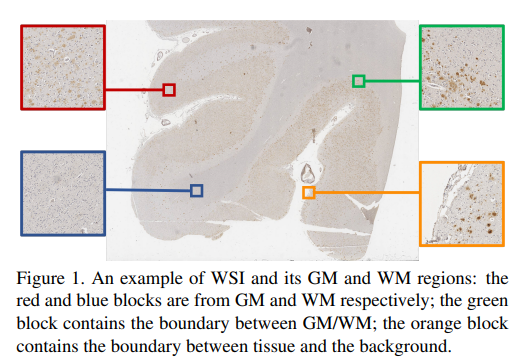
해당 framework를 알츠하이머 감지를 위한 segmentation에 적용하여 실험하였음. WSI를 background / GM / WM으로 segmentation. 아래 이미지 참고.
Semi-supervised Active learing
Semi-supervised learning
훈련 데이터셋 는 labeled set 을 가지며, unlabeled 데이터셋 는 를 가짐. 는 번째 patch를 의미하며, 의 경우 해당 patch가 background인지, GM인지, WM인지 나타냄( ).
주어진 데이터를 기반으로, classifier 을 학습하며, 아래의 loss function을 가짐. 아래에서 이며 임.
은 per-sample supervised loss, 는 regularization을 의미함.
- 맥락상 는 FixMatch의 consistency regularization을 뜻하는 것 같습니다.
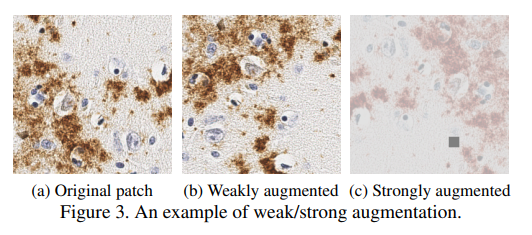
본 논문에서는 semi-supervised learning을 위해 FixMatch에서 제안된 pseudo labeling과 consistency regularization을 사용함. Unlabeled data에 weak augmentation을 적용한 후 최대 prediction 값을 pseudo label로 사용하고, 생성된 pseudo label과 strong augmented prediction 값을 사용하여 cross-entropy loss를 계산함.
적용된 변환 예시는 아래와 같음.
Active Learning
기존의 active learning에서는 샘플 단위로 query를 진행하였음. WSI에서 샘플 단위란 patch 단위를 의미함. 하나의 patch는 충분한 정보를 포함하지 않으며, patch 기반 하나의 patch에만 초점을 맞춘 기준은 labeling query에 대한 무작위적인 선택으로 이어질 수 있음.
따라서, patch-based selection이 아닌 region-based selection을 제안함. 하나의 region 은 로 개의 patch로 분할될 수 있음. 하나의 region을 query 하면, 다음과 같이 labeled data가 추가됨.
는 region 에서 추출된 모든 patch를 의미하며, 는 에 할당된 label을 의미함. 적절한 region을 선택하는 방법은 아래에서 다룸.
CNN 모델(encoder)의 좋은 initializing을 위해, self-supervised learning을 사용한 pre-training process를 적용하였음. 학습시키고자 하는 classifier 라 할 때, self-supervised learning 방법 중 하나인 SimCLR을 사용하여 encoder 를 훈련하였음.
인코더 후 classification layer 를 적용한다면 같은데 우선 논문 그대로 작성하였습니다.
Last layer for the classifier 의 initializing을 위해, GM과 WM 사이의 영역과 tissue와 background 사이의 영역 이미지를 넣어 초기 학습을 진행하였음. 두 영역을 통해 모든 class(GM, WM, background)의 정보를 제공할 수 있음.
Region-based selection
이 섹션에서는 labeling query를 위한 region 선택 방법을 설명함. 기존의 active learning 컨셉과 동일하게, high uncertainty를 가지는 region을 선택함.

Figure 4.에서 볼 수 있듯이, 먼저 WSI를 patch-level로 분할한 뒤 patch-level의 예측 값을 얻음. 이후, patch 별 예측 값의 최댓값을 모은 행렬 을 계산함. 즉, 는 아래의 를 원소로 가짐.
의 index()는 하나의 patch에 대응함. 여러 개의 patch를 region으로 통합시키기 위하여, 커널 를 에 convolution 함.
는 모든 원소의 값이 이고 크기가 인 mean filter로 생각할 수 있음. 이제 의 index는 하나의 region에 대응함. 하나의 region은 개의 patch로 이루어짐. 의 각 원소별 값은 해당 위치 region의 confidence 값(uncertainty의 반대)으로 생각할 수 있음.
논문에서는 각 patch의 크기를 으로, 의 크기를 로 설정하여 각 region의 크기가 이 되게 하였음. 이후 개의 region을 선택하여 oracle에게 query, oracle은 query 된 region에 labeling 작업을 수행함. 중복되는 결과를 위해 이전에 선택되었던 region은 매우 큰 confidence 값을 가지게 하여 다시 선택되지 않도록 함.
아래의 알고리즘은 region-based semi-supervised active learning을 설명함.

- Pre-training 단계 : 앞서 언급하였던 SimCLR(self-supervised learning)을 사용하여 인코더 에 pre-training 진행
- Initialize 단계 : 2개의 region(WM/GM을 포함하는 region, tissue/background를 포함하는 region)을 선택한 후 pixel-wise labeling을 진행(ROI polygon 등). 이후 region을 patch 단위로 분할한 뒤 초기 labeled dataset 를 구성. 전체 데이터셋을 라고 하면, 초기 unlabeled dataset 은 으로 정의
- Fine-tuning 단계 : Loss function → 를 기반으로 파라미터 업데이트. : total number of steps(각 WSI에 대해서로 이해함), : total number of cycles 에 대해,
- unlabeled patch에 대해 prediction을 계산한 후 를 계산. labeled dataset에 속한 region의 경우 의 값을 무한대로 설정 → query 대상으로 선택되지 않도록
- 에서 값이 가장 작은(low confidence, high uncertainty) 개의 region 선택 → 각 region을 patch를 추출, label을 추가한 후 에 추가, 해당 patch를 unlabeled dataset 에서 제거
- 에 대한 반복문이 종료되면, FixMatch에 기반한 semi-supervised learning 수행. Label이 있는 경우 weak augmentation을 적용한 후의 cross-entropy loss, Unlabeled의 경우 Weak + Strong augmentation 기반의 cross-entropy loss를 사용하여 파라미터 업데이트
본 논문에서는 encoder 로 ResNet-18을 사용하였음. 추가적인 실험 세팅 및 실험 결과는 원본 논문을 참고.
