이번에 Kaggle의 Notebook중에 Covid_19 데이터로 XGBOOST를 사용해서 분석해보려고 한다.
https://www.kaggle.com/anshuls235/covid19-explained-through-visualizations의 Notebook을 보면서 코딩을 해보고 중간중간 모르는 부분은 구글링을 통해 학습하려고 한다. 코드 내용은 똑같이 해보려고 한다.
csv파일 불러오기
Covid_19 Data를 불러오기 위해 Pandas라는 패키지를 사용하려고 한다.
파이썬에서는 import를 사용하면 된다.
import pandas as pd그리고 나서 read_csv라는 명령어를 통해 csv파일들을 불러올 것이다.
df = pd.read_csv('경로/covid_19_data.csv')
df_train = pd.read_csv('경로/train.csv')
df_test = pd.read_csv('경로/test.csv')pandas를 이용하여 read_csv를 통해 파일들을 불러올 때 문자열 내에 경로를 복사해주면 된다. 단, 기본적으로 파일 위치 경로를 그냥 복사하면 /가 아닌 \로 복사가 될 것이다. 이를 /로 바꿔줘야 된다.
각각 csv 파일 확인
head()함수를 통해 분석하기 전에 각각 파일의 데이터들을 미리 알아보자
df
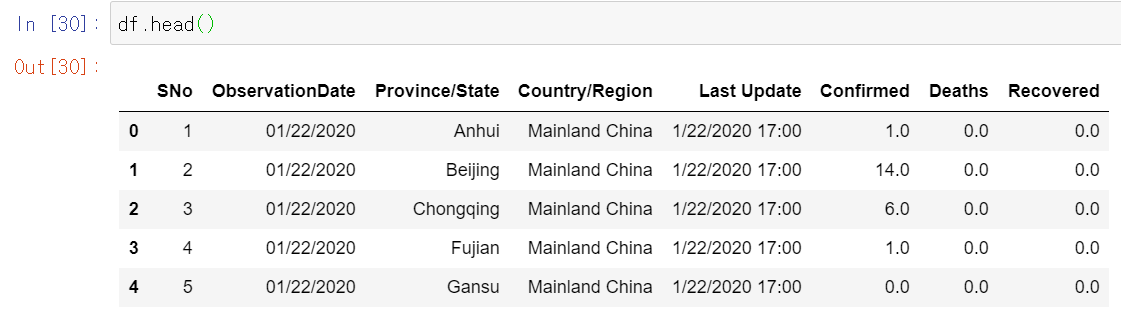
훑어보면 어림짐작해서 나라별 및 도시별 코로나 환자수, 사망자수, 회복자 수를 알 수 있다.
df_train
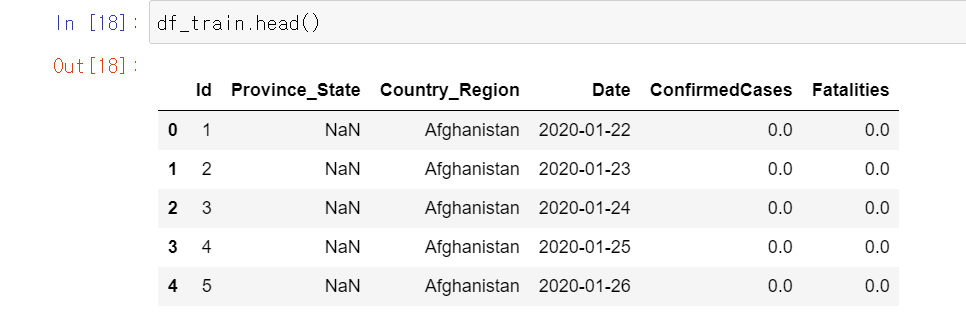
나라별 및 도시별 코로나 환자 수와 사망자를 예측하기 위한 train 케이스
df_test
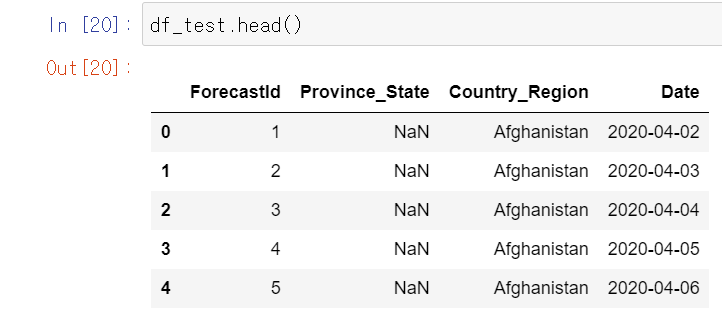
train한 모델을 테스트하기 위한 파일
데이터 전처리
code
df.rename(columns={'ObservationDate':'Date','Province/State':'Province_State',
'Country/Region':'Country_Region','Confirmed':'ConfirmedCases',
'Deaths':'Fatalities'},inplace=True)
df.loc[df['Country_Region']=='Mainland China','Country_Region']='China'
df['Date'] = pd.to_datetime(df['Date'],format='%m/%d/%Y')
df['Day'] = df.Date.dt.dayofyear
df['cases_lag_1'] = df.groupby(['Country_Region','Province_State'])['ConfirmedCases'].shift(1)
df['deaths_lag_1'] = df.groupby(['Country_Region','Province_State'])['Fatalities'].shift(1)
df['Daily Cases'] = df['ConfirmedCases'] - df['cases_lag_1']
df['Daily Deaths'] = df['Fatalities'] - df['deaths_lag_1'] df.rename
read_csv를 통해 Data Frame을 불러왔다. 이 불러온 df의 열 이름을 바꿔주려고 한다.
df.rename(columns = {'ObservatopmData' : 'Date', 'Province/State' : 'Province_State',
'Country/Region' : 'Country_Region', 'Confirmed' : 'ConfirmedCases',
'Deaths' : 'Fatalities'}, inplace = True)rename의 형식을 보면 다음과 같다.
df.rename(columns = {'a' : 'b'}, inplace = True)위 명령어를 실행하면 df의 컬럼중 a이라는 이름을 가진 열이 b라고 이름이 변경이 된다.
참고로 index를 변경해주려면 columns를 index로 바꿔주면 된다.
df.rename(index = {'a' : 'b'}, inplace = True)rename에는 여러 Parameter이 존재한다. 그 중에 inplace는 새로운 Data Frame를 만들어줄지에 대한 Parameter이다.
만약 inplace = True 라면 복사본은 무시된다.
이 외에도 여러가지가 존재하는데 이는
https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.rename.html
에서 확인해보면 된다.
참고로 열과 인덱스는 다음과 같다.
.jpg)
인덱스는 Row와 같다.
DataFrame 인덱싱
데이터프레임을 인덱싱 하는 명령어는 loc[]와 iloc[]가 있는데 여기선 loc[]가 쓰였으니 이에 대해서 공부해 보려고 한다.
df["row", "column"]의 방법으로 쓰면 된다. 첫 번째 매개변수에는 인덱싱할 row를 적고, 두 번째에는 column을 넣으면 된다. row나 column에는 이름을 넣어도 되고, 리스트를 넣어도 되고, 리스트 슬라이싱을 넣어도 된다.
df.loc[df['Country_Region']=='Mainland China','Country_Region']='China'
df['Country_Region'] == "Mainland China'인 Row와 Country_Region에 해당하는 Column을 인덱싱해서 China로 변환시켜줬다.
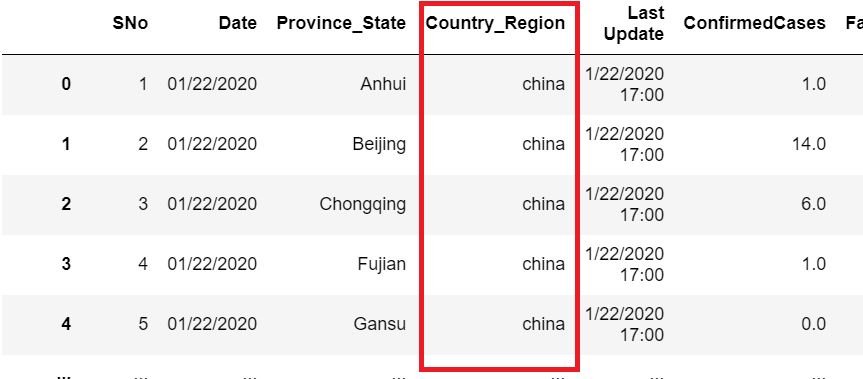
시간 처리법
df['Date'] = pd.to_datetime(df['Date'],format='%m/%d/%Y')
df['Day'] = df.Date.dt.dayofyear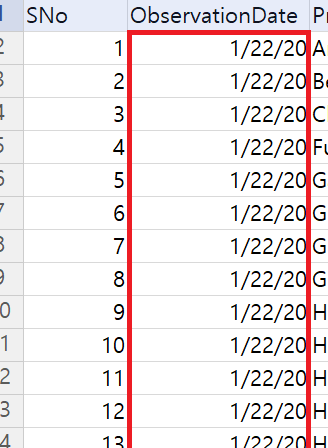
ObservationDate를 rename을 통해 Date로 이름을 바꿨었다.
to_datetime명령어를 통해 문자열(object)을 datetime64 타입으로 변경시켜줄 수 있다.
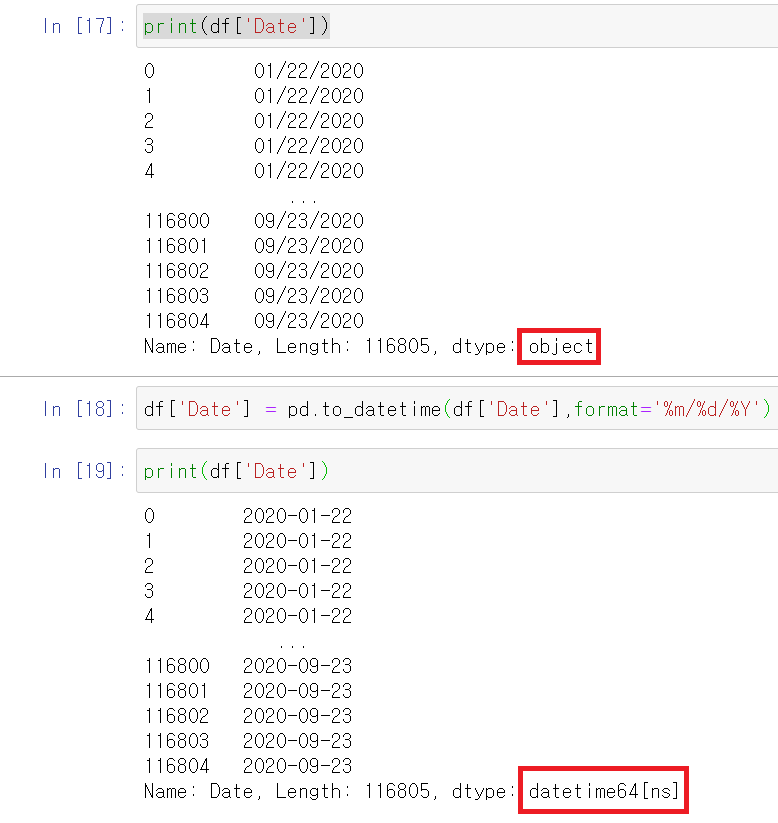
to_datetime 첫번째 파라미터로 list를 입력하면 datetimeIndex가 반환되고 Pandas의 Series 타입을 입력하면 datetime64형태의 Series 타입이 반환된다.
- %Y : 년도 ex) 2020
- %m : 월 ex) 01 ~ 12
- %d : 일 ex) 01 ~ 31
- %H : 시 ex) 01 ~ 23
- %M : 분 ex) 00 ~ 59
- %S : 초 ex) 00 ~ 59
%m/%d/%Y -> 12/07/2020
df['Day'] = df.Date.dt.dayofyeardf['Date']로 설정해도 된다. 연 기준 몇일째인지 알려준다. 예를 들어 2월 2일을 dayofyear로 연 시작일로부터 32번째 날짜임을 알 수 있다.
dt.quarter: 분기(숫자)dt.weekday_name: 요일 이름(문자)dt.quarter: 요일 이름 (숫자), 월-0 화-1 수-2 ... == dt.dayofweekdt.quarter: 연 기준 몇주째 == dt.weekdt.quarter: 월 일수(숫자) == dt.daysinmonth
groupby
df['cases_lag_1'] = df.groupby(['Country_Region','Province_State'])['ConfirmedCases'].shift(1)
df['deaths_lag_1'] = df.groupby(['Country_Region','Province_State'])['Fatalities'].shift(1)groupby는 같은 값을 하나로 묶어 통계내거나 집계하는 명령어이다.

이 코드의 정확한 메커즘은 모르겠지만 이렇게 해석했다.
.jpg)
df를 colA와 col B로 이뤄졌다고 했을 때 다음과 같은 groupby를 실행해서 col C 열을 만드려고 한다.
df['col C'] = df.groupby(['col A'])['col B'].shift(1)그러면 'A'에 관해서만 보자면, 'A'의 첫번째 col B값 '1'이 두번째 'A'로, 두번째 'A'의 값이 세 번째 'A'의 값으로 보낸다.
그러면 이 메커니즘을 코로나 데이터에도 똑같이 적용하면 Date별마다 국가별 및 도시별의 확진자 수와 사망자 수가 있을 것이다.
그러면 그 확진자 및 사망자 수를 Date별로 한칸 아래씩 shift한다.
그리고 난 다음에
df['Daily Cases'] = df['ConfirmedCases'] - df['cases_lag_1']
df['Daily Deaths'] = df['Fatalities'] - df['deaths_lag_1'] 
당일 확진자 및 사망자 수에 전일 확진자와 사망자 수를 빼서 증감 수를 알 수 있다.
시각화 준비 클래스 생성
추가 모듈
import pycountry
import pycountry_convert as pc
import plotly.express as pxcode
class country_utils():
def __init__(self):
self.d = {}
def get_dic(self):
return self.d
def get_country_details(self,country):
"""Returns country code(alpha_3) and continent"""
try:
country_obj = pycountry.countries.get(name=country)
if country_obj is None:
c = pycountry.countries.search_fuzzy(country)
country_obj = c[0]
continent_code = pc.country_alpha2_to_continent_code(country_obj.alpha_2)
continent = pc.convert_continent_code_to_continent_name(continent_code)
return country_obj.alpha_3, continent
except:
if 'Congo' in country:
country = 'Congo'
elif country == 'Diamond Princess' or country == 'Laos' or country == 'MS Zaandam'\
or country == 'Holy See' or country == 'Timor-Leste':
return country, country
elif country == 'Korea, South' or country == 'South Korea':
country = 'Korea, Republic of'
elif country == 'Taiwan*':
country = 'Taiwan'
elif country == 'Burma':
country = 'Myanmar'
elif country == 'West Bank and Gaza':
country = 'Gaza'
else:
return country, country
country_obj = pycountry.countries.search_fuzzy(country)
continent_code = pc.country_alpha2_to_continent_code(country_obj[0].alpha_2)
continent = pc.convert_continent_code_to_continent_name(continent_code)
return country_obj[0].alpha_3, continent
def get_iso3(self, country):
return self.d[country]['code']
def get_continent(self,country):
return self.d[country]['continent']
def add_values(self,country):
self.d[country] = {}
self.d[country]['code'],self.d[country]['continent'] = self.get_country_details(country)
def fetch_iso3(self,country):
if country in self.d.keys():
return self.get_iso3(country)
else:
self.add_values(country)
return self.get_iso3(country)
def fetch_continent(self,country):
if country in self.d.keys():
return self.get_continent(country)
else:
self.add_values(country)
return self.get_continent(country)생성자
클래스 이름과 같은 함수를 생성자라고 한다. 클래스 내부에 init라는 함수를 만들면 객체를 생성할 때 처리할 내용을 작성할 수 있다.
try-except
try-except 구문은 오류 예외 처리 기법이다.
try:
...
except:
...try 블록 수행 중 오류가 발생하면 except 블록이 수행이 된다. 오류가 발생하지 않는다면 그냥 try 블록만 실행
try:
...
except 발생 오류:
...특정 오류만 발생했을 때 except를 실행한다.
try:
...
except 발생 오류 as 오류 메세지 변수:
...특정 발생 오류 및 오류 메세지의 내용 까지 알고 싶을 때 사용
pycountry 모듈
list(pycountry.countries)위 코드를 실행하면 다음과 같은 내용을 알 수 있다.
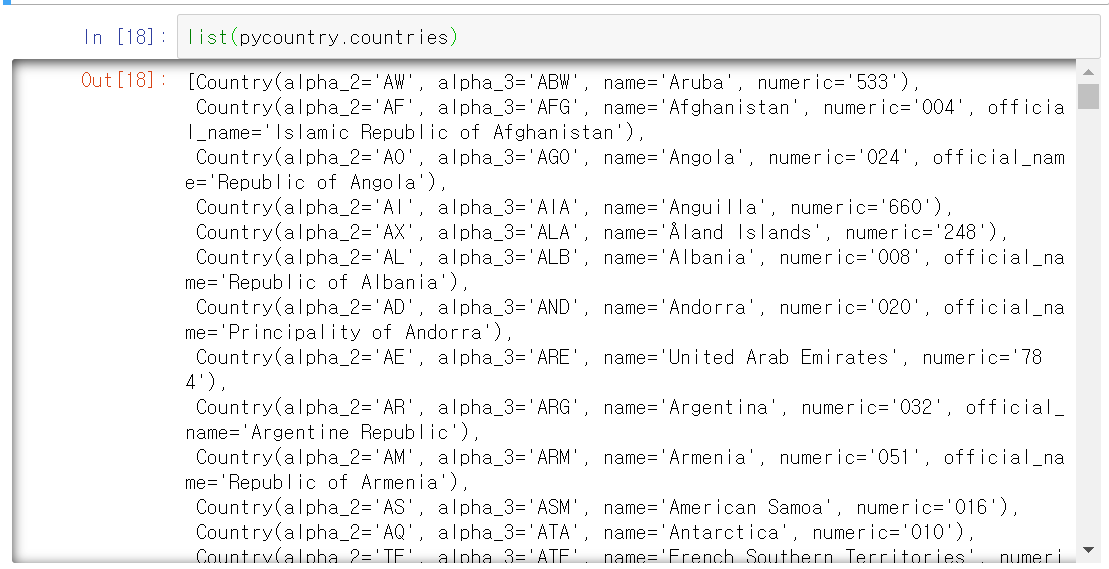 ![]
![]
각 나라들에 대한 ISO 3166이다.
정확한지는 모르겠지만 요약하자면
- alpha_2 : 나라 이름 중 대표 2글자
- alpha_3 : 나라 이름 중 대표 3글자 (KOR같은)
- name : 나라 이름
- numeric : 나라들에 대한 숫자로 표현한 것
countries.get()
country_obj = pycountry.countries.get(name='Benin')
print(country_obj)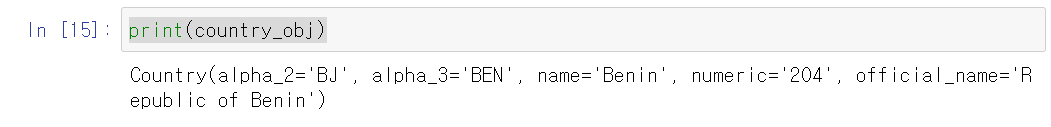
countries.get 함수를 통해 특정 나라에 대한 정보를 추출할 수 있다.
매개변수에는 name``, alpha_2, alpha_3, numeric`를 사용할 수 있다.
countries.search_fuzzy()
country_obj = pycountry.countries.get(name='Korea')
print(country_obj)한국에 대한 정보를 보고 싶어서 Korea로 countries.get 함수를 통해 검색해봤는데 결과는?

'None'라는 결과를 볼 수 있을 것이다. 이런 경우를 위해 search_fuzzy라는 함수가 있다. 이 함수에 대해 검색을 해보면
There’s also a “fuzzy” search to help people discover “proper” countries for names that might only actually be subdivisions. The fuzziness also includes normalizing unicode accents. There’s also a bit of prioritization included to prefer matches on country names before subdivision names and have countries with more matches be listed before ones with fewer matches:
해석을 해보면 세분화 될 수 있는 이름에 대해 적절한 국가를 찾는다고 한다.
한국을 예로 들면 원래 한국의 풀네임은 Korea, Republic of이다. 만약 get()함수로 검색을 하려면
country_obj = pycountry.countries.get(name='Korea, Republic of')로 해야하는데... 너무 길고 불편하다. 그럴 때를 위해 fuzzy를 쓸 수 있다.
if country_obj is None:
c = pycountry.countries.search_fuzzy(country)get()함수 결과물이 None일 때 search_fuzzy()를 쓰도록 조건문을 생성해줬다.
pycountry.countries.search_fuzzy('Korea')똑같이 Korea로 예를 들면 결과물은 다음과 같이 출력된다.

북한과 남한에 대한 정보를 모두 출력이 가능하다. 또 다른 예제로, 미국을 America로 해보면?
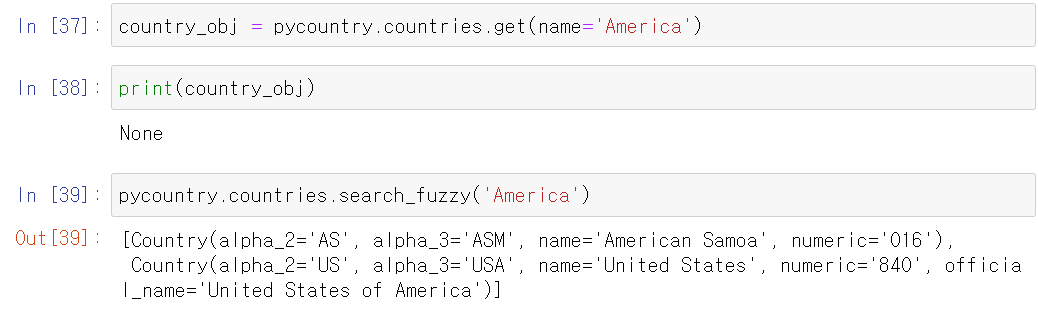
pycountry-convert 모듈
이 모듈은 pycountry 패키지에서 conversion 기능을 제공하는 확장 모듈이다.
continent_code = pc.country_alpha2_to_continent_code(country_obj.alpha_2)
continent = pc.convert_continent_code_to_continent_name(continent_code)country_alpha2_to_continent_code()
pycountry 패키지로 얻었던 alpha_2를 통해 대륙 이름으로 전환시켜준다.
| 대륙(영문) | 대륙(한글) | 대륙 코드 |
|---|---|---|
| Africa | 아프리카 | AF |
| Antarctica | 남극 | AN |
| Asia | 아시아 | AS |
| Australia(Oceania) | 호주(오세아니아) | OC |
| Europe | 유럽 | EU |
| North America | 북 아메리카 | NA |
| South America | 남 아메리카 | SA |
country_obj = pycountry.countries.search_fuzzy('Korea')[0]
continent_code = pc.country_alpha2_to_continent_code(country_obj.alpha_2)
한국(북한)의 대륙 코드인 AS를 출력한다.
convert_continent_code_to_continent_name()
대륙 코드를 대륙 이름으로 전환시켜준다.
continent = pc.convert_continent_code_to_continent_name(continent_code)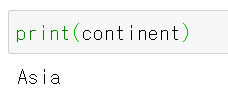
AS를 Asia로 변환 완료!
클래스 설명
treemap으로 시각화하는 코드에서
df_tm['continent'] = df_tm.apply(lambda x: obj.fetch_continent(x['Country_Region']), axis=1)df_tm이라는 데이터 프레임에 continent라는 열에 country_utils 클래스의 fetch_continent라는 함수를 사용했다. apply와 lambda에 대해서는 추후에 알아보도록 하자.
그럼 이 코드를 실행하면 어떻게 진행이 될까?
일단 이 클래스의 생성자를 확인하면
def __init__(self):
self.d = {}self.d라는 딕셔너리를 생성한다.
그리고 함수 실행 순서를 확인하기 위해 코드를 확인해보자.
.jpg)
1.fetch_continent()
def fetch_continent(self,country):
if country in self.d.keys():
return self.get_continent(country)
else:
self.add_values(country)
return self.get_continent(country)위 코드에서 확인했듯이 fetch_continent는 매개변수로 나라 이름을 받는다. 그래서 클래스의 d 딕셔너리에 해당하는 나라 이름의 key가 존재하는지 안하는지 판단하고 해당하는 조건문을 실행한다.
2.get_continent()
만약 d라는 딕셔너리의 key에 fetch_continent의 매개변수로 받은 나라 이름 country가 존재하면 그 나라에 해당하는 대륙 이름을 반환하려고 한다.
def get_continent(self,country):
return self.d[country]['continent']그래서 get_continent 함수를 통해 d 딕셔너리의 나라의 continent value를 반환한다.
3. add_values()
def add_values(self,country):
self.d[country] = {}
self.d[country]['code'],self.d[country]['continent'] = self.get_country_details(country)파이썬의 딕셔너리 자료구조를 처리하는 방법을 여기를 통해 알 수 있다.
맨 처음 fetch_continent()함수를 실행하면 d 딕셔너리에는 아무것도 존재하지 않는다.
self.d = {}이것을 육안으로 확인하기 위해 일반 변수 ex를 만들어 보았다.
ex = {}
print(ex)코드를 실행해보면
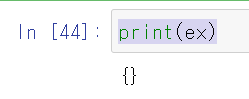
빈 딕셔너리 {}가 생성되는 것을 알 수 있다.
매개변수로 country를 받는데 korea를 받는다고 가정해보자.
self.d[country] = {}
ex['Korea'] = {} # country가 'korea'일 때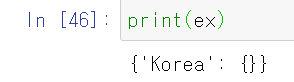
c언어에서의 배열처럼 []를 통해 key를 생성.
마지막으로 value를 생성하는 부분을 보자.
self.d[country]['code'],self.d[country]['continent'] = self.get_country_details(country)
ex['Korea']['code'], ex['Korea']['continent'] = 'KOR', 'Asia'get_conuntry_details() 함수 코드를 보면
# try 부분
return country_obj.alpha_3, continent
# except 부분
return country, country
# 또는
return country_obj[0].alpha_3, continent처럼 alpha_3과 country 를 반환한다. country 두개를 반환하는 것은 특이 케이스이다.
그래서 Korea는 alpha_3 = 'KOR', continent = 'Asia'라서 위와 같이 짰다.
결과는??

key = Korea에 대한 딕셔너리 생성!!
treemap 시각화
code
df.ConfirmedCases = np.abs(df.ConfirmedCases)
df_tm = df.copy()
date = df_tm.Date.max()
df_tm = df_tm[df_tm['Date']==date]
obj = country_utils()
df_tm.Province_State.fillna('',inplace=True)
df_tm['continent'] = df_tm.apply(lambda x: obj.fetch_continent(x['Country_Region']), axis=1)
df_tm["world"] = "World"
fig = px.treemap(df_tm, path=['world', 'continent', 'Country_Region'], values='ConfirmedCases',
color='ConfirmedCases', hover_data=['Country_Region'],
color_continuous_scale='dense', title='Current share of Worldwide COVID19 Cases')
fig.update_layout(width=700,template='seaborn')
fig.show()abs()
넘파이 모듈에 존재하는 abs()함수는 절대값으로 처리해주는 함수이다.
x = np.array([-2, -1, 0, 1, 2])
x # array([-2, -1, 0, 1, 2])
np.abs(x) # array([2, 1, 0, 1, 2])copy()
판다스의 함수로 데이터프레임의 복사본을 만들어주는 함수이다.
원래 df_1 = df_2로 복사를 해주면 원본 데이터 프레임이 수정될 시 복제된 데이터 프레임도 똑같이 수정이 된다.
하지만 pd.df.copy()로 복사를 해줄 경우, 이 명령을 실행할 당시의 데이터 프레임의 상태를 복제해주므로, 원본 데이터 프레임이 수정된다고 해서 복제된 데이터 프레임이 수정되진 않는다.
fillna()
fillna()의 이름을 보다보면 뭐가 한가지 생각이 날 것이다. fill과 na의 합성어처럼 보이는 이 이름은 결측치를 처리하기 위해 사용하는 함수이다.
이 함수는 pandas 패키지에 포함되어 있다. pandas는 두가지 타입의 결측치를 표현한다.
None: 파이썬 코드에서 누락된 데이터에 자주 사용되는 Python singleton 객체 개념NaN: 숫자가 아님을 뜻하는 결측치로 표준 IEEE 부동 소수점 표현을 사용하는 모든 시스템에서 인식되는 특수 부동 소수점 값을 의미
이fillna()함수를 한번 사용해보자.
import numpy as np
import pandas as pd
x = {'First' : [100,90,np.nan,95],
'Secont' : [30,45,56, np.nan],
'Third' : [np.nan, 40, 80, 98]}
df = pd.DataFrame(x)
세 개의 NaN 결측치를 포함하고 있는 데이터 프레임을 만들어 보았다.
df.fillna(0)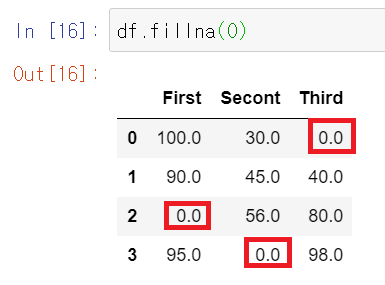
결측치를 0으로 채워보았다.
그럼 위에서 처럼 ''로 채워보자. 그리고 inplace가 무엇인지 알아보자
df.fillna('', inplace=True)0으로 채웠을 때 inplace 조건을 넣지 않았는데, 그럼 원본의 df를 확인해보면
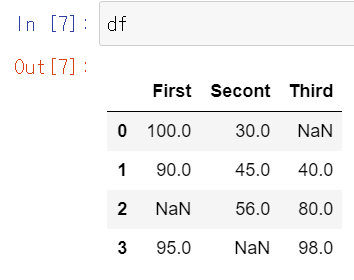
원본 데이터가 변하지 않았다.
그런데 inplace = True로 설정을 해주면 원본 데이터가 변환된다.

fillna()에는 method도 있다. pad와 bfill이라는 method가 있는데

pad는 앞의 숫자를 채워 넣고, bfill은 뒤의 숫자를 채워 넣는다. 맨 앞이나 뒤는 해당하는 숫자가 없기 때문에 NaN으로 남아져 있다.
treemap()
treemap()을 위해 plotly.express 패키지를 사용한다.
plotly에 대해서는 이 사이트에 가서 더 알아볼 수 있다.
import plotly.express as px
fig = px.treemap(df_tm, path=['world', 'continent', 'Country_Region'], values='ConfirmedCases',
color='ConfirmedCases', hover_data=['Country_Region'],
color_continuous_scale='dense', title='Current share of Worldwide COVID19 Cases')treemap은 데이터 프레임의 각 열을 treemap의 구역으로써 표현한다.
fig = px.treemap(
names = ["Eve","Cain", "Seth", "Enos", "Noam", "Abel", "Awan", "Enoch", "Azura"],
parents = ["", "Eve", "Eve", "Seth", "Seth", "Eve", "Eve", "Awan", "Eve"]
)
이 코드를 보면 treemap은 부모-자식으로 구분하여 시각화를 한다고도 생각을 할 수 있다. 이 중에 Noam을 보면, Noam의 부모는 Seth이고, Seth의 부모는 Eve이다.
.jpg)
그래서 fig에서 처럼 시각화로 표현이 된다. 이처럼 계층적인 데이터를 처리하는데 효과적인 시각화이다. 이러한 계층적 데이터는 데이터 프레임으로도 저장이 되는데, 이 계층적 데이터는 서로 다른 계층 수준에 해당하는 열에 저장이 된다. 이해가 어려우니 px.data.tips() 데이터로 설명을 해보자.
px.data.tips() 데이터는 성별, tip, bill 등을 포함하는 데이터 프레임이다.
이 때 treemap으로 표현하기 위해 path라는 파라미터를 사용한다. 이 path는 데이터 프레임의 열에 해당하는 값을 넣어주면 된다.
fig = px.treemap(df, path = ['day','sex', 'time'], values = 'total_bill').png)
그러면 'day', 'sex', 'time'의 값을 계층적으로 표현해준다. values는 각 해당 하는 계층의 총 'total_bill'의 값을 갖는다.
여기다가 'total_bill'의 차이를 쉽게 보기 위해 color까지 추가해준다면?
fig = px.treemap(df, path = ['day','sex', 'time'], values = 'total_bill', color = 'total_bill').png)
'total_bill'의 최소와 최대값 사이를 그라데이션 color로 표현이 가능하다.
범주형 변수에도 color이 가능한데... 딱히 유용성이 없다.
그리고 색깔도 커스터마이징 할 수 있는데, color_continuous_scale 파라미터를 사용한다.
fig = px.treemap(df, path = ['day','sex', 'time'], values = 'total_bill', color = 'total_bill', color_continuous_scale = 'hot')
#또는
fig = px.treemap(df, path = ['day','sex', 'time'], values = 'total_bill',
color = 'total_bill', color_continuous_scale = px.colors.sequential.Hot).png)
color_continuous_scale에는 다양한 Built_in color이 존재한다.
fig = px.colors.sequential.swatches_continuous().png)
hover_data에 대해서는 정확하게 파악하지는 못했다. 그런데 어떤 작용을 하는지는 아는데, plot 위에 커서를 두었을 때 뜨는 창에 대한 변화가 생긴다.
- hover_data 적용 전

- hover_data 적용 후
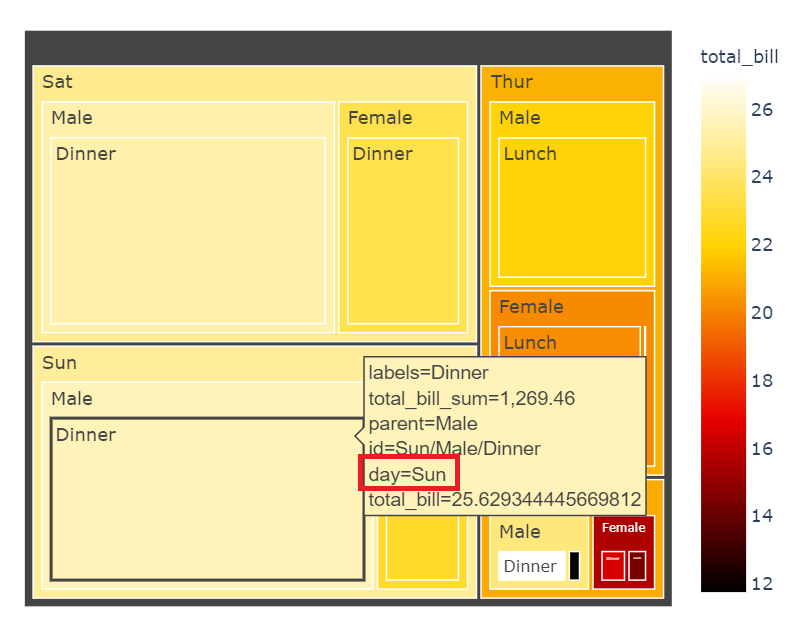
hover_data = ['day'] 파라미터를 적용시켰을 시 커서를 위에다 두면 해당하는 정보가 추가적으로 뜬다. 그런데 hover_data = ['sex']를 했을 때에는 또 아무 변화가 적용되지 않아 더 알아보아야 할 것 같다.
Daily 시각화
이번엔 날마다의 확진자 수와 사망자 수의 경향을 알기 위해서 Daily 기준으로 시각화를 해보려고 한다. 이번에는 treemap 시각화가 아닌 선 그래프를 이용하여 나타내볼 것이다.
code
데이터 준비
def add_daily_measures(df):
df.loc[0, 'Daily Cases'] = df.loc[0, 'ConfirmedCases']
df.loc[0, 'Daily Deaths'] = df.loc[0, 'Fatalities']
for i in range (1, len(df)):
df.loc[i, 'Daily Cases'] = df.loc[i,'ConfirmedCases'] - df.loc[i-1, 'ConfirmedCases']
df.loc[i, 'Daily Deaths'] = df.loc[i, 'Fatalities'] - df.loc[i-1, 'Fatalities']
df.loc[0, 'Daily Cases'] = 0
df.loc[0, 'Daily Deaths'] = 0
return dfloc를 이용하여 Daily Cases와 Daily Deaths를 다시 구한다.
데이터 프레임 생성
df_world = df.copy()
df_world = df_world.groupby('Date', as_index= False)['ConfirmedCases', 'Fatalities', 'Daily Cases', 'Daily Deaths'].sum()
df_world = add_daily_measures(df_world)Date를 기준으로 ConfirmedCases, Fatalities, Daily Cases, Daily Deaths의 합을 구한다.
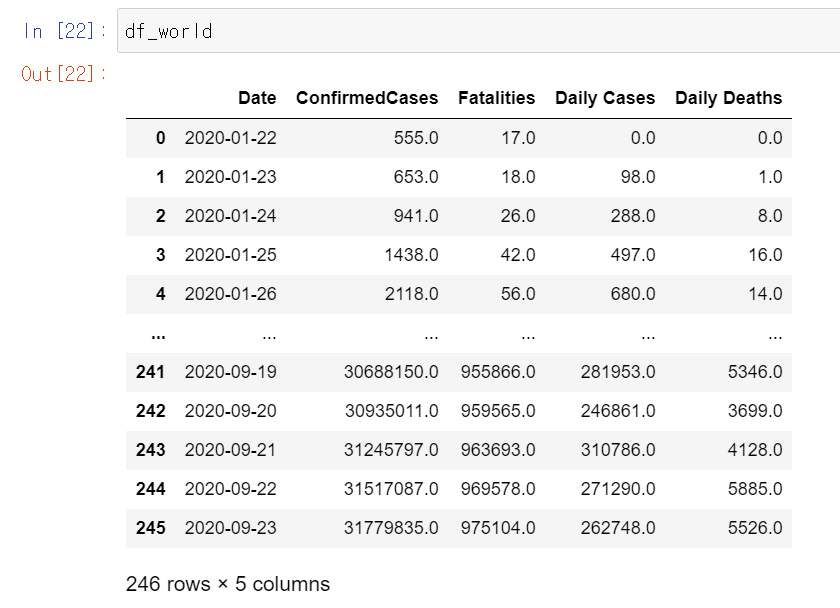
여기서 궁금한 점은 왜 Daily Cases가 ConfirmedCases와 비교했을 때 맞지 않는가... 이다. 분명히 위에서 lag_1을 구해서 ConfirmedCases에서 빼서 구한 값일 텐데... Fatalities는 정상적으로 잘 나오는데 ConfirmedCases만 이상하게 나온다.
그리고 위에서 만든 함수를 가지고 Data Frame를 재생성한다. 여기서 Daily Cases를 다시 구하기 때문에 일단 넘어간다.
시각화 그래프 생성 함수
def draw_graph(df,x,y1,y2,title,days=7):
colors = dict(case='#4285F4',death='#EA4335')
df['cases_roll_avg'] = df[y1].rolling(days).mean()
df['deaths_roll_avg'] = df[y2].rolling(days).mean()
fig = make_subplots(specs=[[{"secondary_y": True}]])
fig.add_trace(go.Scatter(name='Daily Cases',x=df[x],y=df[y1],mode='lines',
line=dict(width=0.5,color=colors['case'])),
secondary_y=False)
fig.add_trace(go.Scatter(name='Daily Deaths',x=df[x],y=df[y2],mode='lines',
line=dict(width=0.5,color=colors['death'])),
secondary_y=True)
fig.add_trace(go.Scatter(name='Cases: <br>'+str(days)+'-Day Rolling average',
x=df[x],y=df['cases_roll_avg'],mode='lines',
line=dict(width=3,color=colors['case'])),
secondary_y=False)
fig.add_trace(go.Scatter(name='Deaths: <br>'+str(days)+'-Day rolling average',
x=df[x],y=df['deaths_roll_avg'],mode='lines',
line=dict(width=3,color=colors['death'])),
secondary_y=True)
fig.update_yaxes(title_text='Cases',title_font=dict(color=colors['case']),secondary_y=False,nticks=5,
tickfont=dict(color=colors['case']),linewidth=2,linecolor='black',gridcolor='darkgray',
zeroline=False)
fig.update_yaxes(title_text='Deaths',title_font=dict(color=colors['death']),secondary_y=True,nticks=5,
tickfont=dict(color=colors['death']),linewidth=2,linecolor='black',gridcolor='darkgray',
zeroline=False)
fig.update_layout(title=title,height=400,width=700,
margin=dict(l=0,r=0,t=60,b=30),hovermode='x',
legend=dict(x=0.01,y=0.99,bordercolor='black',borderwidth=1,bgcolor='#EED8E4',
font=dict(family='arial',size=10)),
xaxis=dict(mirror=True,linewidth=2,linecolor='black',gridcolor='darkgray'),
plot_bgcolor='rgb(255,255,255)')
return fig이동평균(rolling)
측정값 x1, x2, ···가 계속해서 얻어질 때 각 값에서 시작하여 순서대로 일정 개수를 취하여 구한 상가(相加) 평균의 전체.
예를 들면 (x1+x2+x3)/3, (x2+x3+x4)/3, (x3+x4+x5)/3, ···
주식에서 자주 사용하는 5일 평균선같은 경우가 이에 해당한다.
여기서는 Daily Cases와 Daily Deaths의 7일 이동평균을 구하기 위해 pandas 패키지의 rolling 함수를 이용했다.
df['cases_roll_avg'] = df[y1].rolling(days).mean()
df['deaths_roll_avg'] = df[y2].rolling(days).mean()df의 y1과 y2 열의 7일 이동평균선을 df의 각각 새로 만든 열에다 추가한다.
make_subplots
plotly 모듈의 함수. 말 그대로 subplot들을 만들 수 있게 환경을 생성해주는 함수이다. 파라미터들을 몇가지 알아보자
https://plotly.com/python-api-reference/generated/plotly.subplots.make_subplots.html
파라미터들은 여기서 더 자세히 알 수 있다.
rows: subplot grid의 row 개수를 설정cols: subplot grid의 col 개수를 설정shared_xaxes: 데카르트(카티지언) subplot의 x축을 공유shared_yaxes: 데카르트(카티지언) subplot의타 y축을 공유specs: 각 subplot의 type을 명세한다.
리스트나 딕셔너리 형태로 표현한다.
ex1. specs=[[{},{}], [{'colspan':2}, None]]
ex2. specs=[[{'rowspan':2},{}], [None, {}]]
None은 빈 suplot 셀에 사용한다.
여기서 사용한secondary_y는 subplot 오른쪽에 추가적인 y축을 생성해준다.Daily Cases와Daily Deaths의 스케일이 다르기 때문에 설정.
add_trace
참고 : https://plotly.com/python/creating-and-updating-figures/
자 이제 subplot의 틀을 만들었으면 본격적인 subplot을 만들어보자.
다른 코드를 가지고 예를 들어보면
fig = make_subplots(rows=1, cols=3)
fig.add_trace(go.Scatter(y=[4, 2, 1], mode='lines'), row=1, col=3)
fig.add_trace(go.Bar(y=[2,1,3]), row=1, col=2)
fig.add_trace(go.Scatter(y=[4, 2, 1]), row=1, col=1)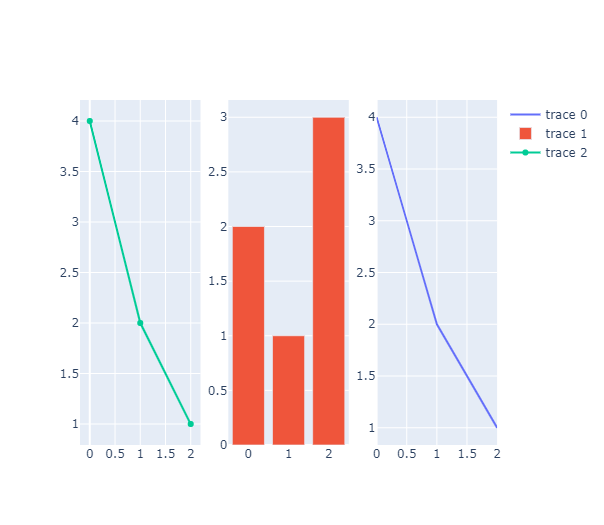
이렇게 여러개의 그래프를 한번에 나타내줄 수 있다. 물론 그래프끼리도 겹치기가 가능하다.
fig.add_trace(go.Bar(y=[2,1,3]), row=1, col=3)bar 그래프를 (1,3)에다 넣어주면?
.png)
보다시피 이렇게 겹치기가 가능!!
update_yaxes
tickfont=dict(color=colors['case']),linewidth=2,linecolor='black',gridcolor='darkgray',
zeroline=False)이번엔 y축을 수정하는 작업을 해보자

위에서 만든 그래프를 가지고 수정을 해보자

fig.update_yaxes(title_text='Cases',title_font=dict(color='#4285F4'),nticks=10,
tickfont=dict(color='#4285F4'),linewidth=2,linecolor='red',gridcolor='blue',
zeroline=False).png)
시각화
fig = draw_graph(df_world,
'Date',
'Daily Cases',
'Daily Deaths',
'<b>Worldwide: Daily Cases & Deaths</b><br> with 7-Day Rolling average').png)
이를 Bar Chart로 표현도 가능하다.
fig = go.Figure(data=[
go.Bar(name='Cases', x=df_world['Date'], y=df_world['Daily Cases']),
go.Bar(name='Deaths', x=df_world['Date'], y=df_world['Daily Deaths'])
])
fig.add_trace(go.Scatter(name='Cases:7-day rolling average',x=df_world['Date'],y=df_world['cases_roll_avg'],marker_color='black'))
fig.add_trace(go.Scatter(name='Deaths:7-day rolling average',x=df_world['Date'],y=df_world['deaths_roll_avg'],marker_color='darkred'))
fig.update_layout(barmode='overlay', title='Worldwide daily Case and Death count',hovermode='x',
template='seaborn',width=700,xaxis=dict(mirror=True,linewidth=2,linecolor='black',showgrid=False),
yaxis=dict(mirror=True,linewidth=2,linecolor='black'),legend=dict(orientation='h',x=0.1,y=-0.1))
fig.show()go.Figure()함수로 여러 Bar 그래프를 한 플롯 안에 표현이 가능하다.
파라미터 data로 리스트로 Figure을 묶어서 사용한다. 여기선 add_trace로 Scatter 그래프를 따로 표현했지만 Figure 안에 한번에 묶을 수 있다.
fig = go.Figure(data=[
go.Bar(name='Cases', x=df_world['Date'], y=df_world['Daily Cases']),
go.Bar(name='Deaths', x=df_world['Date'], y=df_world['Daily Deaths']),
go.Scatter(name='Cases:7-day rolling average',x=df_world['Date'],y=df_world['cases_roll_avg'],marker_color='black'),
go.Scatter(name='Deaths:7-day rolling average',x=df_world['Date'],y=df_world['deaths_roll_avg'],marker_color='darkred')
]).png)
결과물은 다음과 같이 Date에 따라 확진자수, 사망자 수를 Bar 차트로 확인할 수 있고, 7일 이동평균선을 Scatter 그래프로 확인 가능하다.
Map Visualization
Code
df_map = df.copy()
df_map['Date'] = df_map['Date'].astype(str)
df_map = df_map.groupby(['Date','Country_Region'], as_index=False)['ConfirmedCases','Fatalities'].sum()
df_map['iso_alpha'] = df_map.apply(lambda x: obj.fetch_iso3(x['Country_Region']), axis=1)
df_map['log(ConfirmedCases)'] = np.log(df_map.ConfirmedCases + 1)
df_map['log(Fatalities)'] = np.log(df_map.Fatalities + 1)
px.choropleth(df_map,
locations="iso_alpha",
color="log(ConfirmedCases)",
hover_name="Country_Region",
hover_data=["ConfirmedCases"] ,
animation_frame="Date",
color_continuous_scale=px.colors.sequential.dense,
title='Total Confirmed Cases growth(Logarithmic Scale)')전세계 지도를 가지고 시각화 하는 방법이다. 이 Visualization에서는 animation도 넣어줄 수 있다. Date별 확진자 또는 사망자 수의 변경을 Animation Frame으로 보기 위해 변수 datetime으로 변경해 주었던 Date를 다시 str로 재 수정했다.
그리고 날짜별 각 수를 확인하려면 날짜별, 나라별 사망자수, 확진자수를 합해줘야 해서 groupby를 통해 데이터 프레임을 수정해준다.
마지막으로 데이터 프레임에 alpha_3을 넣어주기 위해 위에서 작성했던 함수 fetch_iso3()함수를 이용했다.
axis=1은 열 방향으로 col의 연산을 적용한다. 그러면 axis=0은 row 방향 연산이라는걸 예상할 수 있다.
.png)
여기다가 scope 파라미터를 추가로 넣어주면?
자세하게 보고 싶은 곳의 공간 시각화를 볼 수 있다.
px.choropleth(df_map,
locations="iso_alpha",
scope='asia',
color="log(ConfirmedCases)",
hover_name="Country_Region",
hover_data=["ConfirmedCases"] ,
animation_frame="Date",
color_continuous_scale=px.colors.sequential.dense,
title='Total Confirmed Cases growth(Logarithmic Scale)').png)
이를 똑같이 사망자수에 적용해준다.
px.choropleth(df_map,
locations="iso_alpha",
color="log(Fatalities)",
hover_name="Country_Region",
hover_data=["Fatalities"],
animation_frame="Date",
color_continuous_scale=px.colors.sequential.OrRd,
title = 'Total Deaths growth(Logarithmic Scale)').png)
확진자 및 사망자 Top 10 나라
code
last_date = df.Date.max()
df_countries = df[df['Date']==last_date]
df_countries = df_countries.groupby('Country_Region', as_index=False)['ConfirmedCases','Fatalities'].sum()
df_countries = df_countries.nlargest(10,'ConfirmedCases')
df_trend = df.groupby(['Date','Country_Region'], as_index=False)['ConfirmedCases','Fatalities'].sum()
df_trend = df_trend.merge(df_countries, on='Country_Region')
df_trend.drop(['ConfirmedCases_y','Fatalities_y'],axis=1, inplace=True)
df_trend.rename(columns={'Country_Region':'Country', 'ConfirmedCases_x':'Cases', 'Fatalities_x':'Deaths'}, inplace=True)
df_trend['log(Cases)'] = np.log(df_trend['Cases']+1)# Added 1 to remove error due to log(0).
df_trend['log(Deaths)'] = np.log(df_trend['Deaths']+1)가장 최근 Date로 하여금 확진자 및 사망자 수 top 10 나라를 선별하려고 한다. Date.max()로 가장 최근 날짜를 뽑아낸 후, df_countries 데이터 프레임을 생성하는데 이는 df에서 Date.max()인 행만 뽑아내준다.
상위 10개의 나라를 추출하기 위해 pd.DataFrame.nlargest()를 사용.
heapq 모듈의 nlargest와는 기능은 같으나 pd.nlargest는 데이터 프레임에 사용
pd.nlargest : https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.nlargest.html
날짜별 top 10 나라들의 경향을 확인하기 위하여 df_trend 데이터프레임을 만들것인데, 우선 Date와 Country_Region별 ConfirmedCases 및 Fatalities의 합을 구한다.


이 두 데이터 프레임을 Country_Region을 기준으로 merge시키면
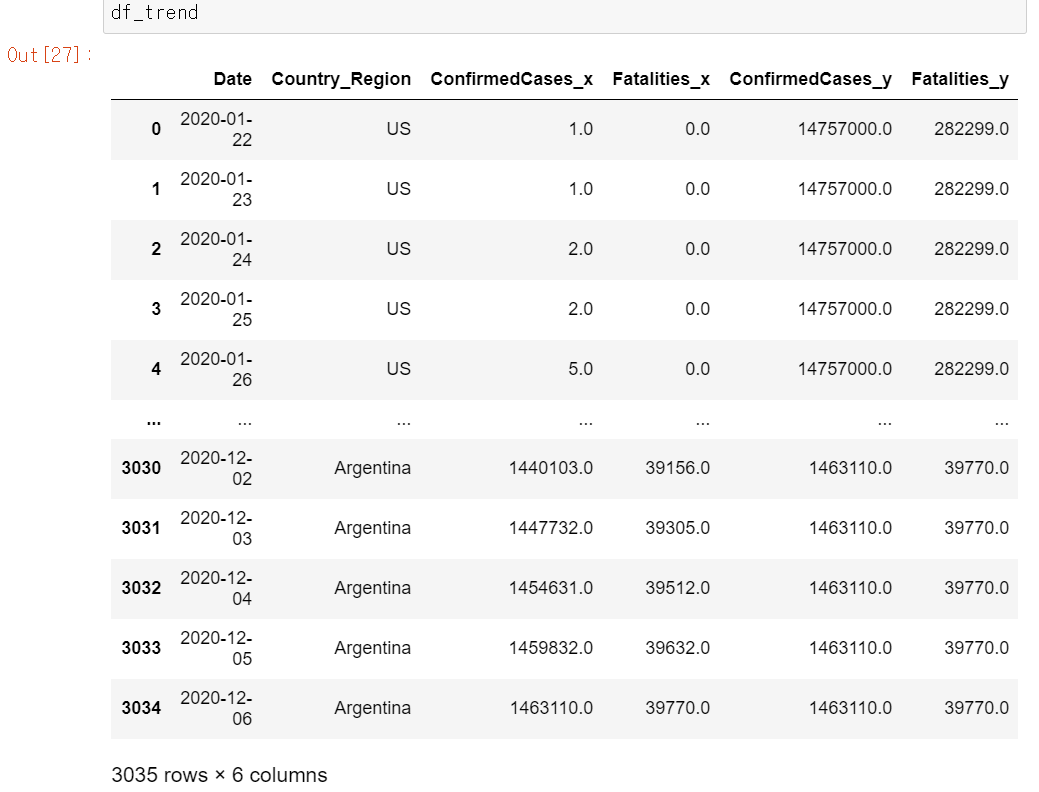
ConfirmedCases와 Fatalities가 중복되므로 _x, _y로 구별이 되는데 여기서 _y에 해당하는 해당 나라의 총 확진자, 사망자 수는 경향을 확인하는데 필요 없으므로 drop시킨다.
fig = px.line(df_trend, x='Date', y='Cases', color='Country', title='COVID19 Total Cases growth for top 10 worst affected countries')
fig.update_layout(hovermode='closest',template='seaborn',width=700,xaxis=dict(mirror=True,linewidth=2,linecolor='black',showgrid=False),
yaxis=dict(mirror=True,linewidth=2,linecolor='black'))
fig.show().png)
마찬가지로 사망자수도 line 시각화를 해주자.
fig = px.line(df_trend, x='Date', y='Deaths', color='Country', title='COVID19 Total Deaths growth for top 10 worst affected countries')
fig.update_layout(hovermode='closest',template='seaborn',width=700,xaxis=dict(mirror=True,linewidth=2,linecolor='black',showgrid=False),
yaxis=dict(mirror=True,linewidth=2,linecolor='black'))
fig.show().png)
- log 스케일
fig = px.line(df_trend, x='Date', y='log(Cases)', color='Country', title='COVID19 Total Cases growth for top 10 worst affected countries(Logarithmic Scale)')
fig.update_layout(hovermode='closest',template='seaborn',width=700,xaxis=dict(mirror=True,linewidth=2,linecolor='black',showgrid=False),
yaxis=dict(mirror=True,linewidth=2,linecolor='black'))
fig.show().png)
fig = px.line(df_trend, x='Date', y='log(Deaths)', color='Country', title='COVID19 Total Deaths growth for top 10 worst affected countries(Logarithmic Scale)')
fig.update_layout(hovermode='closest',template='seaborn',width=700,xaxis=dict(mirror=True,linewidth=2,linecolor='black',showgrid=False),
yaxis=dict(mirror=True,linewidth=2,linecolor='black'))
fig.show().png)
df_map['Mortality Rate'] = round((df_map.Fatalities/df_map.ConfirmedCases)*100, 2)Mortality Rate는 Fatalities / ConfirmedCases로 구하는데 소수점 세번째 자리에서 반올림하기 위해 round 함수를 사용. round함수에서 두번째 파라미터는 소수 몇번째 자리까지 표현할지를 의미한다.
px.choropleth(df_map,
locations='iso_alpha',
color='Mortality Rate',
hover_name='Country_Region',
hover_data=['ConfirmedCases', 'Fatalities'],
animation_frame='Date',
color_continuous_scale=px.colors.sequential.Magma_r,
title='Worldwide Daily Variation of Mortality Rate')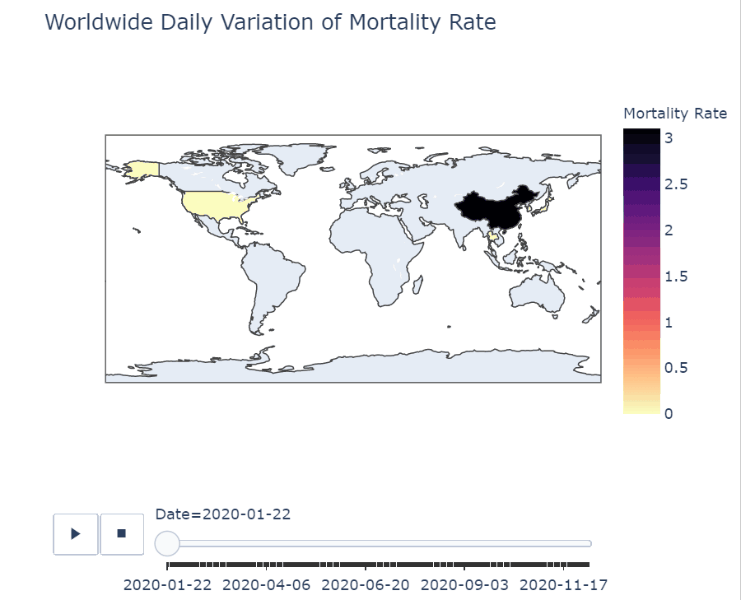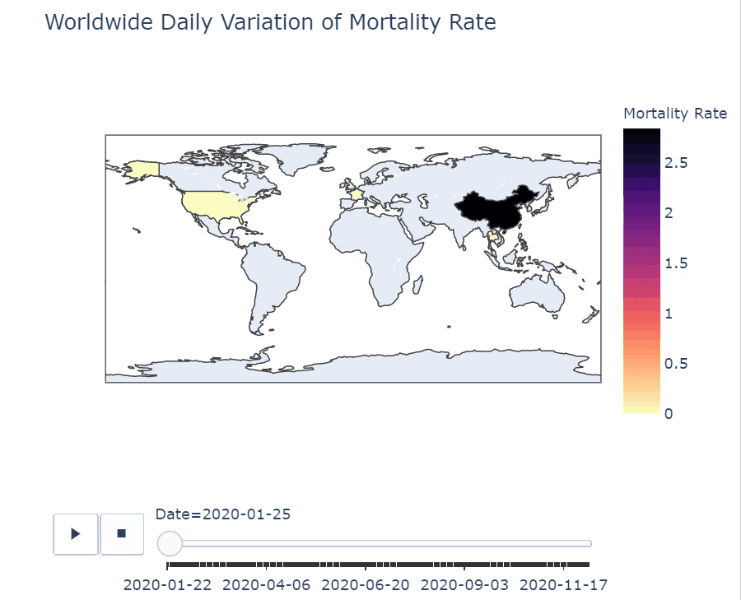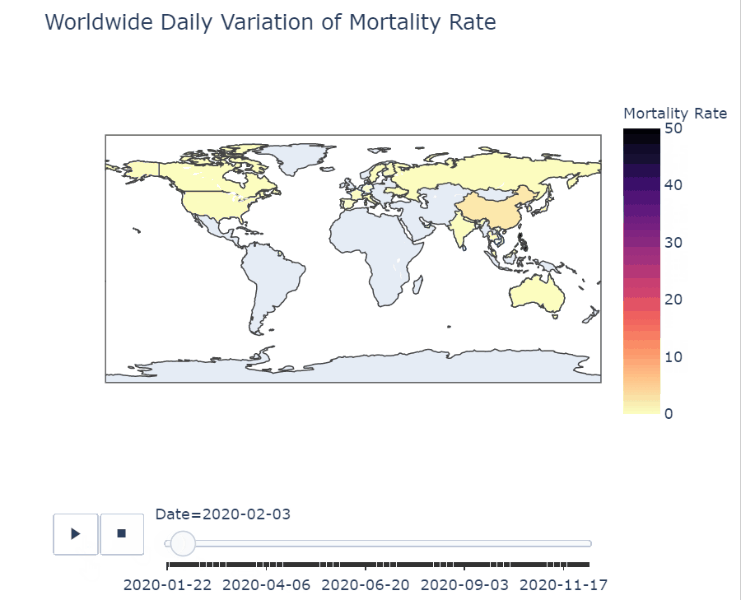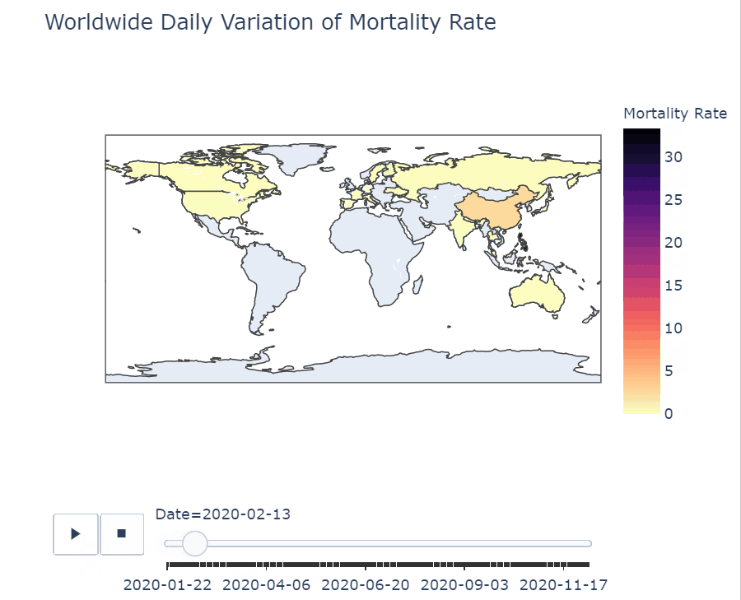
top 10 나라에서의 Mortality Rate를 구해서 경향을 시각화하자.
df_trend['Mortality Rate'] = round((df_trend.Deaths/df_trend.Cases)*100, 2)
fig = px.line(df_trend, x='Date', y='Mortality Rate', color='Country', title='Variation of Mortality Rate \n(Top 10 worst affected countries)')
fig.update_layout(hovermode='closest', template='seaborn', width=700,
xaxis=dict(mirror=True, linewidth=2, linecolor='black', showgrid=False),
yaxis=dict(mirror=True, linewidth=2, linecolor='black')).png)
USA COVID19 Visualization
우선 미국 주의 약어를 따로 딕셔너리 변수로 생성했다. 그리고 df 데이터에서 Country_Region이 US인 행만 따로 뽑아냈다.
us_state_abbrev = {
'Alabama': 'AL',
'Alaska': 'AK',
'American Samoa': 'AS',
'Arizona': 'AZ',
'Arkansas': 'AR',
'California': 'CA',
'Colorado': 'CO',
'Connecticut': 'CT',
'Delaware': 'DE',
'District of Columbia': 'DC',
'Florida': 'FL',
'Georgia': 'GA',
'Guam': 'GU',
'Hawaii': 'HI',
'Idaho': 'ID',
'Illinois': 'IL',
'Indiana': 'IN',
'Iowa': 'IA',
'Kansas': 'KS',
'Kentucky': 'KY',
'Louisiana': 'LA',
'Maine': 'ME',
'Maryland': 'MD',
'Massachusetts': 'MA',
'Michigan': 'MI',
'Minnesota': 'MN',
'Mississippi': 'MS',
'Missouri': 'MO',
'Montana': 'MT',
'Nebraska': 'NE',
'Nevada': 'NV',
'New Hampshire': 'NH',
'New Jersey': 'NJ',
'New Mexico': 'NM',
'New York': 'NY',
'North Carolina': 'NC',
'North Dakota': 'ND',
'Northern Mariana Islands':'MP',
'Ohio': 'OH',
'Oklahoma': 'OK',
'Oregon': 'OR',
'Pennsylvania': 'PA',
'Puerto Rico': 'PR',
'Rhode Island': 'RI',
'South Carolina': 'SC',
'South Dakota': 'SD',
'Tennessee': 'TN',
'Texas': 'TX',
'Utah': 'UT',
'Vermont': 'VT',
'Virgin Islands': 'VI',
'Virginia': 'VA',
'Washington': 'WA',
'West Virginia': 'WV',
'Wisconsin': 'WI',
'Wyoming': 'WY'
}
df_us = df[df['Country_Region'] == 'US']
df_us['Date'] = df_us['Date'].astype(str)
df_us['state_code'] = df_us.apply(lambda x: us_state_abbrev.get(x.Province_State, float('nan')), axis=1)
df_us['log(ConfirmedCases)'] = np.log(df_us['ConfirmedCases'] + 1)
df_us['log(Fatalities)'] = np.log(df_us.Fatalities + 1)df_us 데이터 프레임의 state_code에 위의 미국 주에 대한 약어를 넣어주기 위해 apply()함수에서 get()함수를 이용한다.
만약 us_state_abbrev 딕셔너리 안에 해당하는 미국 주가 존재하지 않으면 default값 float('nan')을 반환한다. 왜 float('nan')인지는 아직 모르겠다.

px.choropleth(df_us,
locationmode='USA-states',
scope="usa",
locations="state_code",
color='log(ConfirmedCases)',
hover_name='Province_State',
hover_data=["ConfirmedCases"],
animation_frame='Date',
color_continuous_scale=px.colors.sequential.Darkmint,
title='Total Cases growth for USA(Logarithmic Scale)')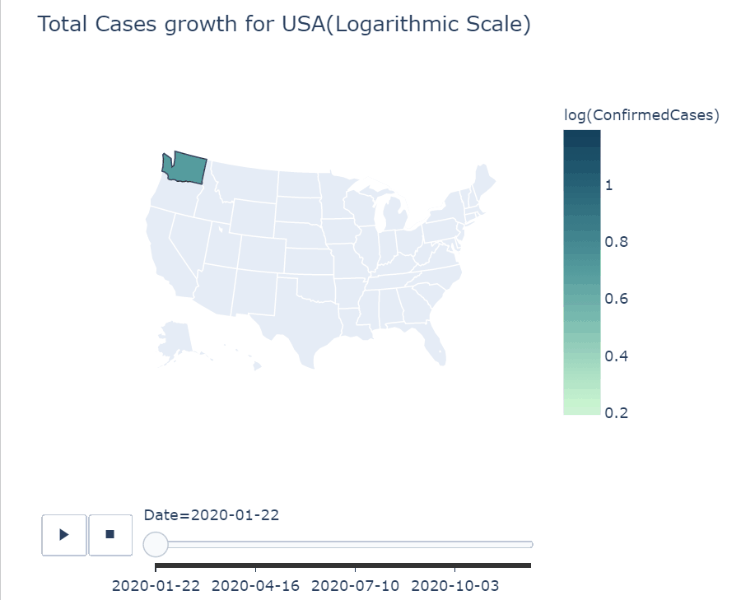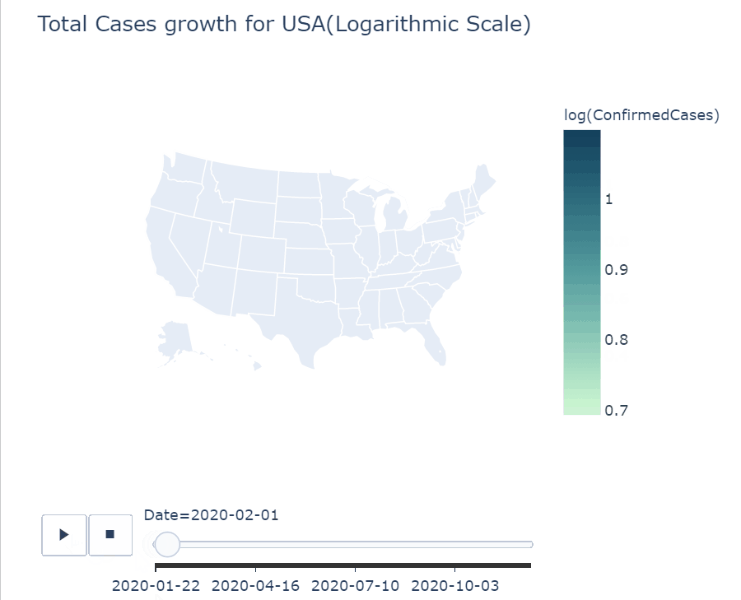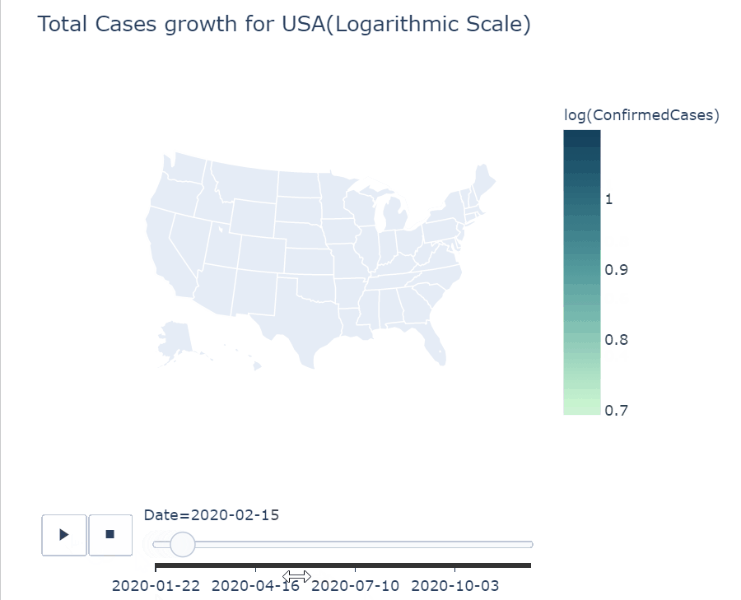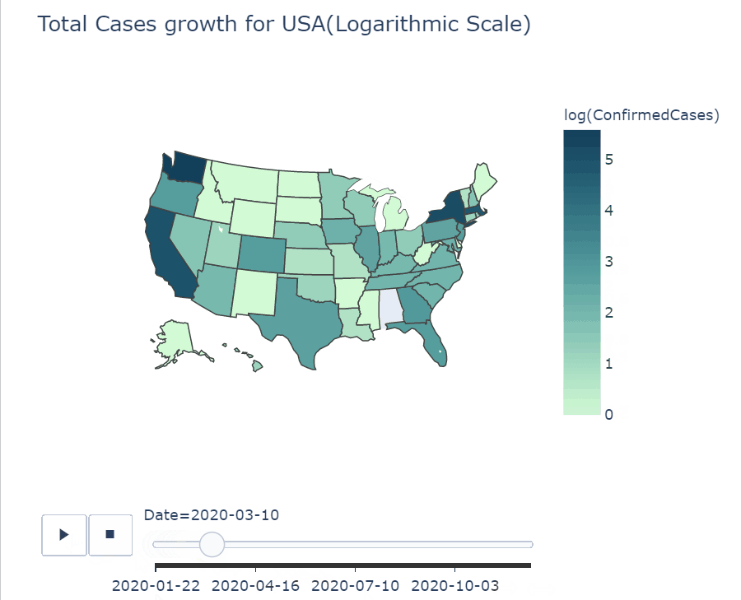
만약 locations 파라미터를 주의 풀네임으로 하면
locations="Province_State",.png)
적용이 안된다.
px.choropleth(df_us,
locationmode='USA-states',
scope='usa',
locations='state_code',
color='log(Fatalities)',
hover_name='Province_State',
hover_data=['Fatalities'],
animation_frame='Date',
color_continuous_scale=px.colors.sequential.OrRd,
title="Total deaths growth for USA(Logarithmic Scale)").png)
파이썬에서는 pandas 모듈에서 데이터 프레임에 적용되는 query라는 메서드를 지원한다.
이 메서드는 조건식을 문자열로 입력받아 해당 조건에 만족하는 데이터프레임의 행을 추출하는 함수이다.
df_usa = df.query("Country_Region=='US'")
df_usa = df_usa.groupby('Date', as_index=False)['ConfirmedCases', 'Fatalities', 'Daily Cases', 'Daily Deaths'].sum()
df_usa = add_daily_measures(df_usa)위에서 query함수를 통해 country == 'US'에 해당하는 행을 출력했다.
이는
df_us = df[df['Country_Region'] == 'US']와 같다.
이를 Bar chart로 표현할 수 있다.
fig = go.Figure(data=[
go.Bar(name='Cases', x=df_usa['Date'], y=df_usa['Daily Cases']),
go.Bar(name='Deaths', x=df_usa['Date'], y=df_usa['Daily Deaths'])
])
fig.update_layout(barmode='overlay', title='Daily Cases and Deaths count(USA)')
fig.update_layout(hovermode='closest', template='seaborn',width=700,
xaxis=dict(mirror=True, linewidth=2, linecolor='black',showgrid=False),
yaxis=dict(mirror=True, linewidth=2, linecolor='black')).png)
