# EDA
1. read_csv(), info(), shape, head()
: csv 파일을 Pandas DataFrame class 로 불러오기 위해 read_csv() 매서드를 활용
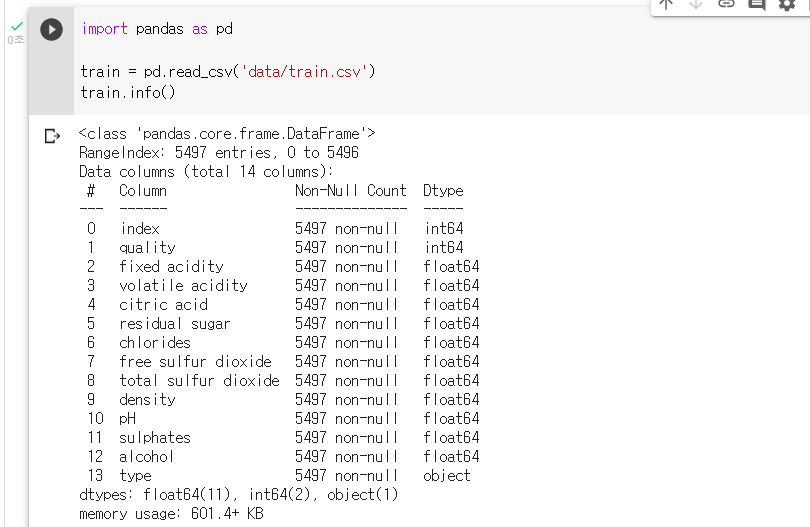
df = pd.read_csv(‘경로'): info() 매서드를 활용하여 데이터의 피쳐수와 컬럼명, 결측치여부, Dtype 에 대한 정보를 알 수 있다.
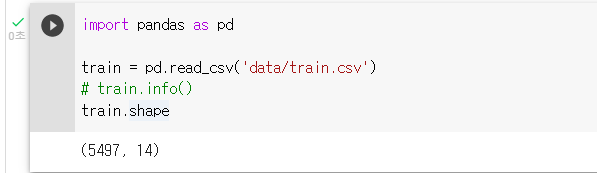
df.info(): shape 어트리뷰트를 쓰면 데이터의 행갯수, 열갯수를 출력해 데이터의 크기를 파악할 수 있다.
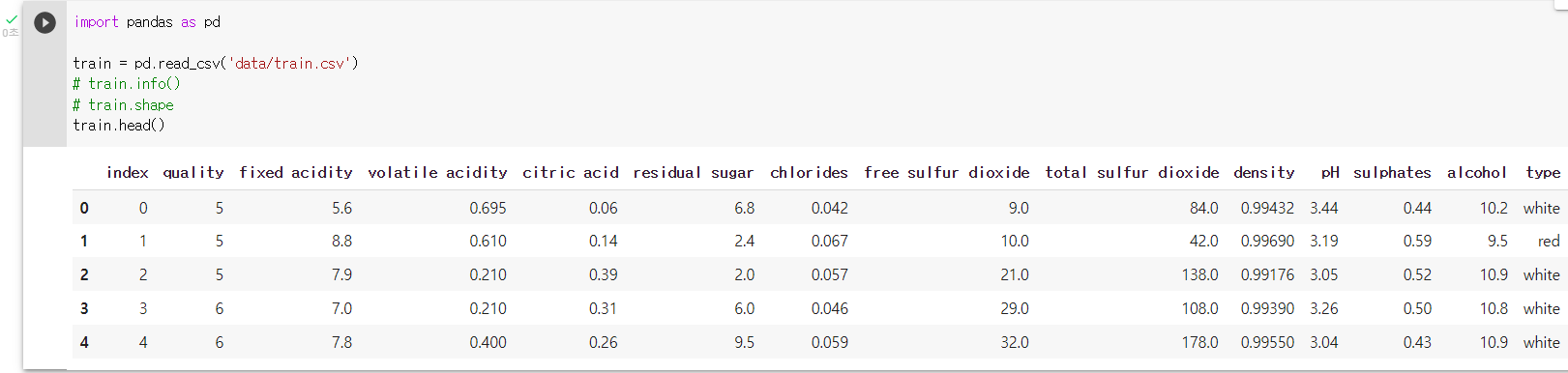
df.shape: head() 매서드를 통해 데이터의 대략적인 정보를 알 수 있다.
df.head(): read_csv() 매서드로 train.csv 파일을 df class 로 불러오기
train = pd.read_csv('data/train.csv'): info() 매서드로 데이터의 정보를 확인하기
train.info()
: shape 어트리뷰트로 행, 열을 파악하기train.shape
: head() 매서드로 데이터의 각 컬럼의 정보를 조사하기train.head()
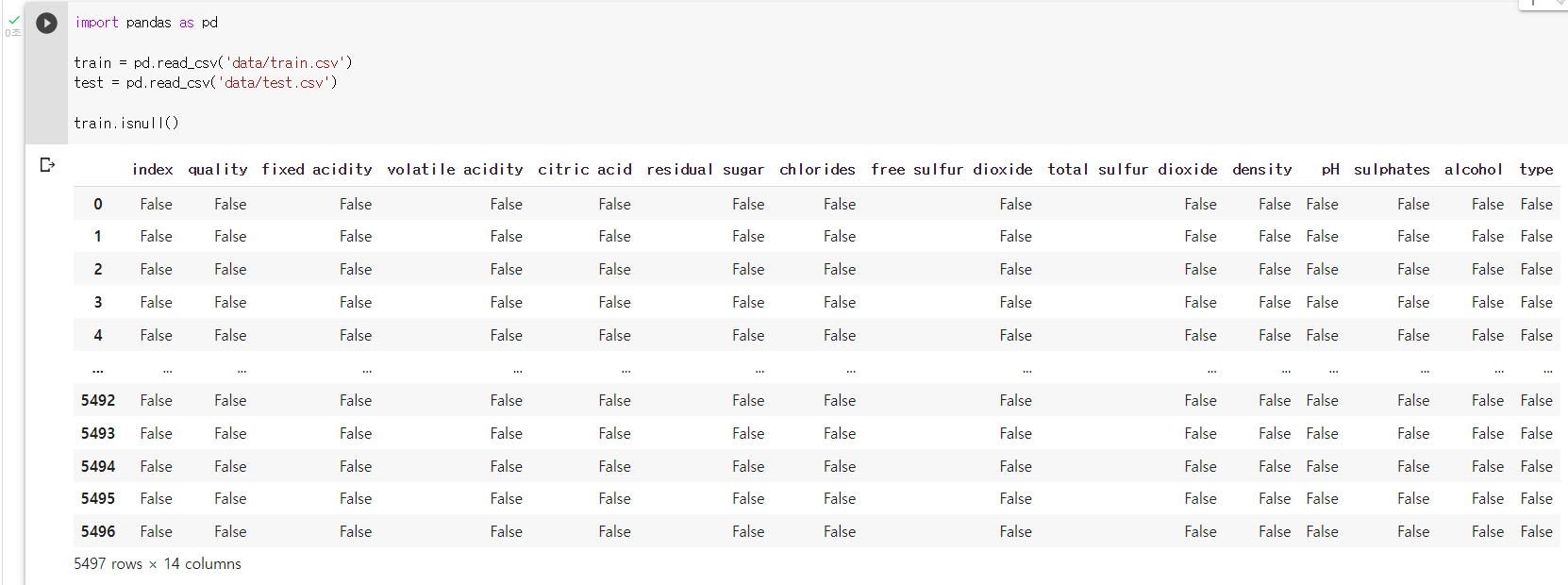
2. 결측치 유무 확인하기 isnull().sum()
df.isnull().sum(): isnull() 매서드로 결측치 존재여부를 확인하기
train.isnull()
test.isnull()
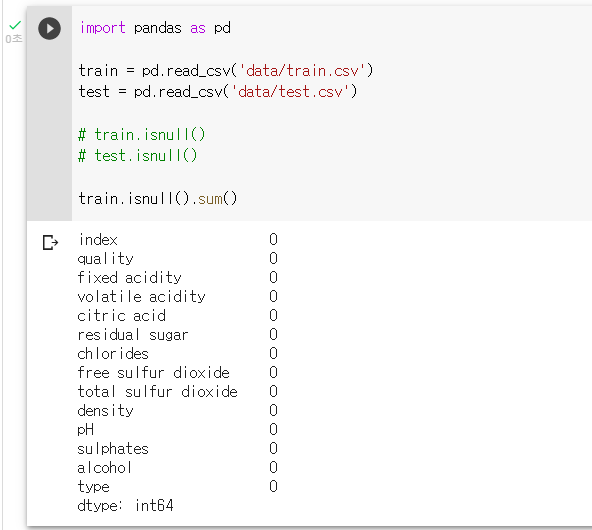
: sum() 매서드로 결측치의 갯수를 출력하기train.isnull().sum()
test.isnull().sum()
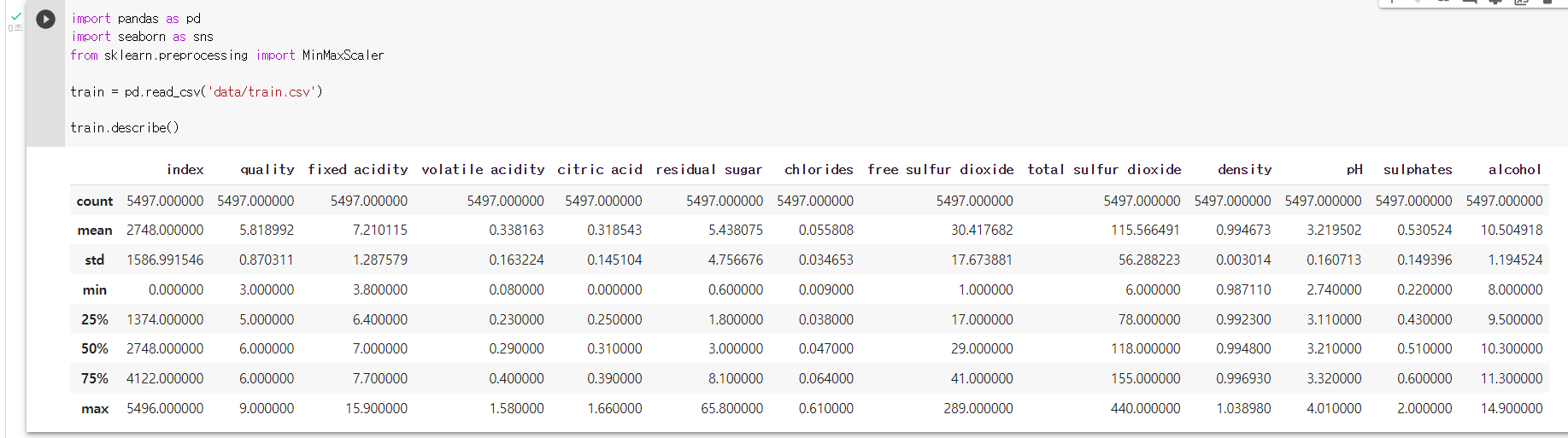
3. 수치데이터 특성 보기 (describe())
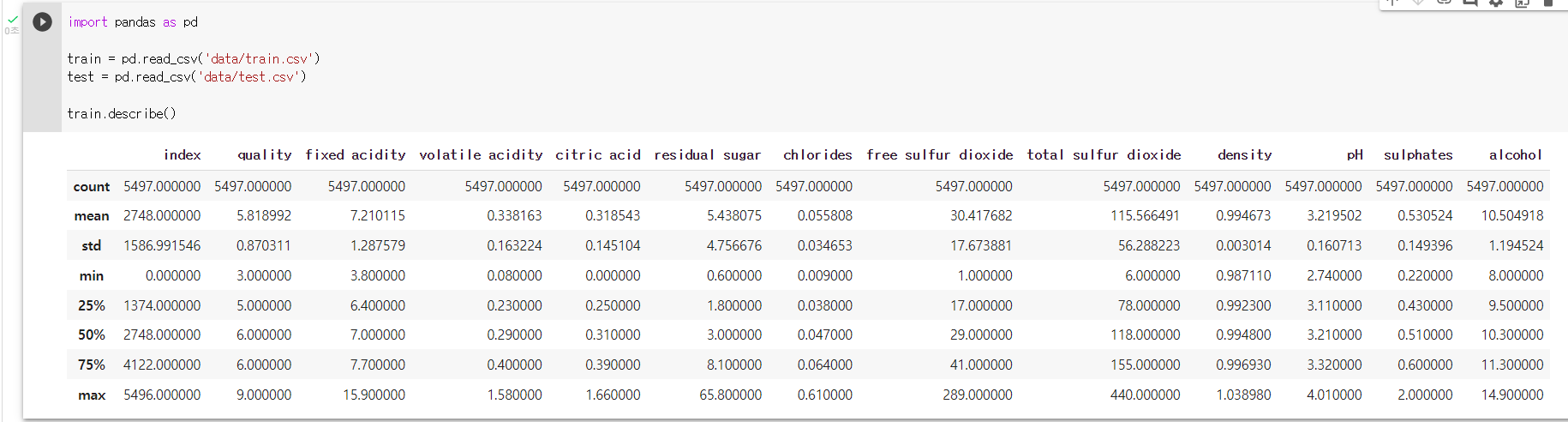
: DataFrame에 describe() 메서드를 실행시키면 각 열에 대해 요약이 수행된다.
: 결측치는 제외하고 수치형 데이터에 한해 데이터 요약이 수행된다.
: 기본적으로 count, mean, std, min , 1 분위수, 2 분위수, 3 분위수, max 값이 출력된다.
df.describe(): describe() 매서드로 데이터 요약을 출력하기
train.describe()
4. 타깃 변수 분포 시각화 seaborn distplot()
: matplotlib, seaborn 라이브러리로 시각화를 출력할 수 있다.
: 시각화 결과를 통해 머신러닝 방향성을 잡을 수 있다.
import matplotlib
import matplotlib.pyplot as plt
import seaborn as sns: 시각화를 진행할 때는 보통 copy() 매서드로 복사본을 생성한 후 진행한다.
dfcopy = df.copy(): seaborn 의 distplot() 매서드를 이용한다.
sns.distplot(df['피쳐명'], kde=True, bins=None)
df['피쳐명']: 출력하고자 하는 컬럼
kde: '그래프에 선을 출력할지 여부'
bins: '출력할 막대그래프 갯수': matplotlib 의 axis() 매서드로 그래프 축의 최솟값, 최댓값을 지정할 수 있다.
plt.axis(['x 축 최솟값, x 축 최댓값, y 축 최솟값, y 축 최댓값']): matplotlib 의 title() 매서드로 그래프의 제목을 지정할 수 있다.
plt.title('제목'): matplotlib 의 show() 매서드로 그래프를 출력할 수 있다.
plt.show(): 한글 글꼴 다운로드
!sudo apt-get install -y fonts-nanum !sudo fc-cache -fv !rm ~/.cache/matplotlib -rf: 데이터 다운로드
!wget 'https://bit.ly/3i4n1QB' import zipfile with zipfile.ZipFile('3i4n1QB', 'r') as existing_zip: existing_zip.extractall('data'): 라이브러리 불러오기
import pandas as pd # 시각화에 필요한 라이브러리를 import import matplotlib import matplotlib.pyplot as plt import seaborn as sns# 쥬피터노트에서 결과를 출력하도록 설정 %matplotlib inline# 글꼴 설정 plt.rc('font', family='NanumBarunGothic')# 마이너스 기호 출력 plt.rc('axes', unicode_minus=False)# 분석에 문제가 없는 경고 메세지는 숨긴다. import warnings warnings.filterwarnings('ignore'): 데이터 불러오기
train = pd.read_csv('data/train.csv'): copy() 매서드로 학습 데이터의 복사본을 생성하기
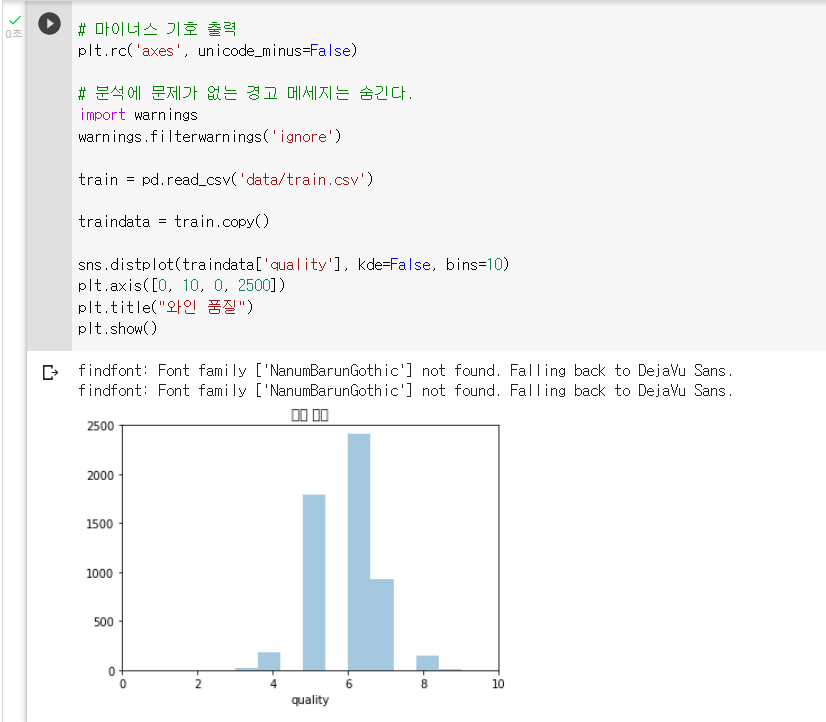
traindata = train.copy(): 타깃 변수(와인품질)의 분포를 시각화하기
1) seaborn 의 displot() 으로 어느 변수를 시각화할지 옵션에 지정하기
2) matplot 의 axis() 로 각 축의 최소, 최대 값을 지정하기
3) matplot 의 title() 로 그래프의 제목을 지정하기
4) matplot 의 show() 로 그래프를 출력하기sns.distplot(traindata['quality'], kde=False, bins=10) plt.axis([0, 10, 0, 2500]) # [x 축 최솟값, x 축 최댓값, y 축 최솟값, y 축 최댓값] plt.title("와인 품질") # 그래프 제목 지정 plt.show() # 그래프 그리기
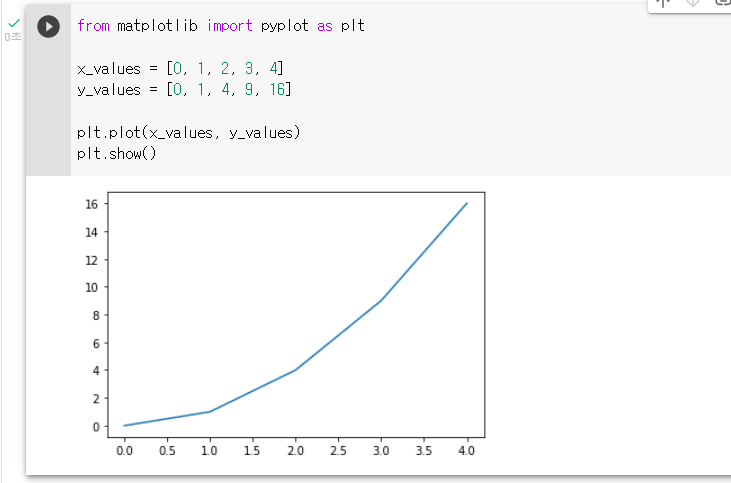
5. Matplotlib 선 그래프 그리기 (plot())
: 데이터 시각화 라이브러리 Matplotlib를 불러오고 plot() 매서드를 활용해서 그래프 그려보기
from matplotlib import pyplot as plt
plt.plot(x, y)
plt.show(): x축 지점의 값들로 정할 리스트를 생성하기
x_values = [0, 1, 2, 3, 4]: y축 지점의 값들로 정할 리스트를 생성하기
y_values = [0, 1, 4, 9, 16]: plot() 매서드를 활용해 그래프를 그리기
plt.plot(x_values, y_values): 그래프를 화면에 보여주기
plt.show()
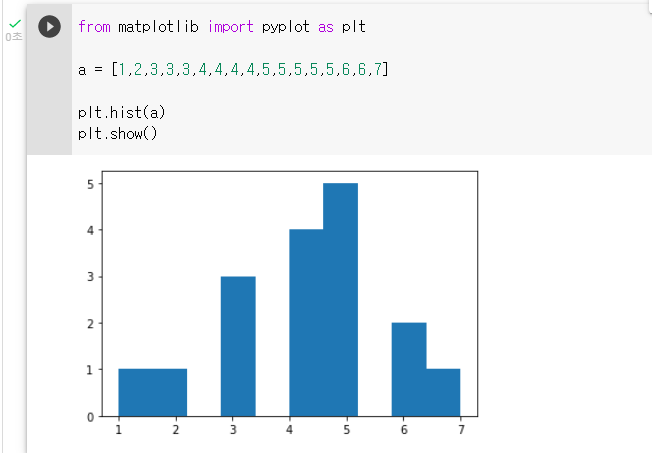
6. Matplotlib 히스토그램 그리기 (hist())
: 히스토그램 (Histogram) 은 도수분포표를 그래프로 나타낸 것
: 변수들의 분포정도를 볼 때 유용
from matplotlib.pyplot as plt
plt.hist(x, y)
plt.show(): 변수 분포를 갖는 리스트를 생성하기
a = [1,2,3,3,3,4,4,4,4,5,5,5,5,5,6,6,7]: hist() 매서드를 활용해 그래프를 그리기
plt.hist(a): 그래프를 화면에 보여주기
plt.show()
# 전처리
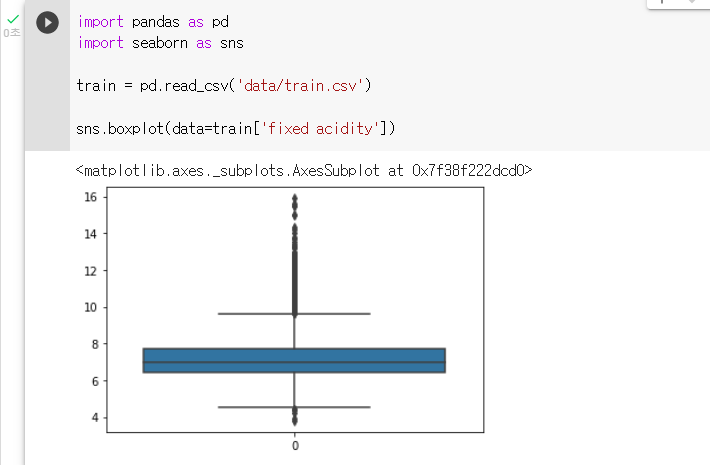
1. 이상치 탐지 seaborn_boxplot()
: 이상치를 탐지하는 대표적인 방법은 IQR (Inter Qunatile Range) 로, 사분위 값의 편차를 이용
: 이를 boxplot 그래프로 볼 수 있다.
import seaborn as sns
sns.boxplot(data=train['fixed acidity']): 라이브러리 불러오기
import pandas as pd import seaborn as sns: read_csv() 매서드로 train.csv 파일을 df class 로 불러오기
train = pd.read_csv('data/train.csv'): boxplot() 매서드로 'fixed acidity' 피쳐의 이상치를 확인하는 코드 작성하기
2. 이상치 제거 IQR(criterion='mse')
: IQR을 통해서 이상치를 제거
: IQR이란, 3분위수(75%에 위치한 값) - 1분위수(25%에 위치한 값)를 의미
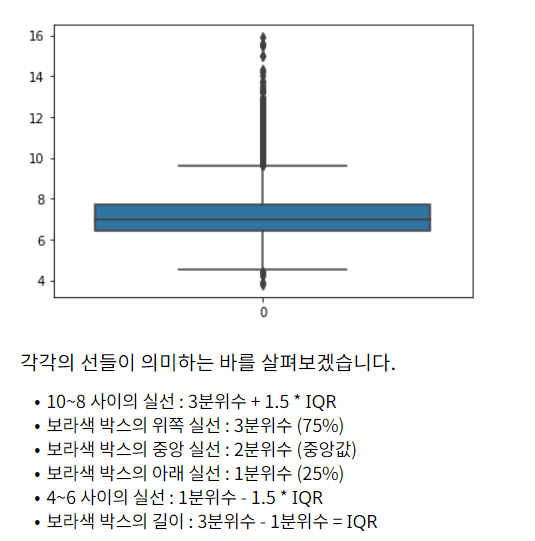
: boxplot의 4와 6사이의 실선보다 작고, 8과 10 사이의 실선보다 큰 데이터 포인트들을 이상치로 판단하고 제거해보기
: "fixed acidity"가 25%인 값을 "quantile_25" 라는 변수에 만들기
quantile_25 = np.quantile(train['fixed acidity'], 0.25): "fixed acidity"가 75%인 값을 "quantile_75" 라는 변수에 만들기
quantile_75 = np.quantile(train['fixed acidity'],0.75): quantile_75와 quantile_25의 차이를 "IQR"이라는 변수에 만들기
IQR = quantile_75 - quantile_25: quantile_25보다 1.5 * IQR 작은 값을 "minimum"이라는 변수에 만들기
minimum = quantile_25 - 1.5 * IQR: quantile_75보다 1.5 * IQR 큰 값을 "maximum"이라는 변수에 만들기
maximum = quantile_75 + 1.5 * IQR: "fixed acidity"가 minimum보다 크고, maximum보다 작은 값들만 "train2"에 저장하기
train2 = train[(minimum <= train['fixed acidity']) & (train['fixed acidity'] <= maximum)]: train2.shape를 통해서, 총 몇개의 행이 되었는지 확인하기
train2.shape
: 몇개의 이상치가 있는지도 계산해보기train.shape[0] - train2.shape[0]
3. 수치형 데이터 정규화 MinMaxScaler()
: 의사결정 나무나, 랜덤포레스트 같은 “트리 기반의 모델”들은 대소 비교를 통해서 구분하기 때문에, 숫자의 단위에 크게 영향을 받지 않는다.
: 하지만 Logistic Regression, Lasso 등과 같은 “평활 함수 모델”들은 숫자의 크기와 단위에 영향을 많이 받는다.
: 따라서, 수치형 데이터 정규화를 통해 모든 모델에 잘 어울리는 데이터를 만들어준다.
: "Min Max Scailing"기법은 가장 작은 값은 0으로, 가장 큰 값은 1로 만들어주는 방법이다.
: 그 사이의 값들은 비율에 따라서 0~1 사이에 분포하게 된다.
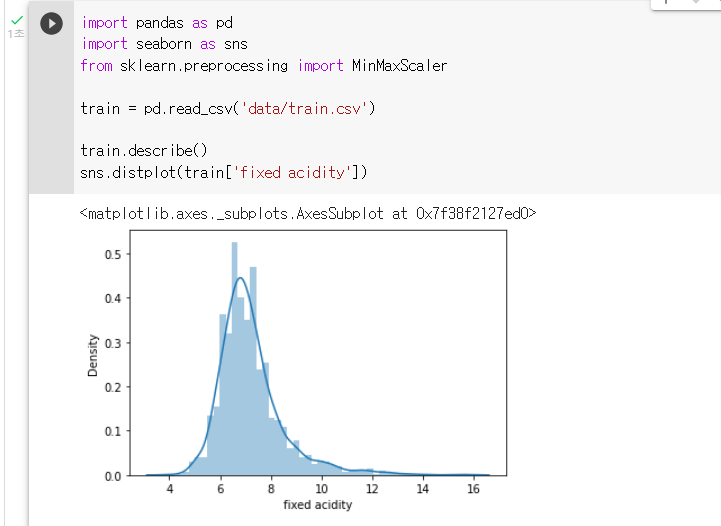
: describe를 통해 "fixed acidity"의 데이터의 분포가 어떻게 생겼는지 짐작해보기
train.describe()
: seaborn의 displot을 통해 "fixed acidity"의 distplot을 그려보기sns.distplot(train['fixed acidity'])
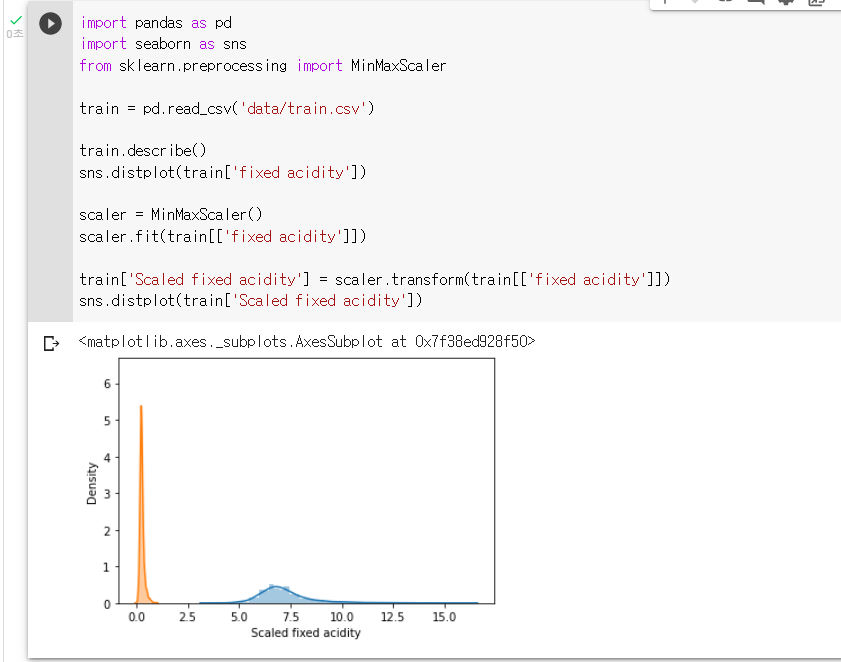
: MinMaxScaler를 "scaler"라는 변수에 지정하기scaler = MinMaxScaler(): "scaler"를 학습시켜주기
scaler.fit(train[['fixed acidity']]): "scaler"를 통해 train의 "fixed acidity"를 바꾸어 "Scaled fixed acidity"라는 column에 저장하기
train['Scaled fixed acidity'] = scaler.transform(train[['fixed acidity']]): seaborn의 displot을 통해 "Scaled fixed acidity"의 distplot을 그려보기
sns.distplot(train['Scaled fixed acidity'])
4. 원-핫 인코딩 OneHotEncoder()
: 컴퓨터는 “문자”로 된 데이터를 학습할 수 없기 때문에 “type”같은 피쳐들은 컴퓨터가 읽어서 학습 할 수 있도록 “인코딩” 해주어야 한다.
: 인코딩의 방법 중 하나인 One-Hot Encoding은 말 그대로, ‘하나만 Hot 하고, 나머지는 Cold한 데이터”라는 의미이다.
: 자신에게 맞는 것은 1로, 나머지는 0으로 바꾸어 줍니다.
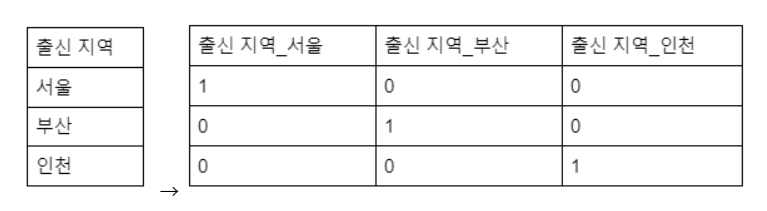
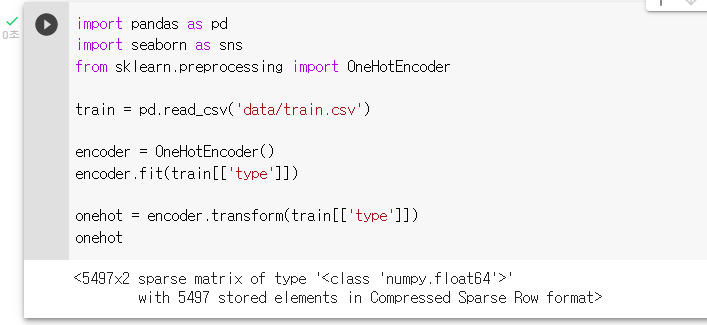
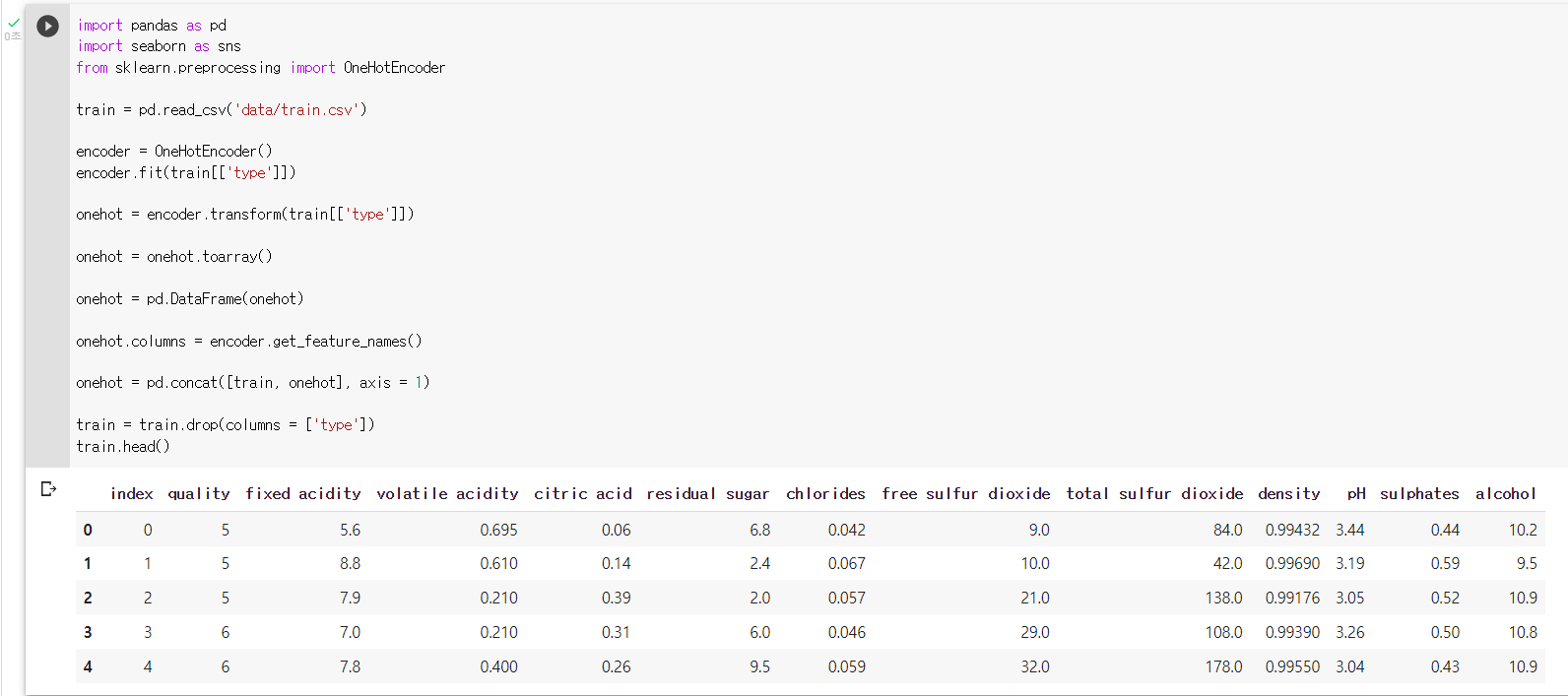
: "OneHotEncoder"를 "encoder"라는 변수에 저장해보기
encoder = OneHotEncoder(): "encoder"를 사용해 train의 "type" 피쳐를 학습시켜보기
encoder.fit(train[['type']]): "encoder"를 사용해 train의 "type"피쳐를 변환해 "onehot"이라는 변수에 저장해보기
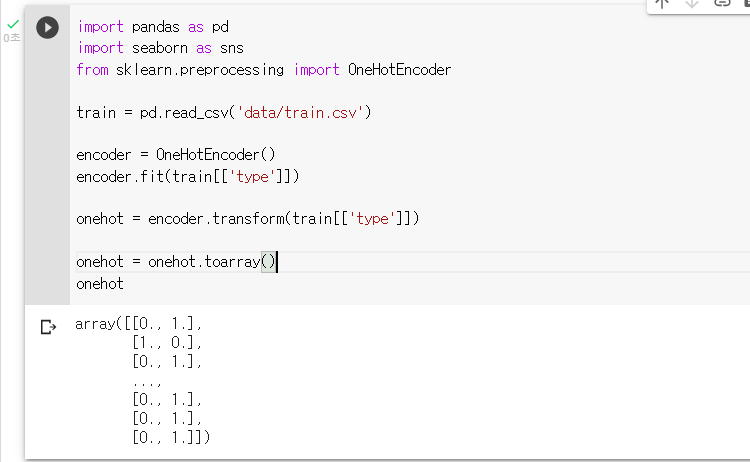
onehot = encoder.transform(train[['type']])
: "onehot"이라는 변수를 array 형태로 변환해 보기onehot = onehot.toarray()
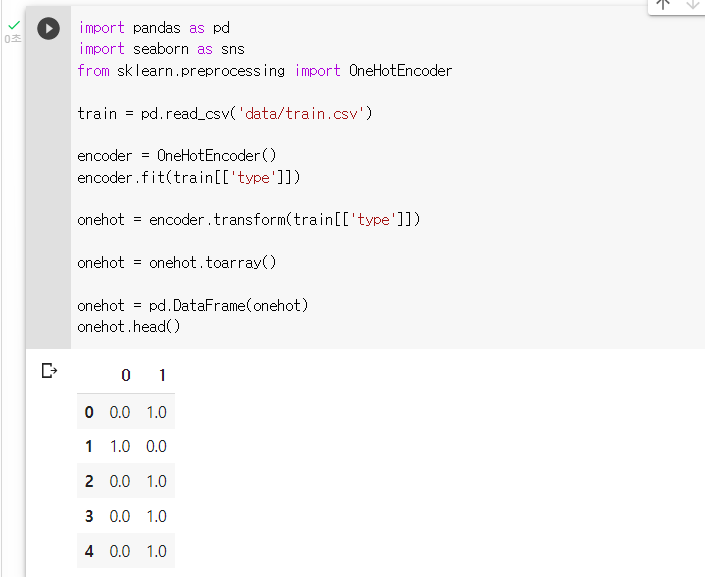
: "onehot"이라는 변수를 DataFrame 형태로 변환해 보기onehot = pd.DataFrame(onehot)
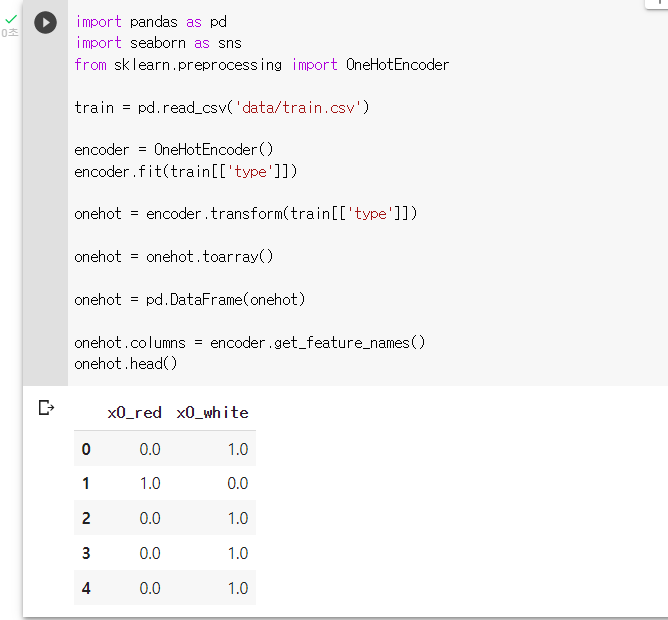
: encoder의 "get_feature_names()"를 사용해 column 이름을 바꿔보기onehot.columns = encoder.get_feature_names()
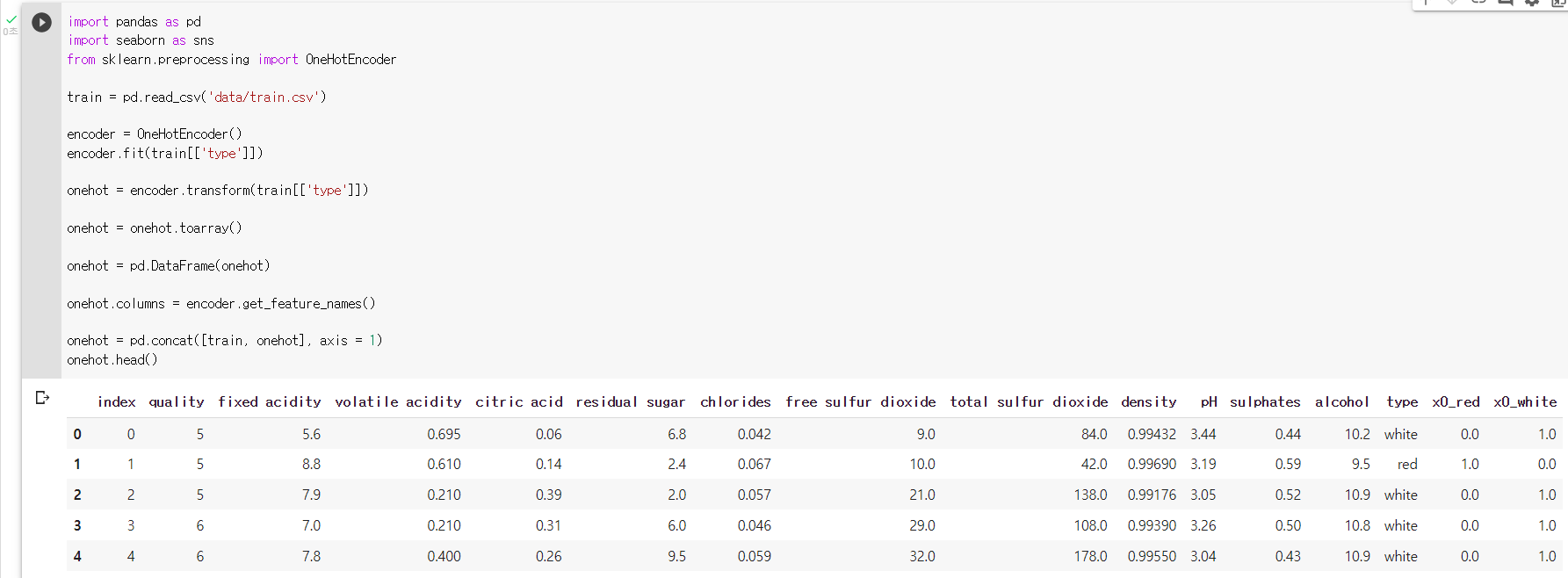
: onehot을 원본데이터인 train에 병합시켜보기onehot = pd.concat([train, onehot], axis = 1)
: train의 "type" 변수를 제거해주기train = train.drop(columns = ['type'])
: 코드를 더 단순화 시켜보기train = pd.read_csv('data/train.csv') encoder = OneHotEncoder() onehot = pd.DataFrame(encoder.fit_transform(train[['type']]).toarray(), columns = encoder.get_feature_names()) train = pd.concat([train,onehot], axis = 1).drop(columns = ['type']) train.head()
# 모델링
1. 모델 정의 RandomForestClassifier()
: “와인 품질 분류”는 말 그대로, 와인의 품질이 어느정도일지를 예측하는 문제이기 때문에, “분류 모델”을 사용해서 예측해본다.
: 분류 모형은 어떤 그룹에 속할지를 예측하는 모형이다.
: 랜덤포레스트 분류 모형을 불러오기
from sklearn.ensemble import RandomForestClassifier: 랜덤포레스트 분류 모형을 "random_forest"라는 변수에 저장하기
random_forest = RandomForestClassifier()
2. 모델 실습 RandomForestClassifier()
: 랜덤포레스트 분류 모형을 "random_classifier"라는 변수에 저장하기
random_classifier = RandomForestClassifier(): "X"라는 변수에 train의 "quality" 피쳐를 제거하고 저장하기
X = train.drop(columns = ['quality']): "y"라는 변수에 정답인 train의 "quality" 피쳐를 저장하기
y = train['quality']: "random_classifier"를 X와 y를 이용해 학습시켜보기
random_classifier.fit(X,y)
3. 교차 검증 정의 K-Fold
Hold-out
: 단순하게 Train 데이터를 (train, valid)라는 이름의 2개의 데이터로 나누는 작업이다.
: 예측 성능을 가늠해보기 위해 보통 train : valid = 8:2 혹은 7:3의 비율로 데이터를 나눈다.
: Hold-out의 문제점은 데이터의 낭비이다.
: trian과 test로 분할하게 된다면, 20%의 데이터는 모델이 학습할 기회도 없이, 예측만하고 버려지게 된다.
:그래서 "모든 데이터를 학습하게 해보자!"라는 생각에서 나온 것이 "교차검증", 즉 K-Fold이다.
교차검증
: "모든 데이터를 최소한 한 번씩 다 학습하게 하자!"
: valid 데이터를 겹치지 않게 나누어 N개의 데이터셋을 만들어 낸다.
: sklearn에 model_selection 부분 속 KFold를 불러오기
from sklearn.model_selection import KFold: KFold에 n_splits = 5, shuffle = True, random_state = 0이라는 인자를 추가해 "kf"라는 변수에 저장해보기
kf = KFold(n_splits = 5, shuffle = True, random_state = 0): 반복문을 통해서 1번부터 5번까지의 데이터에 접근해보기
for train_idx, valid_idx in kf.split(train) : train_data = train.iloc[train_idx] valid_data = train.iloc[valid_idx]
4. 교차검증 실습 K-Fold
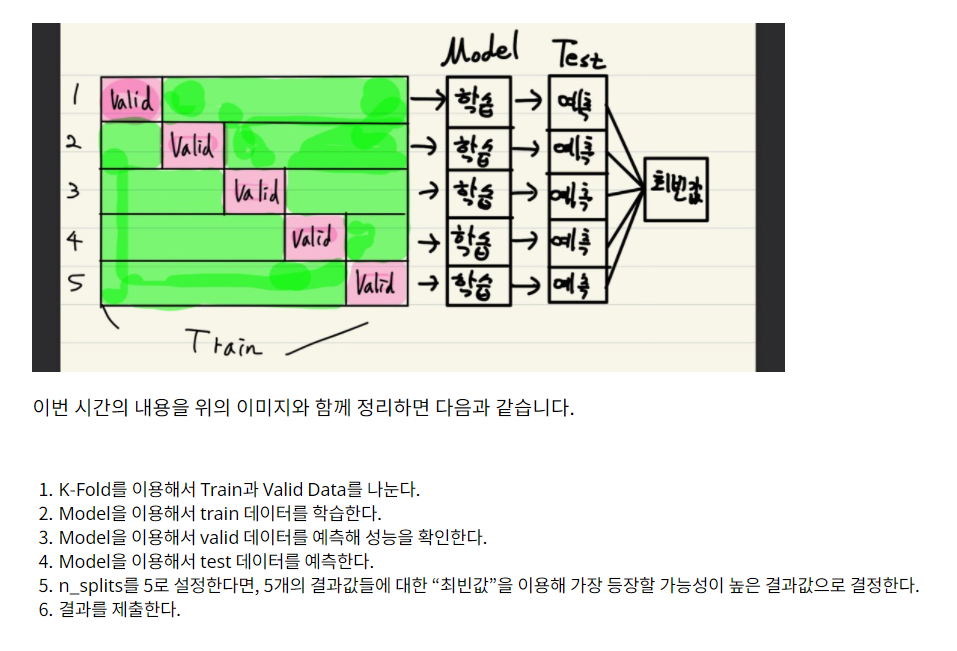
: "X"라는 변수에 train의 "index"와 "quality"를 제외하고 지정하기
X = train.drop(columns = ['index','quality']): "y"라는 변수에는 "quality"를 지정하기
y = train['quality']: "kf"라는 변수에 KFold를 지정하기
: n_splits는 5, shuffle은 True, random_state는 0으로 설정하기kf = KFold(n_splits = 5, shuffle = True, random_state = 0): "model"이라는 변수에 RandomForestClassifier를 지정하기
model = RandomForestClassifier(random_state = 0): valid_scores라는 빈 리스트를 하나 만들기
valid_scores = []: test_predictions라는 빈 리스트를 하나 만들기
test_predictions = []: 지난 시간에 다루었던 kf.split()을 활용해, 반복문으로 X_tr, y_tr, X_val, y_val을 설정하기
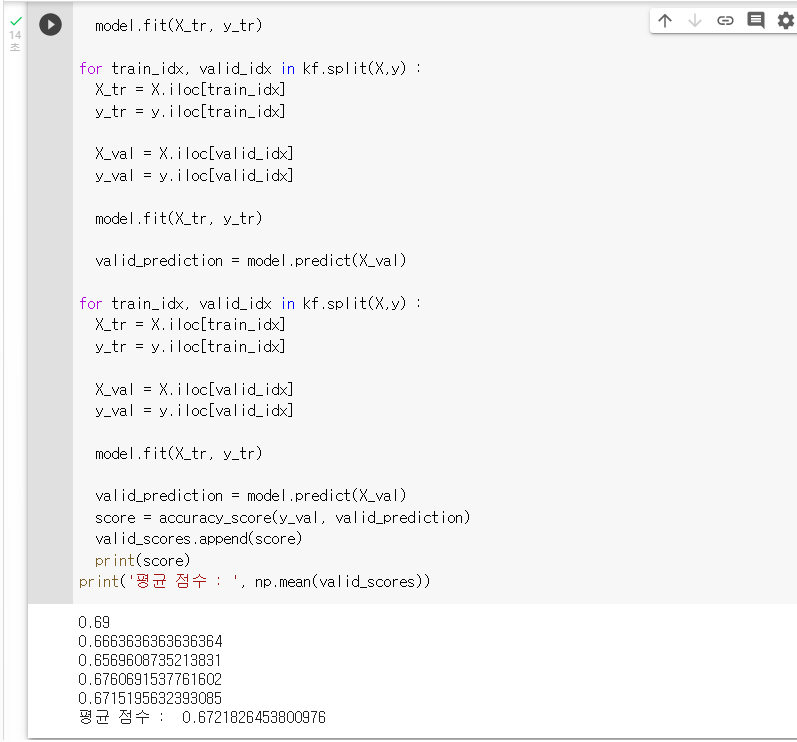
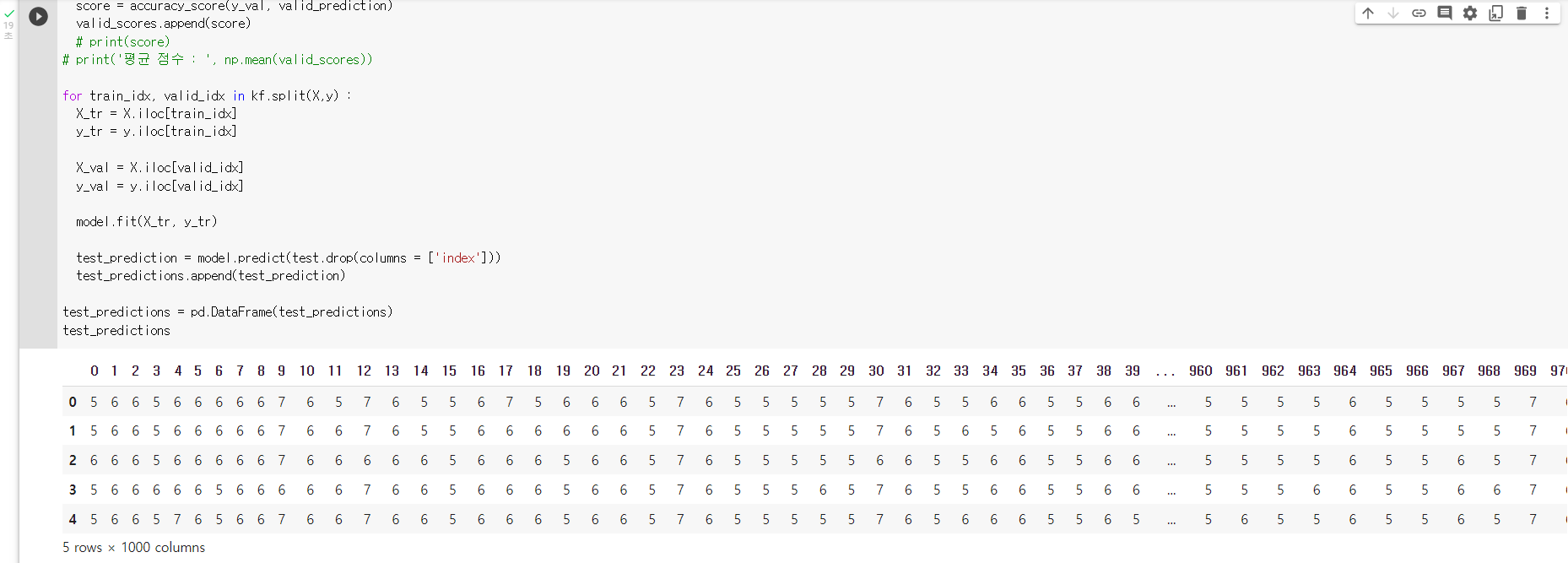
for train_idx, valid_idx in kf.split(X,y) : X_tr = X.iloc[train_idx] y_tr = y.iloc[train_idx] X_val = X.iloc[valid_idx] y_val = y.iloc[valid_idx]: 앞의 문제에 이어서 반복문 속에서 model.fit(X_tr, y_tr)을 활용해 모델을 학습하기
for train_idx, valid_idx in kf.split(X,y) : X_tr = X.iloc[train_idx] y_tr = y.iloc[train_idx] X_val = X.iloc[valid_idx] y_val = y.iloc[valid_idx] model.fit(X_tr, y_tr): 앞의 문제에 이어서 반복문 속에서 "valid_prediction"이라는 변수에 model.predict(X_val)의 결과를 저장하기
for train_idx, valid_idx in kf.split(X,y) : X_tr = X.iloc[train_idx] y_tr = y.iloc[train_idx] X_val = X.iloc[valid_idx] y_val = y.iloc[valid_idx] model.fit(X_tr, y_tr) valid_prediction = model.predict(X_val): 앞의 문제에 이어서 반복문 속에서 accuracy_score를 이용해, 모델이 어느정도의 예측 성능이 나올지 확인하기
: "valid_prediction"의 점수를 scores에 저장하기
: 반복문에서 빠져나온 후에 np.mean()을 활용해 평균 점수를 예측하기for train_idx, valid_idx in kf.split(X,y) : X_tr = X.iloc[train_idx] y_tr = y.iloc[train_idx] X_val = X.iloc[valid_idx] y_val = y.iloc[valid_idx] model.fit(X_tr, y_tr) valid_prediction = model.predict(X_val) score = accuracy_score(y_val, valid_prediction) valid_scores.append(score) print(score) print('평균 점수 : ', np.mean(valid_scores))
: 반복문 속에서 test를 예측해 "test_prediction"이라는 변수에 지정하기
: test_prediction을 지정했다면, "test_precitions"라는 빈 리스트에 넣어주기for train_idx, valid_idx in kf.split(X,y) : X_tr = X.iloc[train_idx] y_tr = y.iloc[train_idx] X_val = X.iloc[valid_idx] y_val = y.iloc[valid_idx] model.fit(X_tr, y_tr) test_prediction = model.predict(test.drop(columns = ['index'])) test_predictions.append(test_prediction): "test_precitions"를 Data Frame으로 만들기
test_predictions = pd.DataFrame(test_predictions)
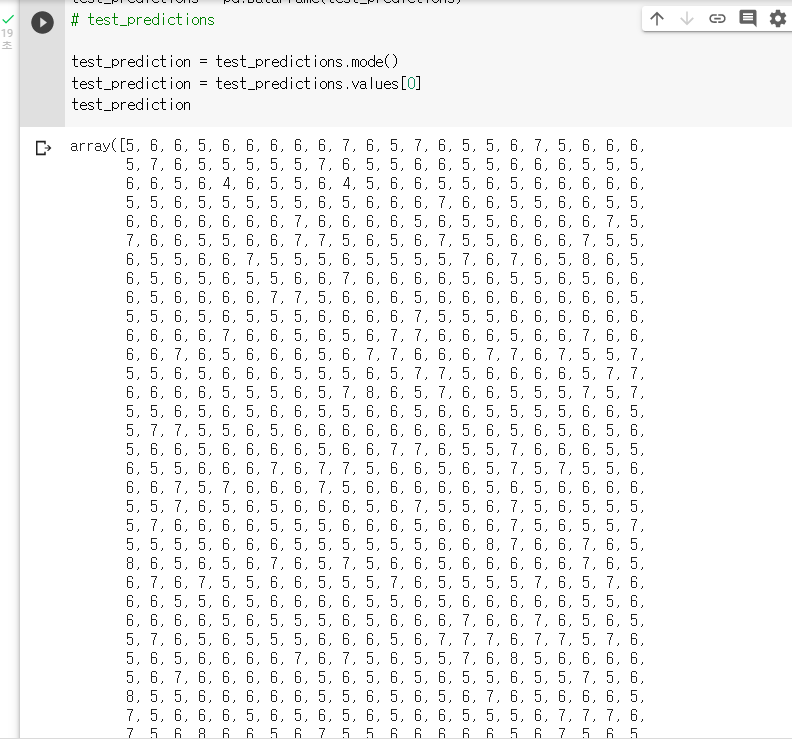
: DF.mode()를 활용해 열별 최빈값을 확인하고, "test_prediction"이라는 변수에 지정하기test_prediction = test_predictions.mode()
: "test_prediction"의 첫 행을 최종 결과값으로 사용하기test_prediction = test_predictions.values[0]
: data의 sample_submission 파일을 불러와 "quality"라는 변수에 "test_precition"을 저장하기sample_submission = pd.read_csv('data/sample_submission.csv') sample_submission['quality'] = test_prediction: "data/submission_KFOLD.csv"에 저장하고, 제출하기
sample_submission.to_csv('data/submission_KFOLD.csv', index=False)
# 튜닝
1. Bayesian Optimization
: Bayesian Optmization은 하이퍼 파라미터 튜닝과 관련된 내용이다.
: 하이퍼 파라미터 튜닝은 Grid Search, Random Search이다.
: 2가지에는 "최적의 값을 찾아갈 수 없다"라는 공통적인 문제점이 있다.
: 이를 해결하기 위한 방법 중 하나가 "Bayesian Optimization"이다.
: bayesian-optimization을 설치하기
pip install bayesian-optimization: bayes_opt 패키지에서 BayesianOptimization을 불러오기
from bayes_opt import BayesianOptimization
2. 그리드, 랜덤 서치 vs Bayesian Optimization
Grid Search
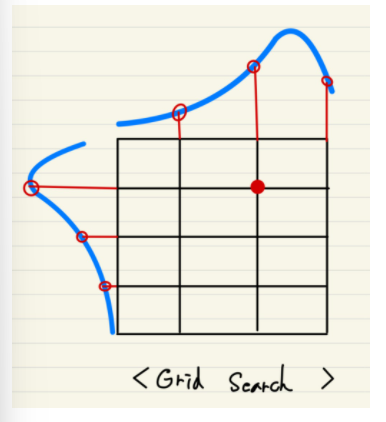
-
기법 : Grid Search는 사전에 탐색할 값들을 미리 지정해주고, 그 값들의 모든 조합을 바탕으로 성능의 최고점을 찾아냅니다.
-
장점
: 내가 원하는 범위를 정확하게 비교 분석이 가능하다. -
단점
: 시간이 오래걸린다.:
: 성능의 최고점이 아닐 가능성이 높다.
: "최적화 검색" (여러개들을 비교 분석해서 최고를 찾아내는 기법)이지, "최적화 탐색"(성능이 가장 높은 점으로 점차 찾아가는 기법)이 아니다.
Random Search
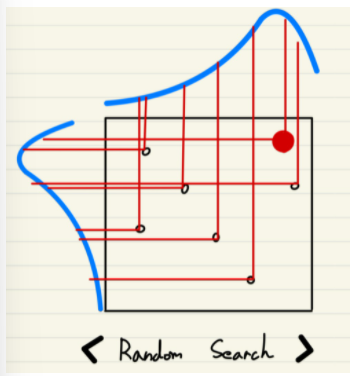
-
기법 : 사전에 탐색할 값들의 범위를 지정해주고, 그 범위 속에서 가능한 조합을 바탕으로 최고점을 찾아냅니다.
-
장점
: Grid Search에 비해 시간이 짧게 걸린다.
: Grid Search보다, 랜덤하게 점들을 찍으니, 성능이 더 좋은 점으로 갈 가능성이 높다. -
단점
: 반대로 성능이 Grid Search보다 낮을 수 있다.
: 하이퍼 파라미터의 범위가 너무 넓으면, 일반화된 결과가 나오지 않는다. (할 때 마다 달라진다)
: seed를 고정하지 않으면, 할 때 마다 결과가 달라진다.
: 마찬가지로, "최적값 검색"의 느낌이지, "최적화 탐색"의 개념이 아니다.
Bayeisan Optimization
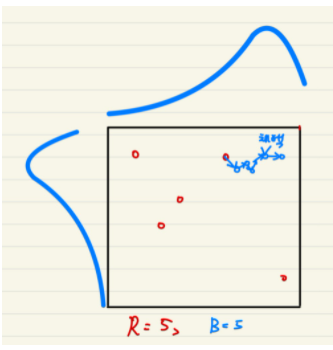
-
기법 : 하이퍼파라미터의 범위를 지정한 후, Random하게 R 번 탐색한 후, B번 만큼 최적의 값을 찾아간다.
-
장점
: 정말 "최적의 값"을 찾아갈 수 있다.
: 상대적으로 시간이 덜 걸린다.
: 엔지니어가 그 결과값을 신뢰할 수 있다. -
단점
: Random하게 찍은 값이 달라질 경우, 최적화 하는데 오래 걸릴 수 있다.
: Random하게 찍은 값이 부족하면, 최적의 값을 탐색하는게 불가능 할 수 있다.
: Rnadom하게 찍은 값이 너무 많으면, 최적화 이전에 이미 최적값을 가지고 있을 수도 있다.
: 그럼에도, Bayesian Optimization은 수동적으로 하이퍼 파라미터를 튜닝하는데 좋은 결과를 가져온다.
3. Bayesian Optimization 실습
: X에 학습할 데이터를, y에 목표 변수를 저장하기
X = train.drop(columns = ['index', 'quality']) y = train['quality']: 랜덤포레스트의 하이퍼 파라미터의 범위를 dictionary 형태로 지정하기
: Key는 랜덤포레스트의 hyperparameter이름이고, value는 탐색할 범위이다.rf_parameter_bounds = { 'max_depth' : (1,3), # 나무의 깊이 'n_estimators' : (30,100), }: 함수 만들기
1) 함수에 들어가는 인자 = 위에서 만든 함수의 key값들
2) 함수 속 인자를 통해 받아와 새롭게 하이퍼파라미터 딕셔너리 생성
3) 그 딕셔너리를 바탕으로 모델 생성
4) train_test_split을 통해 데이터 train-valid 나누기
5) 모델 학습
6) 모델 성능 측정
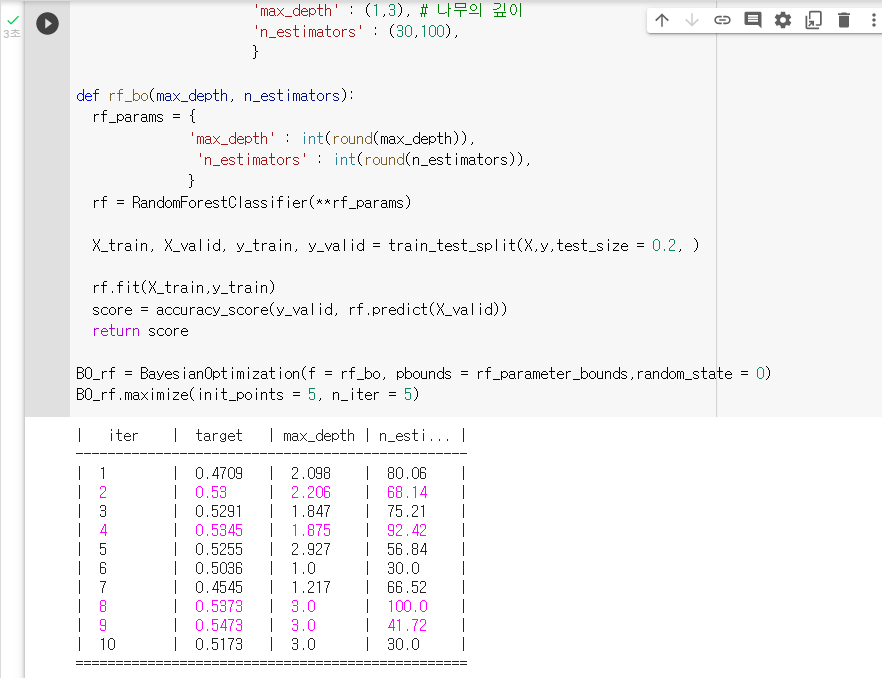
7) 모델의 점수 반환def rf_bo(max_depth, n_estimators): rf_params = { 'max_depth' : int(round(max_depth)), 'n_estimators' : int(round(n_estimators)), } rf = RandomForestClassifier(**rf_params) X_train, X_valid, y_train, y_valid = train_test_split(X,y,test_size = 0.2, ) rf.fit(X_train,y_train) score = accuracy_score(y_valid, rf.predict(X_valid)) return score: "BO_rf"라는 변수에 Bayesian Optmization을 저장하기
BO_rf = BayesianOptimization(f = rf_bo, pbounds = rf_parameter_bounds,random_state = 0): Bayesian Optimization 실행하기
BO_rf.maximize(init_points = 5, n_iter = 5)
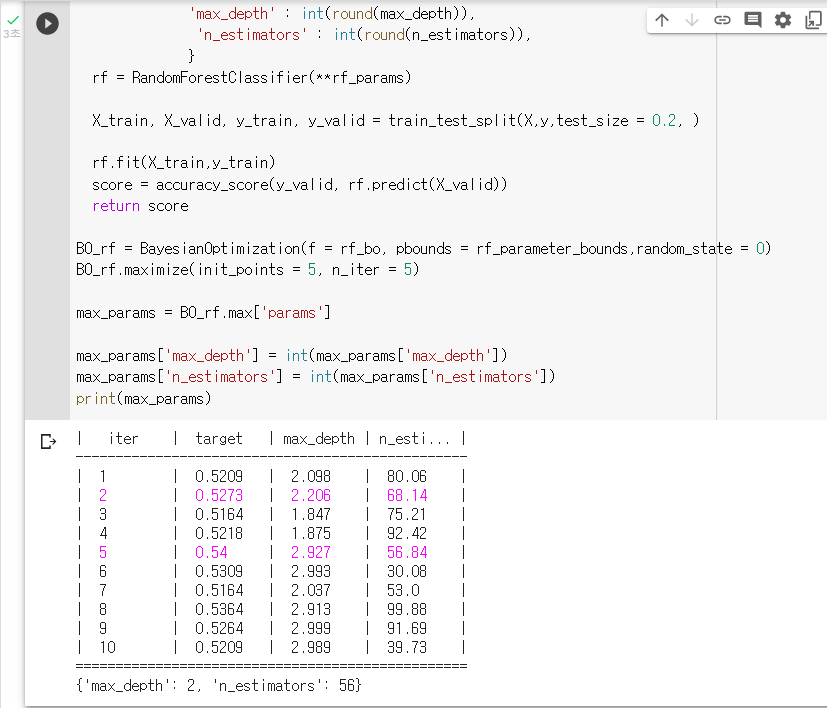
: 하이퍼파라미터의 결과값을 불러와 "max_params"라는 변수에 저장하기max_params = BO_rf.max['params'] max_params['max_depth'] = int(max_params['max_depth']) max_params['n_estimators'] = int(max_params['n_estimators']) print(max_params)
: Bayesian Optimization의 결과를 "BO_tuend_rf"라는 변수에 저장하기BO_tuend_rf = RandomForestClassifier(**max_params)