
0. INTRO
- 머신러닝 모델 개발시 하이퍼파라미터를 최적화해주는 툴들이 몇 가지가 있다. 일반적으로
sklearn라이브러리에 내장된GridSearch나RandomSearch등을 사용하게 된다. - 하지만 여기엔 몇 가지 한계점이 존재한다. 주어진 하이퍼파라미터 조합에서 가장 최적화된 조합을 찾아내주지만 하이퍼파라미터 조합을 결국에는 사용자가 던져줘야 한다는 것이다.
- 이런 경우에 조합을 나름 다양하게 주더라도 최적화 값이 그 조합의 범위에 있지 않을 수도 있고, 조합의 범위에 있다고 하더라도 5나 10의 배수 간격으로 주는 것이 일반적이기에 해당 값이 정말로 최적화된 조합의 값이라고 확신할 수 없다.(조합을 [1,5,10,20] 이렇게 주었는데 16이 최적일 수 있으므로)
Optuna는 하이퍼파라미터 최적화를 보다 효율적으로 수행할 수 있게 도와주는 도구이다. 물론 탐색 범위는 사람이 지정해주어야 하지만 일반적인 서치보다 훨씬 더 촘촘하게 파라미터들을 탐색하여 최적의 조합을 알려준다. 거기에 더해 내장된 메소드를 통해 간단한 시각화도 구현이 가능하다.- 이번 글에서는
Optuna를 활용하여 하이퍼파라미터를 최적화하는 과정을 회귀 모델과 분류 모델에 각각 적용해 볼 것이다.
1. 설치
- pip install 명령을 통해 간단하게 설치가 가능하다.
pip install optuna
pip install plotly==5.24.1 # 시각화를 위해 필요
pip install --upgrade nbformat # 의존성 해결을 위해 필요2. 회귀 모델에 적용
1) 데이터 불러오기
- 간단하게 sklearn에 내장되어 있는 당뇨병 데이터셋(load_diabetes)을 불러와 사용해볼 것이다.
from sklearn.datasets import load_diabetes
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split, cross_val_score
from sklearn.svm import SVR
from sklearn.ensemble import RandomForestRegressor
dataset = load_diabetes()
X = dataset.data
y = dataset.target2) 스케일링 및 split
- sklearn에서 제공되는 데이터셋은 기본적으로 형태가 깔끔해 굳이 스케일링이 필요 없지만 형식상 추가하여 train_test_split 진행
sc = StandardScaler()
X = sc.fit_transform(X)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.25, random_state=42)
X_train.shape, X_test.shape, y_train.shape, y_test.shape
---
((331, 10), (111, 10), (331,), (111,))3) objective 함수 생성
- optuna의 핵심이라고 볼 수 있는
objective함수를 생성한다. - 이 함수는
trial이라는 optuna의 객체를 받아와 여러 하이퍼파라미터 조합에 대해 모델 학습을 진행하고 각 조합마다의 score를 return 해주는 함수이다. - 회귀 모델로는
SVR과RandomForestRegressor이렇게 두 가지 모델을 사용할 것이며 대략적이 형태는 아래와 같다.
def objective(trial):
## 1. 모델 선택
regressor_name = trial.suggest_categorical("regressor", ["SVR", "RandomForest"])
## 2. SVR 하이퍼파라미터 조합 관련 내용
if regressor_name == "SVR":
## SVR의 인자들로 들어가는 파라미터들을 입력하며, 각 파라미터들에는 조합이 될 값의 범위를 입력해준다.
kernel = trial.suggest_categorical("kernel", ['linear', 'poly', 'rbf'])
gamma = trial.suggest_categorical("gamma", ['scale', 'auto'])
c_value = trial.suggest_float("C", 0.001, 10, log=True)
model = SVR(
kernel=kernel,
gamma=gamma,
C=c_value
)
## 3. RandomForest 하이퍼파라미터 조합 관련 내용
elif regressor_name == "RandomForest":
# Random Forest 하이퍼파라미터 탐색
n_estimators = trial.suggest_int('n_estimators', 10, 300)
max_depth = trial.suggest_int('max_depth', 3, 30)
min_samples_split = trial.suggest_int('min_samples_split', 2, 32)
min_samples_leaf = trial.suggest_int('min_samples_leaf', 1, 32)
model = RandomForestRegressor(
n_estimators=n_estimators,
max_depth=max_depth,
min_samples_split=min_samples_split,
min_samples_leaf=min_samples_leaf
)
## 4. 교차 검증을 통한 성능 평가
score = cross_val_score(model, X_train, y_train, cv=5, scoring="r2")
accuracy = score.mean()
## 5. 평균 정확도 return
return accuracy-
suggest 관련 메소드들 설명
-
suggest_categorical(name, choices): 범주형 하이퍼파라미터를 선택하는 함수로 주어진 목록에서 값을 선택 -
suggest_discrete_uniform(name, low, high, q): 주어진 범위에서 간격(q) 단위로 이산적인 실수 값을 샘플링하는 함수 -
suggest_float(name, low, high, step=None, log=False): 연속적인 실수 값을 주어진 범위 내에서 선택하는 함수.step으로 이산적인 샘플링도 가능하며,log=True옵션으로 로그 스케일로도 샘플링. -
suggest_int(name, low, high, step=1, log=False): 주어진 정수 범위 내에서 값을 선택하는 함수.step으로 간격을 설정할 수 있고,log=True로 로그 스케일 사용 가능 -
suggest_loguniform(name, low, high): 주어진 범위에서 값이 로그 스케일로 균등하게 샘플링되는 실수 값을 선택 -
suggest_uniform(name, low, high): 주어진 범위에서 값을 균등하게 샘플링하는 함수
-
4) 최적화 실행
- 여기서
study라는 개념이 나오는데, 이는 하나의 최적화 실험(작업) 단위 라고 이해하면 된다. study를 통해 Optuna는 여러 번의 Trial(개별 하이퍼파라미터 조합을 평가하는 단위)을 생성하고 관리하며, 각 Trial의 결과를 기록하여 최적의 결과를 추적하게 된다.
# Optuna Study 생성 및 실행
study = optuna.create_study(
sampler=optuna.samplers.TPESampler(),
pruner=optuna.pruners.MedianPruner(),
direction="maximize", # 원하는 값을 최대화하는게 목표인지, 최소화하는게 목표인지 설정
)
# trial과 timeout 둘 중 빨리 끝나는대로 최적화 종료
study.optimize(
objective,
n_trials=50,
timeout=600
) - 실행 결과
[I 2024-10-31 15:01:26,886] A new study created in memory with name: no-name-40c2637c-1edc-42e8-b37f-557c69d04de2
[I 2024-10-31 15:01:28,796] Trial 0 finished with value: 0.4209059088132555 and parameters: {'regressor': 'RandomForest', 'n_estimators': 280, 'max_depth': 6, 'min_samples_split': 24, 'min_samples_leaf': 7}. Best is trial 0 with value: 0.4209059088132555.
[I 2024-10-31 15:01:28,817] Trial 1 finished with value: 0.09212503764103638 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'auto', 'C': 0.025654523291531037}. Best is trial 0 with value: 0.4209059088132555.
[I 2024-10-31 15:01:30,150] Trial 2 finished with value: 0.41467996937815615 and parameters: {'regressor': 'RandomForest', 'n_estimators': 209, 'max_depth': 4, 'min_samples_split': 26, 'min_samples_leaf': 11}. Best is trial 0 with value: 0.4209059088132555.
[I 2024-10-31 15:01:30,676] Trial 3 finished with value: 0.41786354813302695 and parameters: {'regressor': 'RandomForest', 'n_estimators': 75, 'max_depth': 17, 'min_samples_split': 20, 'min_samples_leaf': 5}. Best is trial 0 with value: 0.4209059088132555.
[I 2024-10-31 15:01:30,706] Trial 4 finished with value: -0.029951220808408775 and parameters: {'regressor': 'SVR', 'kernel': 'rbf', 'gamma': 'auto', 'C': 0.11561206038739895}. Best is trial 0 with value: 0.4209059088132555.
[I 2024-10-31 15:01:30,727] Trial 5 finished with value: 0.4224911654714198 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'scale', 'C': 0.2796309220141383}. Best is trial 5 with value: 0.4224911654714198.
[I 2024-10-31 15:01:30,749] Trial 6 finished with value: -0.04838451469933349 and parameters: {'regressor': 'SVR', 'kernel': 'poly', 'gamma': 'scale', 'C': 0.0017449030715916737}. Best is trial 5 with value: 0.4224911654714198.
[I 2024-10-31 15:01:32,362] Trial 7 finished with value: 0.40159995815416727 and parameters: {'regressor': 'RandomForest', 'n_estimators': 274, 'max_depth': 24, 'min_samples_split': 19, 'min_samples_leaf': 25}. Best is trial 5 with value: 0.4224911654714198.
[I 2024-10-31 15:01:33,253] Trial 8 finished with value: 0.4115063199608911 and parameters: {'regressor': 'RandomForest', 'n_estimators': 142, 'max_depth': 9, 'min_samples_split': 25, 'min_samples_leaf': 15}. Best is trial 5 with value: 0.4224911654714198.
[I 2024-10-31 15:01:33,282] Trial 9 finished with value: 0.44997612414257926 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'auto', 'C': 3.56127719702921}. Best is trial 9 with value: 0.44997612414257926.
[I 2024-10-31 15:01:33,315] Trial 10 finished with value: 0.4452863395555141 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'auto', 'C': 7.439831438974003}. Best is trial 9 with value: 0.44997612414257926.
[I 2024-10-31 15:01:33,348] Trial 11 finished with value: 0.4456593561741492 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'auto', 'C': 9.558948644002822}. Best is trial 9 with value: 0.44997612414257926.
...
[I 2024-10-31 15:01:36,756] Trial 46 finished with value: 0.44573707833940046 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'scale', 'C': 5.5370103208716985}. Best is trial 16 with value: 0.4622758412775975.
[I 2024-10-31 15:01:37,405] Trial 47 finished with value: 0.4087078254954302 and parameters: {'regressor': 'RandomForest', 'n_estimators': 104, 'max_depth': 11, 'min_samples_split': 12, 'min_samples_leaf': 19}. Best is trial 16 with value: 0.4622758412775975.
[I 2024-10-31 15:01:37,433] Trial 48 finished with value: 0.21973295372619198 and parameters: {'regressor': 'SVR', 'kernel': 'poly', 'gamma': 'scale', 'C': 1.8065925039335826}. Best is trial 16 with value: 0.4622758412775975.
[I 2024-10-31 15:01:37,459] Trial 49 finished with value: 0.4561550501709391 and parameters: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'scale', 'C': 0.9157797590482355}. Best is trial 16 with value: 0.4622758412775975.5) 최적의 하이퍼파라미터 출력
- 사람이 수동으로 줬을 때 나오는 일반적인 정수 조합에 비해 더 최적화된 실수값으로 값이 출력되는 것을 확인할 수 있다.
## 최적의 조합 저장 객체
trial = study.best_trial
print("Value:", trial.value)
print("Params:", trial.params)
---
Value: 0.4622758412775975
Params: {'regressor': 'SVR', 'kernel': 'linear', 'gamma': 'scale', 'C': 1.5994213635883505}6) 최적의 모델 생성 및 학습
best_model_params = study.best_trial.params
## 필요 없는 key 제거
best_model_params.pop("regressor")
## 하이퍼파라미터 조합을 모델에 대입
best_model = SVR(**best_model_params)
## 학습 및 score 출력
best_model.fit(X_train, y_train)
print(best_model.score(X_train, y_train))
print(best_model.score(X_test, y_test))3. 분류 모델에 적용
sklearn의load_wine데이터셋을 불러와DecisionTree,RandomForest,XGBoost세 가지 모델로 학습을 진행하며, 그 과정에서 하이퍼파라미터 튜닝을 통해 최적의 모델을 찾아볼 것이다.- 위에 회귀모델의 코드와 구성은 거의 비슷하기 때문에 전체 코드에 주석을 다는 형식으로 자세한 설명을 대체 할 것이다.
import optuna
from sklearn.model_selection import train_test_split, cross_val_score
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from xgboost import XGBClassifier
from sklearn.datasets import load_wine
## 1. wine 데이터셋 로드 + split 진행
data = load_wine()
X, y = data.data, data.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.25, random_state=42)
## 2. objective 함수 구성
def objective(trial):
# 모델 선택
classifier_name = trial.suggest_categorical("classifier", ["DecisionTree", "RandomForest", "XGBoost"])
if classifier_name == "DecisionTree":
# Decision Tree 하이퍼파라미터 탐색
max_depth = trial.suggest_int("max_depth", 2, 10)
min_samples_split = trial.suggest_int("min_samples_split", 2, 20)
model = DecisionTreeClassifier(
max_depth=max_depth,
min_samples_split=min_samples_split
)
elif classifier_name == "RandomForest":
# Random Forest 하이퍼파라미터 탐색
n_estimators = trial.suggest_int("n_estimators", 50, 200)
max_depth = trial.suggest_int("max_depth", 2, 10)
min_samples_split = trial.suggest_int("min_samples_split", 2, 20)
model = RandomForestClassifier(
n_estimators=n_estimators,
max_depth=max_depth,
min_samples_split=min_samples_split
)
else:
# XGBoost 하이퍼파라미터 탐색
n_estimators = trial.suggest_int("n_estimators", 50, 200)
max_depth = trial.suggest_int("max_depth", 2, 10)
learning_rate = trial.suggest_float("learning_rate", 0.01, 0.3)
model = XGBClassifier(
n_estimators=n_estimators,
max_depth=max_depth,
learning_rate=learning_rate,
eval_metric="mlogloss"
)
# 교차 검증을 통한 성능 평가
score = cross_val_score(model, X_train, y_train, cv=5, scoring="accuracy")
accuracy = score.mean()
return accuracy
## 3. Optuna Study 생성 및 실행
study = optuna.create_study(
sampler=optuna.samplers.TPESampler(),
pruner=optuna.pruners.MedianPruner(),
direction="maximize",
)
study.optimize(
objective,
n_trials=50,
timeout=600
)
## 4. 최적의 하이퍼파라미터 출력
trial = study.best_trial
print("Value:", trial.value)
print("Params:", trial.params)
---
Value: 0.9774928774928775
Params: {
'classifier': 'RandomForest',
'max_depth': 5,
'min_samples_split': 12,
'n_estimators': 166
}4. 시각화
study를 통한 하이퍼파라미터 탐색 조합 과정에 대한 시각화를 간편하게 구현해볼 수 있다.- 단, 여러 모델이 포함된 objective 함수로 작업시에는 한계가 있고, 하나의 모델을 대상으로 했을 때 의미가 있다.
- 아래는 RandomForestRegressor 하나의 모델만 사용하여 하이퍼파라미터 최적화 작업을 한 후 시각화까지 구현한 코드이다.
1) 하나의 분류 모델이 포함된 최적화 코드
import optuna
import pandas as pd
import seaborn as sns
from sklearn.model_selection import train_test_split, cross_val_score
from sklearn.ensemble import RandomForestRegressor
## 데이터셋 load
health = sns.load_dataset('healthexp')
health = pd.get_dummies(health).replace({True:1, False:0})
## 데이터셋 split
X = health.drop(['Life_Expectancy'], axis=1)
y = health['Life_Expectancy']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.25, random_state=20)
## optuna 최적화 적용
def objective(trial):
n_estimators = trial.suggest_int('n_estimators', 10, 300)
max_depth = trial.suggest_int('max_depth', 3, 10)
min_samples_split = trial.suggest_int('min_samples_split', 2, 32)
min_samples_leaf = trial.suggest_int('min_samples_leaf', 1, 32)
model = RandomForestRegressor(
n_estimators=n_estimators,
max_depth=max_depth,
min_samples_split=min_samples_split,
min_samples_leaf=min_samples_leaf
)
score = cross_val_score(model, X, y, n_jobs=-1, cv=5, scoring='neg_mean_squared_error').mean()
return score
study = optuna.create_study(
sampler=optuna.samplers.TPESampler(),
pruner=optuna.pruners.MedianPruner(),
direction="maximize",
)
study.optimize(
objective,
n_trials=50,
timeout=600
) 2) study에 대한 시각화 생성
from optuna.visualization import (
plot_optimization_history,
plot_param_importances,
plot_parallel_coordinate,
plot_rank,
plot_slice
)plot_optimization_history: 최적화 과정에서 각 Trial의 성능 변화를 보여주는 시각화로, 성능 향상 추이를 파악할 수 있습니다.plot_optimization_history(study)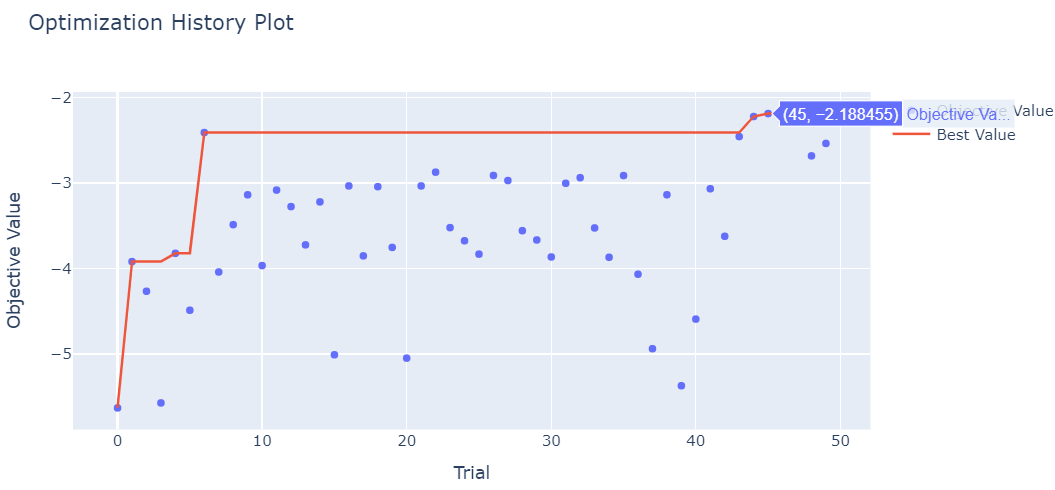
plot_param_importances: 각 하이퍼파라미터가 최적화 결과에 미치는 중요도를 표시해주는 시각화입니다.plot_param_importances(study)
plot_parallel_coordinate: 여러 하이퍼파라미터와 그 값에 따른 성능 변화를 시각적으로 비교하는 평행좌표 그래프를 제공합니다.plot_parallel_coordinate(study)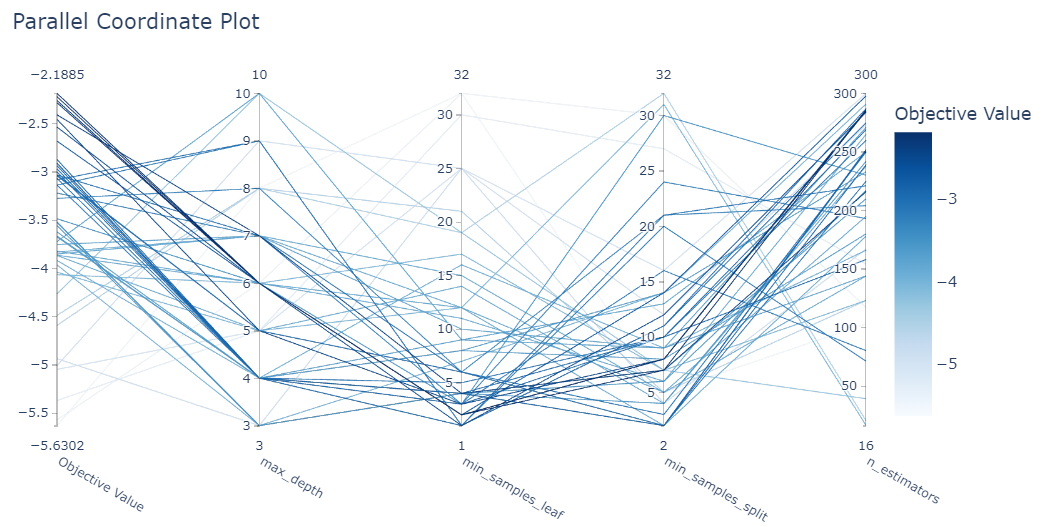
plot_rank: 각 Trial의 순위를 시각화하여 최적화된 결과들의 순위 분포를 보여줍니다.plot_rank(study)
plot_slice: 특정 하이퍼파라미터 값에 따른 성능 변화를 나타내어, 파라미터와 목표 값 간의 관계를 이해할 수 있도록 합니다.plot_slice(study)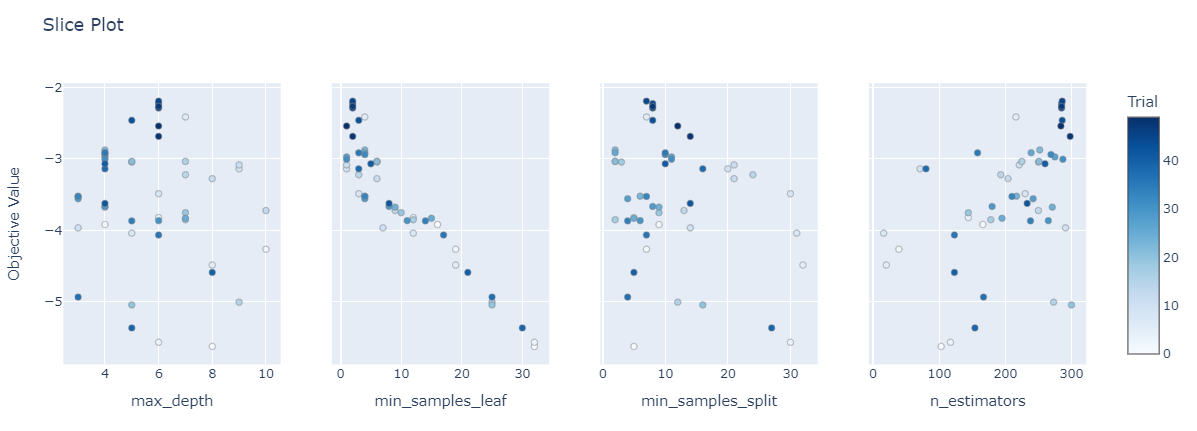
5. OUTRO
- Optuna는 기존의 하이퍼파라미터 최적화 방식인 Grid Search나 Random Search보다 훨씬 세밀하고 효율적인 최적화를 제공한다. Grid Search는 정해진 값으로 일일이 조합을 시도하기 때문에 시간이 많이 소요되고, Random Search는 시간 절약 측면에서는 장점이 있지만 여전히 최적 조합을 놓칠 가능성이 크다.
- 반면에, Optuna는 베이지안 최적화와 같은 고도화된 샘플링 방식을 통해 최적의 하이퍼파라미터를 더욱 정밀하게 탐색할 수 있어, 특히 파라미터의 탐색 범위가 크거나 조합이 다양할 때 효과적으로 활용이 가능하다.
objective함수를 만들고,trial객체를 전달하여study를 생성하는 등 처음에는 직관적으로 와닿지 않는 부분들이 있지만 익숙해지고 어느정도 템플릿화 시켜서 활용한다면 상당히 유용한 최적화 도구가 될 것 같다.
