
segmenatation 수행 시 모델의 아웃풋으로 나온 numpy array를 원본 이미지와 겹쳐보기 위해
나온 numpy array를 다시 nifti 이미지로 저장하는 코드입니다.
예시로 마스크가 있는 CT 이미지를 가져와 마스크를 numpy로 바꾸고 다시 nifti로 변환하고 원본과 비교해보겠습니다.
데이터 가져오기
dataset은 kaggle에 있는 "COVID-19 Lung CT Scan Segmentation" 활용
https://www.kaggle.com/datasets/maedemaftouni/large-covid19-ct-slice-dataset
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import cv2
import tensorflow as tf
import nibabel as nib
data_path='/content/gdrive/MyDrive/Colab Notebooks/data/'
data=pd.read_csv(data_path+'metadata.csv')
data.head()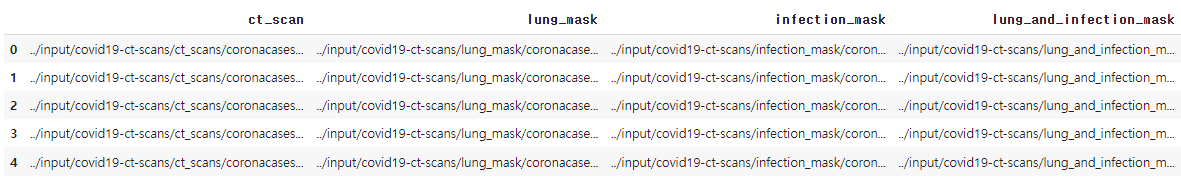
Nifti 읽기
def read_nii(data_path):
ct_scan= nib.load(data_path)
arr=ct_scan.get_fdata()
return arr
sample_ct = read_nii(data_path+data.loc[1,data.columns[0]][26:]) #ct image
sample_lung = read_nii(data_path+data.loc[1,data.columns[1]][26:]) #ct mask
#원본 ct 이미지와 mask 이미지 겹쳐 그리기
plt.imshow(sample_ct[...,150],cmap='bone')
plt.imshow(sample_lung[...,150],cmap='nipy_spectral',alpha=0.5)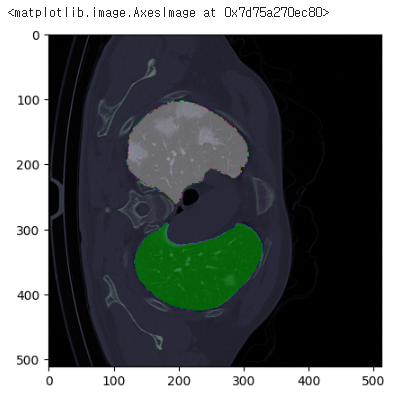
#마스크 이미지
plt.imshow(sample_lung[...,150],cmap='bone')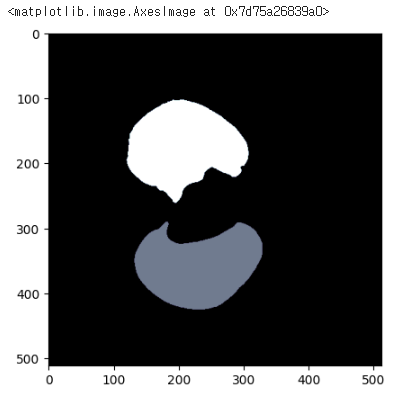
Mask 이미지 임의로 변경하기
data_dict={'img':data_path+data.loc[1,data.columns[1]][26:]}
load=mt.LoadImaged(keys=['img'],image_only=False)
channelfirst=mt.EnsureChannelFirstd(keys=['img'])
resized=mt.Resized(keys=['img'],spatial_size=(128,128,128),mode='trilinear')
data_dict=load(data_dict)
data_dict=channelfirst(data_dict)
data_dict=resized(data_dict)
img=data_dict['img']
plt.imshow(img[0][:,:,50],cmap='bone')
print(img.shape,type(img))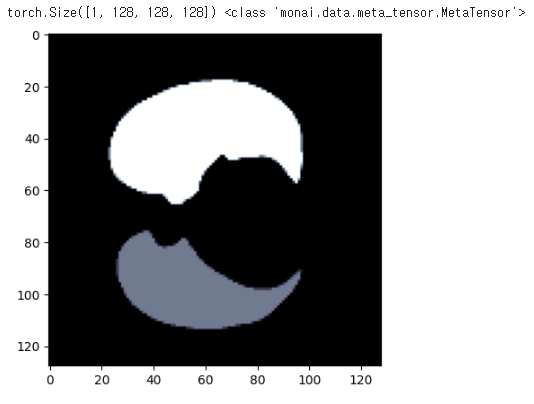
변경 사항 : torch로 변경, shape (1,128,128,128)로 변경
Mask 이미지 다시 Nifti로 복원하기
기존 형태와 맞춰주기 위해 ct 이미지 정보 확인
origin_ct = nib.load(data_path+data.loc[1,data.columns[0]][26:])
print(origin_ct.shape)
zoom을 활용하여 shape을 맞춰줌
기존 ct 이미지와의 origin 맞춰주기 위해 affine 확인
from scipy.ndimage import zoom
origin_shape = (origin_ct.shape[0],origin_ct.shape[1],origin_ct.shape[2])
origin_affine = origin_ct.affine
resized_data=zoom(img,zoom=(1,origin_shape[0]/img.shape[2],origin_shape[1]/img.shape[1],origin_shape[2]/img.shape[2]))
resized_data.shape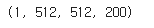
nifti 이미지로 복원
nifti_img=nib.Nifti1Image(resized_data[0],affine=origin_affine,header=origin_ct.header)
plt.imshow(nifti_img.get_fdata()[:,:,150])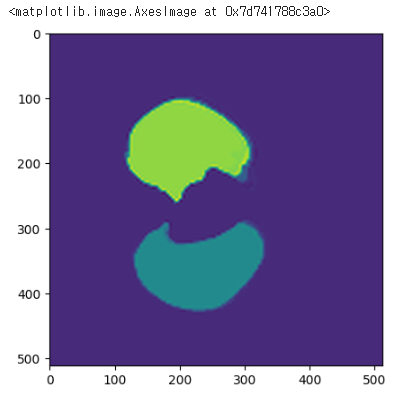
원본 이미지와 비교
fig,ax=plt.subplots(1,2)
ax[0].set_title('resample nifti')
ax[0].imshow(sample_ct[...,150],cmap='bone')
ax[0].imshow(nifti_img.get_fdata()[:,:,150],cmap='nipy_spectral',alpha=0.5)
ax[1].set_title('original nifti')
ax[1].imshow(sample_ct[...,150],cmap='bone')
ax[1].imshow(sample_lung[...,150],cmap='nipy_spectral',alpha=0.5)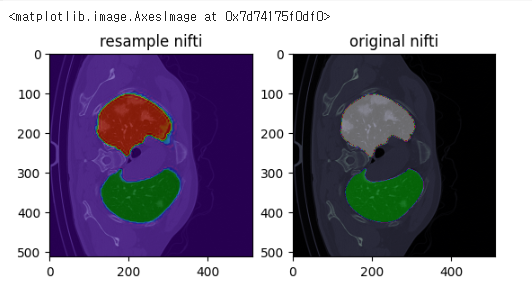
완전히 똑같아지진 않네요
그래도 다시 nifti로 복원했을 때 원본 이미지와 큰 차이는 없어 보입니다.
