
🧩 High Dimensional Data
📌 Eigenvalue, Eigenvector
Eigenvalue와 Eigenvector를 사람에 투영하여 설명한다면
- Eigenvalue = 외형(살이 찌거나 빠지거나, 피부가 그을리는 등)
- Eigenvector = 그사람 그 자체(다이어트를 한다고 해서 사람 자체가 바뀌지는 않음)
그래서 PCA는 Eigenvector를 축으로 하며,
Eigenvalue가 가장 커지는 쪽을 정하여 실행한다. (데이터의 특성을 가장 잘 반영하므로)
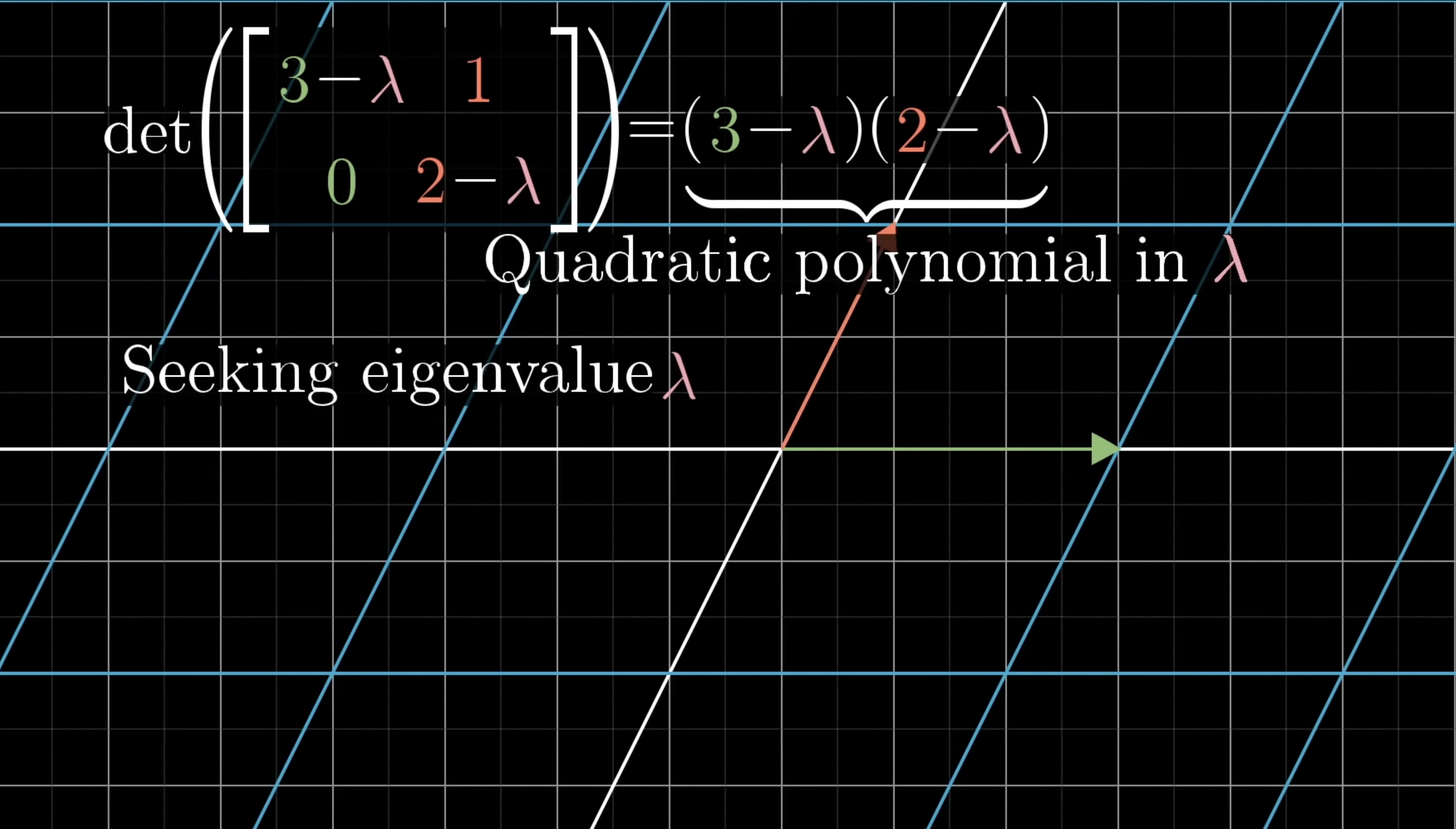
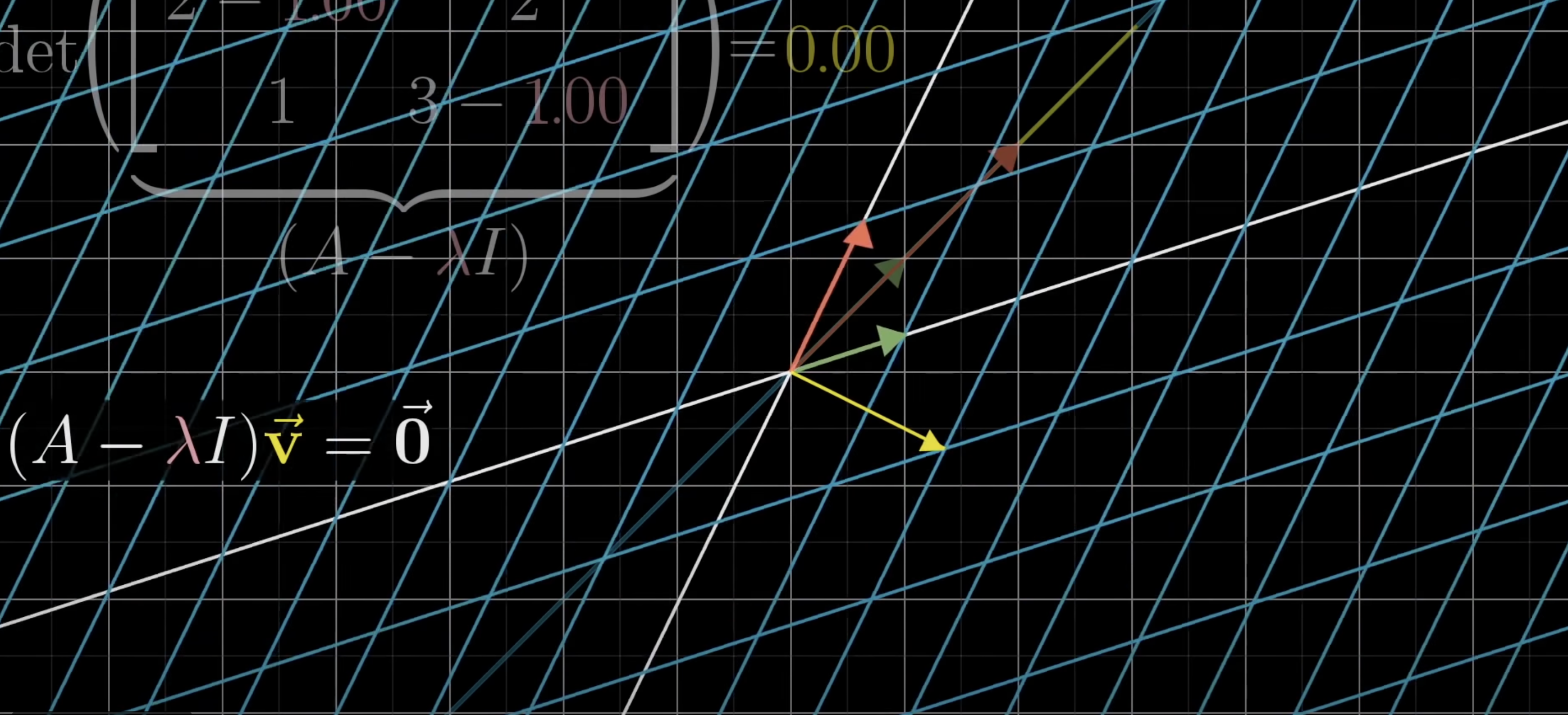 Eigenvector를 축으로 해서 돌아가는 그리드를 생각해보면 된다. 그리드가 우리 입장에서는 선으로 보일 수도, 면으로 보일 수도 있다.
Eigenvector를 축으로 해서 돌아가는 그리드를 생각해보면 된다. 그리드가 우리 입장에서는 선으로 보일 수도, 면으로 보일 수도 있다.
🏁 내 코드
import numpy as np
v = np.array([[4,2],[2,4]])
value = np.linalg.eigvals(v)
vector = np.linalg.eig(v)
print('eigenvalue=',value)
print('eigenvector=',vector)✔️np.linalg.eigvals()
numpy를 이용해 eigenvalue 구함
✔️ np.linalg.eig()
numpy를 이용해 eigenvector 구함
📌 PCA(Principal Component Analysis)
예를 들어, 고차원의 데이터 feature[a,b,c,d] 를 처리하고 비교한다고 생각해보면
[a,b], [a,c], [a,d], [b,c], [b,d], [c,d] ...
이처럼 feature가 많으면 많을 수록 분석하기가 힘들고, Overfitting이 일어난다.
Overfitting: 데이터를 분석하여 pattern만 뽑아내고 싶은데, noise까지 함께 저장되는 현상
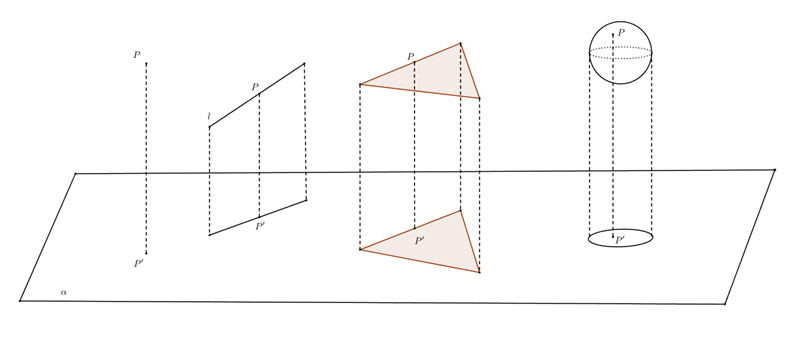
이런 현상을 피하기 위해 고차원의 데이터를 정사영하여 주성분으로 차원을 축소하는 방법.
차원을 축소하기 위해서 Eigenvalue, Eingevector 가 필요하다.
🏁 내 코드
import pandas as pd
from sklearn.preprocessing import StandardScaler, Normalizer
from sklearn.decomposition import PCA
print('Data= \n',features)
scaler = StandardScaler() # 다른 데이터들을 비교 가능하게 만들어줌
features = pd.DataFrame(scaler.fit_transform(features),columns=['bill_length_mm','bill_depth_mm','flipper_length_mm','body_mass_g']) # 노말라이즈된 데이터 얻음
print('\n Standardized features=', features)
pca = PCA(n_components=2) # 2차원으로 축소 PCA 실행
pc_df = pd.DataFrame(pca.fit_transform(features), columns=['PC1','PC2']) # PCA 시행하여 변환된 주성분으로 데이터 프레임 생성
ratio = pca.explained_variance_ratio_ # variance ratio
✔️ scaler = StandardScaler()
scale을 맞춰줘야 하는 이유?
✔️ pca = PCA(n_components=None)
pca: 주성분을 뽑아내는 것
n_components: 주성분을 몇 개로 할 것인지 정하기
