Abstract
U-net 은 CV 중에서도 Biomedical segmentation 을 위해 나온 합성곱 신경망이다.
기존 FCN(Fully Convolutional Network) 을 수정하고 확장시키면서 적은 데이터로 빠르게 효과적인 성능을 뽑아낼 수 있었다고 한다.
어떻게 그렇게 할 수 있었는지, 함께 공부해보자.
본 논문에서 집중해야 할 U-net의 main topics 들은 다음과 같다.
Data Augmentation
- DNN 기반의 CV 모델이 막대한 양의 데이터를 가지고 학습했을 때에만 성공적이었다.
- U-net 은 강한 Augmentation 기법을 가지고 데이터를 효율적으로 사용할 수 있도록 했다.
Architecture
- End to end 방식을 활용하여 간단하고 빠른 구조를 가져감
- 대칭적인 수축 - 팽창 구조를 사용
-> 수축 : context capture - 전체적인 맥락. 숲!
-> 팽창 : localization - 지협적인 정보. 나무!
Introduction
FCN 이 무엇인지 부터 봐보자.
FCN 은 기존의 Visual Recognition task 의 Single Class Label.
즉 하나의 이미지가 결국 무엇인지를 판단하는 것에서, 각 픽셀이 무엇인지를 판단하는 단계로 넘어가 Segmentation 을 하는 목적으로 나온 녀석.
Segmentation Task 에서는 이미지의 위치정보 가 정말 중요한데, FC layer 를 거치려고 하는 순간 벡터화 되고, 그렇게 되면 위치정보는 사라짐!
- 그래서, Fully - Convolutional 한 네트워크를 고안해냈으니, 그것이 FCN 이다!
- 높이와 너비 (즉 이미지의 위치정보) 로는 정보축약을 하지 않고, 채널에 대해서만 축약을 하여 위치정보를 유지할 수 있도록 함.
FCN 은 downsampling - upsampling 이라는 것을 하는데,
Down Sampling
- 우리가 아는 CNN의 구조와 유사한데, FC layer 만 없는 구조다
- FC layer 를 1 x 1 conv 로 대체해서, 채널 정보 축약 - 피쳐맵 형성
Up Sampling
- Feature map 을 다시 input img 사이즈로 키워주는 역할
- 정보가 축약된 녀석을 다시 부풀리는 과정, Convolution의 반대 개념인 Transposed Convolution을 이용 (여기서도 Parameter 학습 일어남)
- 영역의 대푯값을 가져다가 축약하는 Conv <-> 해당 영역의 대표값으로 나머지 값들을 추론하는 Up Sampling
- 부풀려서 값을 채워넣고, 남은 값들에 bilinear interpolation 방식으로 내분값들을 넣음.
근데 이 저자들이 보기에 FCN 도 조금의 단점이 존재했다!
기존 Sliding Window 방식
- Sliding Window 방식을 사용해서 input img 를 싹 돌면, 겹치는 부분도 너무 많고, 하나하나 다 conv 연산을 거쳐야 하기 때문에 너무 느림
Localization Vs Context
- 지협적인 정보의 정확도를 높이기 위해서 필터를 작게하면, 전체 input 의 맥락을 잘 모름
- 전체 input 의 맥락을 잘 보기 위해서 필터를 키우면, 또 지협적인 정확도가 떨어짐
- 이러한 Trade off 가 존재
저자들은 이 FCN을 발전시켰는데, 한번 아키텍쳐와 함께 봐보자.
Network Architecture

기존 FCN 의 또 다른 특징 중 하나는 Skip Connection 인데, 이는 Upsampling 시의 정확도를 높여주기 위해, 이전 Layer 의 Feature map을 넘겨받아 더해주는 것이다.
위 아키텍쳐가 바로 U-net 이다.
U-net 은 좌측의 input 부근의 Contraction Path 와 우측 output 부근의 Expansive Path , 그리고 그 둘을 이어주는 Bottle neck 부분으로 이루어져 있다.
Contraction Path (수축 경로)
- 이 부분은 흔히 아는 convolution 연산을 하는 부분이다.
- 2번의 3 x 3 convolution 연산 이후 ReLU activation Func 을 사용한다. 패딩은 따로 하지 않는다.
- 2 x 2 , stride = 2 로 max pooling 을 진행
- 이때 각 단계에서 나온 Feature Map 을 crop 하여 저장해 놓는다.
(Crop 을 하는 이유는, Padding 이 없기 때문에 input path 와 output path 의 사이즈 차이가 나기 때문이다.)
Bottle Neck
- 3 x 3 conv , ReLU * 2
- Drop Out
Expansive Path (확장 경로)
- up sampling 을 하는 구간이다. 똑같이 3 x 3 convolution 연산을 두번 한다.
- 그 후 2 x 2 max pooling 대신에, 같은 scale 의 up convolution 연산을 한다.
- 이때, 각 단계에서 대응되는 Contraction Path 의 Feature map 을 부착한다! (Conv 연산 전에) => Skip Connection 효과
U-net 의 구조는 확장과 팽창 구조가 알파벳 U 처럼, symmetric 하다는 특징이 있다.
구조를 배웠으니 이제 U-net 만의 특별한 방법들을 알아보자.
Specific Strategies
U-net의 Patch 탐색 방식
- U-net 은 sliding window 방식 대신, 중복되지 않은 patch 로 데이터를 구성함.
- 중복되는 부분을 제거하여 필요 이상의 검증을 제거.
- 시간과 연산 측면에서 많은 낭비를 줄임.
Mirroring Extrapolate & Overlap-Tile Method
- U-net 은 padding 을 사용하지 않고, conv 연산을 수행
(항상 출력<입력)- 그래서, 특정 부분에 대한 segmentation 을 하려면 그것보다 큰 게 들어가야 함.
- 그런데 끄트머리에 있는 애들은 어떻게 해야할까?
- 추가적으로 필요한 부분에 대하여 mirroring 을 하여 채워넣음
- 그리고, contraction 에서는 겹치지만 expansive 에선 겹치지 않도록 진행.
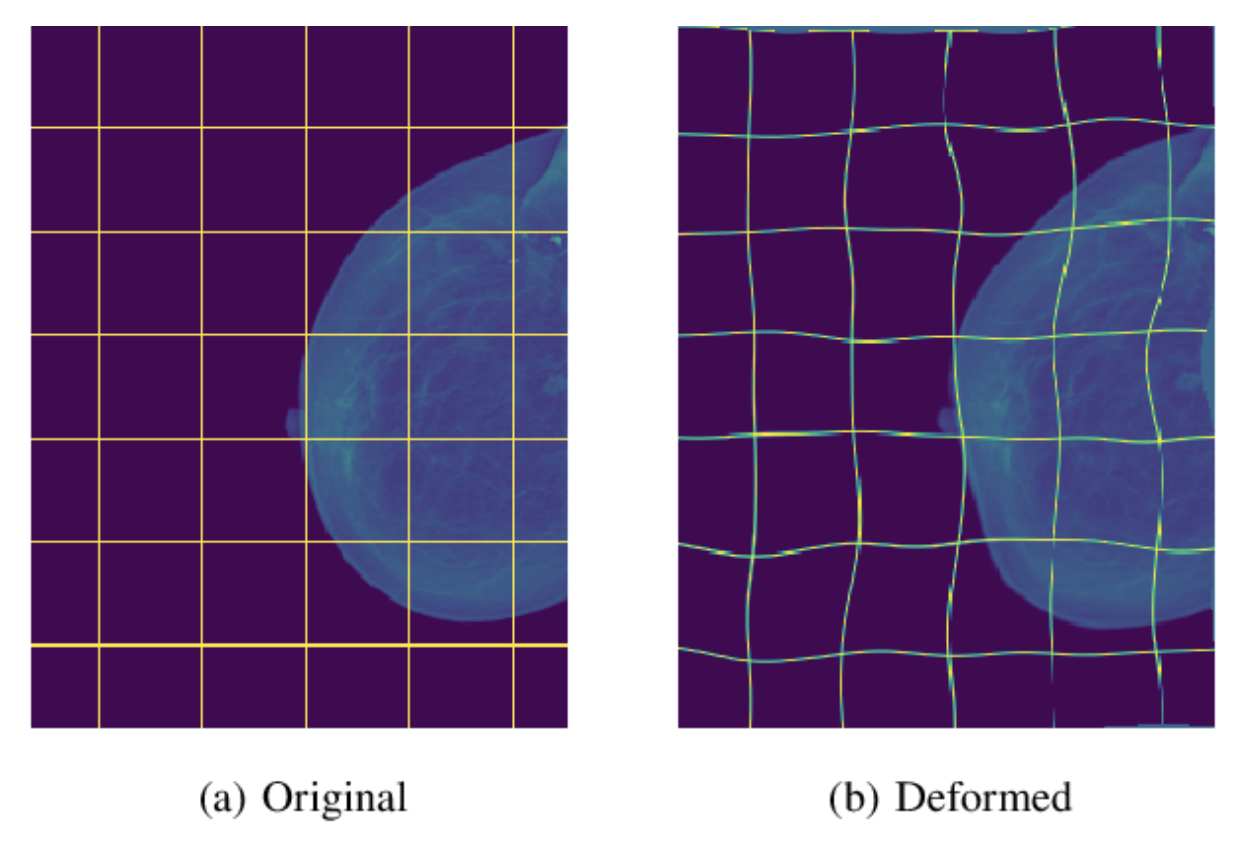
Data Augmentation- Elastic Deformation
- 저자들은 적은양의 데이터로 좋은 효과를 내기를 바라며 해당 모델을 구성했음.
- Data Augmentation 기법에 큰 관심을 기울임
- 일반적인 증강 방식 말고도, 특히 Biomedical task 에 어울리는 Elastic Deformation 기법을 사용함
- 이는 data metric위 임의의(Gaussian Dist) 벡터를 조정하여 굴곡을 줌으로써 생물학적인 특성에 더 강건하도록 만들어줌
Training
U-net 을 training 한 방법에는 biomedical data 에서 Augmentation 을 잘 수행하기 위한 노력들이 드러나 있다! 한번 봐보자
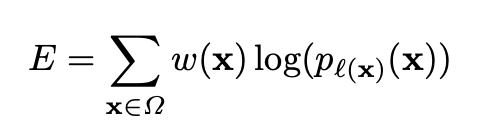
이게 바로 U-net의 Loss Func 이다.

여기서 W(x) 는 학습되는 가중치이고, 뒤에 달려있는 P_k(x) 는 추론 값이다. 
여기서 k 는 해당 클래스 , K는 전체 클래스의 수를 의미한다.
ak(x)는 x 라는 pixel 에서 작용하는 activation function을 의미한다.
그렇기 때문에 고로 전체 클래스들에서 해당 클래스에 해당 픽셀이 포함되어 있을 확률, 즉 추론 값을 의미한다.
W(x)가 이제 좀 재미있는데,

W(x) 는 x 라는 픽셀에 얼마나 큰 가중치를 부여할지를 나타내는 부분이다.
Wc(x) 는 x가 속한 class 의 빈도수를 말한다.
그리고 그 뒤에 붙은 항이 이제 좀 특별하다.

세포들은 위와 같이 아주 딱 달라붙어 있는 경우가 많아서, 그 경계를 잘 학습해야 보다 정확한 segmentation 이 가능하다고 한다.
그래서 이들은 '경계'의 부분에 따로 더 큰 가중치를 주어, 결과적으로 Loss 값을 늘리므로써 '경계'를 더 잘 분류하겠다는 목표를 가지고 해당 term을 만들었다.
위 식에서 d1 은 해당 픽셀로부터 가장 가까운 세포 경계 와의 거리이고 d2 는 그 다음으로 가까운 세포 경계와의 거리이다.
Conclusion
구조도 구조인데 Augmentation 이나 Loss function 부분을 자신들의 목적에 맞게 만진 부분이 아주 재밌었던 것 같다. 사실 막 재밌진 않았다.
References
https://velog.io/@jeewoo1025/What-is-end-to-end-deep-learning
https://www.youtube.com/watch?v=O_7mR4H9WLk
국민대학교 박종혁 교수님 ; 231024-비전AI와비즈니스-세분화