knn과 k-means에 대해 공부한 개념과 이후 작성한 간단한 코드에 대한 글이다.
knn은 classification을 위해 사용하는 방법이고, kmeans는 clustering을 위해 사용하는 방법으로 알려져있다.
Brief Explanation: knn, k-means
1. knn
KNN == K Nearest Neighbor
지도학습 알고리즘 중 하나로 주변의 k개의 데이터를 보고 어떤 데이터의 class를 판단하는 방법이다.
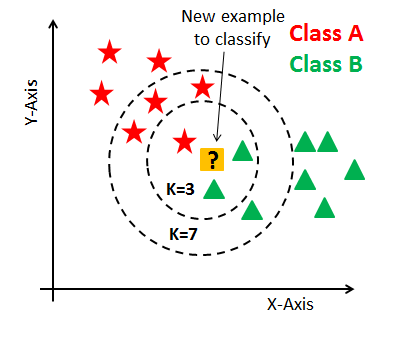
knn은 다른 알고리즘들과는 다르게 모델이 학습을 따로 하지 않고 training data를 저장해 두었다가 새로운 데이터를 분류하기 위한 경우에 계산을 위해 training data가 사용된다. 모델을 만드는 시간이 들지 않기 때문에 다른 모델보다 빠르다고 알려져 있다. 모델을 만들지 않는다는 의미에서 Lazy model이라고 불린다.
모델을 만들지 않고 새로운 input에 대한 분류를 할 수 있는 이유는 새로운 데이터 주변 k개의 데이터를 보고 많이 분포한 class로 분류를 하기 때문이다. 위의 이미지를 보면 k가 3일때는 주변에 위치한 가장 가까운 3개의 데이터를 보고 B class로 분류할 수 있지만, k가 7인 경우에는 A class로 분류된다. 여기서 알 수 있듯 knn에서는 적당한 k 값을 정하는 것이 분류의 정확도에 영향을 준다. 또한 다수결에 의해 분류가 되기 때문에 k는 보통 홀수를 사용한다.
가까운 k개의 training data를 계산하기 위해서 거리를 구하는 공식이 사용되는데, 아래 이미지에 있는 metrics들이 주로 많이 사용된다.

knn을 구현하기 위한 scikit-learn의 KNeighborsClassifier 모듈에서는 metric parameter를 사용해 거리 구하는 방식을 변경할 수 있다. "minkowski"방식이 dafault로 설정되어 있다.
About Cross-validation
knn은 모델을 학습하는게 아니라서 모델 학습을 위해 k-fold를 사용할 필요는 없지만
최적의 k를 구하기 위해서 k-fold를 사용하기도 한다.
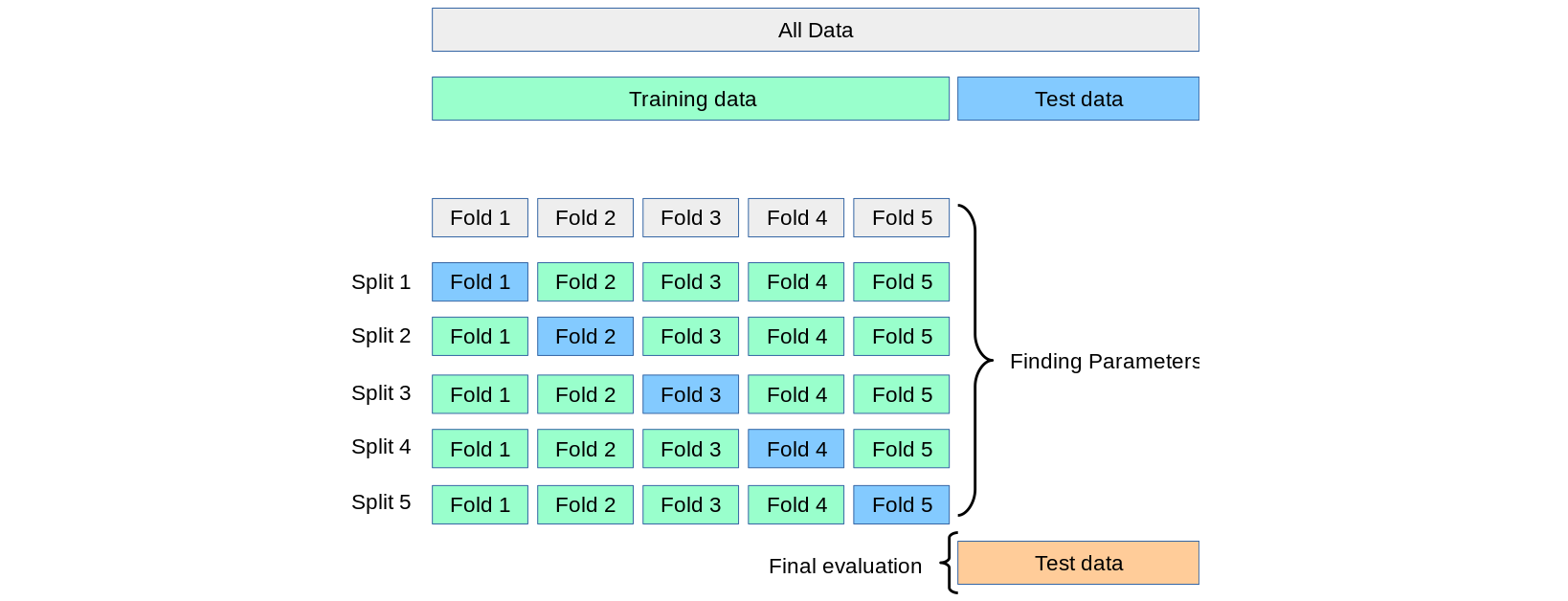
위 그림은 k가 5인 경우에 k-fold가 수행되는 과정을 그림으로 나타낸 것이다. 원본 데이터를 5부분으로 나누고 초록 부분은 training data로, 파란 부분은 testing data로 사용해서 model을 학습시키는데, 이 방법으로 데이터가 너무 적어 학습이 잘 되지 않거나 모든 데이터를 사용해 학습시켜 발생하는 과적합(overfitting) 문제를 줄일 수 있다.
- k-fold를 사용해 knn에서의 최적의 k를 찾는 코드
from sklearn.model_selection import cross_val_score
from sklearn.neighbors import KNeighborsClassifier
score_list = []
for k in range(1,101): # 1~100까지의 후보 k
knn_classifier = KNeighborsClassifier(n_neighbors=k)
# 'x' is features and 'y' is target data.
# 'cv' determines cross-validation splitting number -> 10 k-fold
accuracy_score = cross_val_score(knn_classifier,x,y,cv=10,scoring ="accuracy")
score_list.append(accuracy_score)
opt_k = score_list.index(max(score_list))2. k-means
k-means clustering기법은 비지도 학습 중 클러스터링 기법 중 하나이다. k는 cluster의 개수를 의미하는데 cluster를 나누는 과정은 다음과 같다.

- 임의로 k개의 centroid를 정한다.
- 각 점은 cluster의 centroid와 자신의 거리를 계산해서 가장 가까운 cluster에 속한다.
- 모든 점이 cluster에 속하면, 각 cluster의 centroid(중심)를 다시 계산한다.
- 바뀐 centroid에 따라 2,3을 반복한다.
- 더 이상 centroid가 변하지 않으면 cluster가 정해진 것이다.
이런 방식으로 k-means가 작동한다.
About Dataset: Heart disease
13개의 feature을 사용해 heart disease가 있는지(1), 없는지(0) 분류한다.
사용한 초기 데이터의 target은 0(no presence)부터 4까지의 정수로 표현되어 있어서 0을 제외한 나머지 숫자를 1로 바꾸는 과정을 학습 전에 먼저 하였다. target data refining 과정은 다음과 같은 코드를 사용해서 간단히 구현하였다.
# to change the target data to 0 and 1
# 0 means 'No heart disease', 1 means 'heart disease'
df['num'] = df['num']>0
df['num'] = df['num'].map({True:1, False:0})df는 모든 feature data와 target data를 담고있는 DataFrame이고, df['num']가 target data가 있는 열이다.
- Features: 'age', 'sex', 'cp', 'trestbps', 'chol', 'fbs', 'restecg', 'thalach', 'exang', 'oldpeak', 'slope', 'ca', 'thal'
- Target: integer number - 0 or 1
사용한 데이터는 UCI Machine Learning Repository에서 제공하는 데이터인데, 더 자세한 정보를 알고 싶다면 webSite를 방문하는 것을 추천한다.
About code
Environment
- Jupyter notebook - python3.6
- used modules: scikit-learn, numpy, pandas, matplotlib, seaborn
Common part: Read and Refine the data
- 데이터를 읽고 feature은 x에 target은 y에 저장한다.
import pandas as pd
from sklearn.preprocessing import scale
df = get_data()
# seperate target(y) from features(x) and scale x
y = df.pop('num').values
x = pd.DataFrame(scale(df.values),columns=cols)- 데이터를 읽어오는
get_data함수
import numpy as np
def get_data():
# features
cols = ['age','sex','cp','trestbps','chol','fbs','restecg','thalach','exang','oldpeak','slope','ca','thal','num']
# URL to download the dataset
url ='https://archive.ics.uci.edu/ml/machine-learning-databases/heart-disease/processed.cleveland.data'
df = pd.read_csv(url,index_col=False, header=None, names=cols)
# replace '?' with 'np.nan'
df = df.replace({'?':np.nan})
# drop rows with nans
# In this dataset, only looses 6 rows.
df.dropna(inplace=True)
df = df.astype('float64')
# Refine the target data - binarization
df['num'] = df['num']>0
df['num'] = df['num'].map({False:0, True:1})
return df이 부분은 knn과 k-means가 동일하다.
1. knn
- knn classifier 만들기
from sklearn.model_selection import train_test_split
from sklearn.neighbors import KNeighborsClassifier
# split data into train and test data
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3)
# fitting model with train data
knnc = KNeighborsClassifier(n_neighbors=5)
model = knnc.fit(x_train,y_train)
# predict with test data
y_pred = model.predict(x_test)
# calculate the evaluating values
calc_performance(y_test,y_pred)먼저 train data와 test data를 나누기 위해 train_test_split모듈의 test_size로 비율을 지정(train:test = 7:3)한다.
sklearn.neighbors.KNeighborsClassifier를 사용해 k=5로 하는 classifier(knnc)를 만들어 학습(model)시킨다. 이후 test data로 예측한 결과(y_pred)를 통해 모델의 성능을 평가(cal_performance())한다.
- performance 계산하는 함수
from sklearn.metrics import accuracy_score, precision_score, recall_score, f1_score
from sklearn.metrics import confusion_matrix, ConfusionMatrixDisplay
def calc_performance(y_true,y_pred):
# accuracy
acc = accuracy_score(y_true,y_pred)
print(f" * Accuracy score: {acc:.2f}")
# confusion_matrix
cm = confusion_matrix(y_true,y_pred)
tn,fp,fn,tp = cm.ravel()
# Plotting confision matrix
disp = ConfusionMatrixDisplay(confusion_matrix=cm)
disp.plot()
# precision
pre = precision_score(y_true,y_pred)
print(f" * precision: {pre:.2f}")
# recall(sensitivity)
rec = recall_score(y_true,y_pred)
print(f" * recall: {rec:.2f}")
# specificity(true negative rate)
tnr = tn/(tn+fp)
print(f" * true negative rate: {tnr:.2f}")
# f-measure
fs = f1_score(y_true,y_pred)
print(f" * f1-score: {fs:.2f}")모델의 성능을 평가하기 위한 metrics는 scikit-learn에서 제공하고 있다.
그 중 여기서는 accuracy, confusion matrix, precision,recall, f1-score를 사용하고, 추가로 specificity도 계산하였다.
- confusion matrix: 두개의 class로 분류하는 모델의 경우 아래의 그림처럼 예측해서 얻은 값과 실제 값에 대한 도표를 그릴 수 있는데 이를 confusion matrix라고 한다. TN은 True Negative, FP는 False Positive, FN은 False Negative, TP는 True Positive를 의미한다.
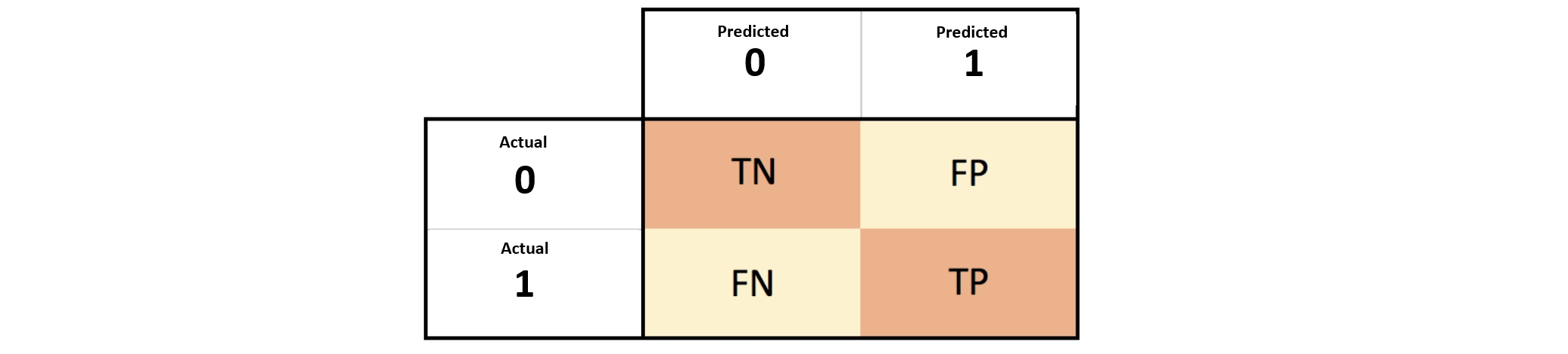
- accuracy: 전체 데이터 중 맞게 예측한 데이터의 비율 = (tp+tn)/(tp+fp+tn+fn)
- precision: positive라고 예측한 것들 중 실제 positive의 비율 = tp/(tp+fp)
- recall(true positive rate): 실제 positive 중 맞게 예측한 positive의 비율 = tp/(tp+fn)
- specificity(true negative rate): 실제 negative 중 맞게 예측한 negative의 비율 = tn/(tn+fp)
- f1 score: precision과 recall의 조화평균 = 2*precision*recall / (precision+recall) = 2tp/(2tp+fn+fp)
더 자세히 알고싶다면 webLink의 설명을 읽어보는 것을 추천한다.
아래의 모듈을 사용해서 한번에 precision, recall, f1-score를 출력하는 방법도 있다.
from sklearn.metrics import classification_report
tmp = classification_report(y_true,y_pred)
print(tmp)2. k-means
- k-means cluster 만들기
from sklearn.cluster import KMeans
# k-means clusteing
estimator = KMeans(n_clusters=2, random_state=106)
estimator.fit_predict(x)
# create a table containing clustered data
table = x.copy()
table['cluster'] = estimator.labels_sklearn.cluster.KMeans를 사용해 0과1에 대한 2개의 클러스터(n_clusters)를 설정하고, cluster의 centroid 초기화를 위한 seed number인 random_state를 설정한다.
feature를 담고 있는 x를 fitting하면 estimator.labels_를 통해 labelling된 cluster(0 or 1)를 얻을 수 있다.
- cluster 그리기
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
def draw_clusters(table):
# run PCA to reduce the dimension
pca = PCA(n_components=2)
new_data = pca.fit_transform(table)
# pc means 'principal component'
pca_df = pd.DataFrame(new_data,columns=['pc1','pc2'])
# drawing clusters
sns.scatterplot(x="pc1", y="pc2", hue=table['cluster'], data=pca_df)
plt.title('K-means Clustering in 2D')
plt.savefig(path_to_save+'kmeans_result.png') #can be omitted
plt.show()Heart disease data는 feature가 13개라서 cluster를 시각적으로 표현하기 위해서는 13차원을 그려야 하는데, 이는 불가능하다. 시각적으로 보이기 위해서는 2차원이나 3차원으로 차원을 축소해야 하고 이를 위해 scikit-learn에서는 sklearn.demoposition.PCA를 제공한다. PCA 모듈을 사용하면 feature들로부터 주성분(principal component)을 만들어 내는데, 여기서 주성분은 13개의 feature 중 몇개가 아니고 새롭게 계산된 값이 나온다.
위의 코드에서는 PCA로 차원축소를 한 후에 matplotlib와 seaborn을 사용해 시각화하였다.
Full Code is HERE!
About result
1. knn
train_dataset으로 학습시킨 knn classifier에 test_dataset을 넣어 예측한 결과의 accuracy를 출력하였다. k-fold를 사용해 교차 검증(cross-validation)을 한 경우에는 각 fold의 accuracy와 평균 accuracy를 출력하도록 하였고, 교차검증을 하지 않은 경우에는 accuracy 이외에도 precision, recall,f1-score과 confusion matrix와 같은 여러가지 evaluating value들을 출력하도록 하였다.
그래서 출력 결과는 다음 이미지처럼 나온다.
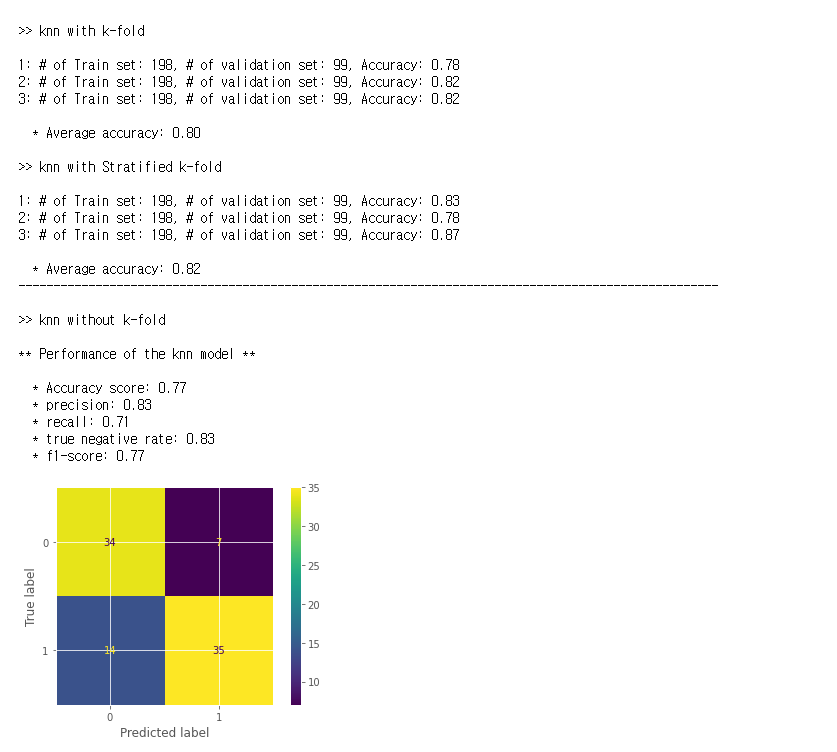
2. k-means
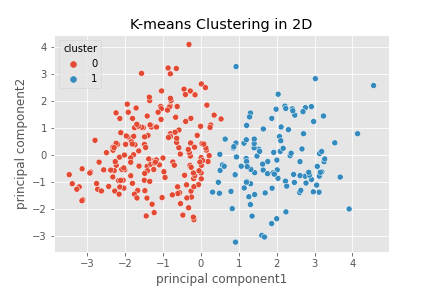
kmeans는 clustering을 하는데, 위 코드에서는 heart disease가 있는지 없는지를 구분하기 위해 그 값을 각각 1과 0으로 하였다. 그래서 2개의 cluster가 생기는데 13개 요인을 모두 포함하는 시각적 이미지는 얻을 수 없기 때문에 2D나 3D로 차원을 축소하여 그린다. 주성분의 개수를 정하기 위해 설명분산값(sklearn.decomposition.PCA.explainedvariance_ratio)을 구해보면 2개의 주성분으로 축소하는 것과 3개의 주성분으로 축소하는 것에 의미있는 차이가 있지 않은 것으로 보여졌다.
그래서 2차원으로 차원축소를 하였고 출력되는 2D diagram은 아래와 같다.
References
📷 Image Source
- knn - scikit-learn 분류
- distance metrics - sklearn.neighbors.DistanceMetric
- Confusion Matrix - Applied Deep Learning with Keras
- Cross validation - Cross-validation: evaluating estimator performance
📋 Referenced sites
- knn - wikipedia & k-최근접이웃 & KNN 최근접 이웃 알고리즘
- K-Fold, Stratified K-Fold - python을 통한 교차검증 & scikit-learn에서의 cross-validation & 사이킷런에서의 교차검증, Kfold 정리
- Cross validation - 최적의 k를 위한 k-fold
- kmeans - wikipedia & K-평균 클러스터링
⭐

