변수 분포 문제
일반화된 모델을 학습하는데 어려움이 있는 분포를 가지는 변수가 있어, 일반화된 모델을 학습하지 못하는 문제 발생.
문제 정의
변수 범위에서 많이 벗어난 아주 작은 값이나 아주 큰 값으로, 일반화된 모델을 생성하는데 악영향을 끼치는 값으로 이상치를 포함하는 레코드를 제거하는 방법
밀도 기반 군집화
DBSCAN 등의 밀도 기반 군집화 기법은 군집에 속하지 않는 샘플을 이상치라 판단하여 제거.
다만 밀도 기반 군집화 모델의 파라미터 튜닝이 어렵다.
sklearn.cluster.DBSCAN(군집화)
주요인자
- eps : 이웃이라 판단하는 반경
- min_samples : 중심점이라 판단하기 위해 eps 내에 들어와야 하는 최소 샘플 수
- metric : 사용하는 거리 척도
- 속성 - .labels_ : 각 샘플이 속한 군집 정보(-1 = 이상치)
실습
다음과 같은 데이터로 실습을 해보자.
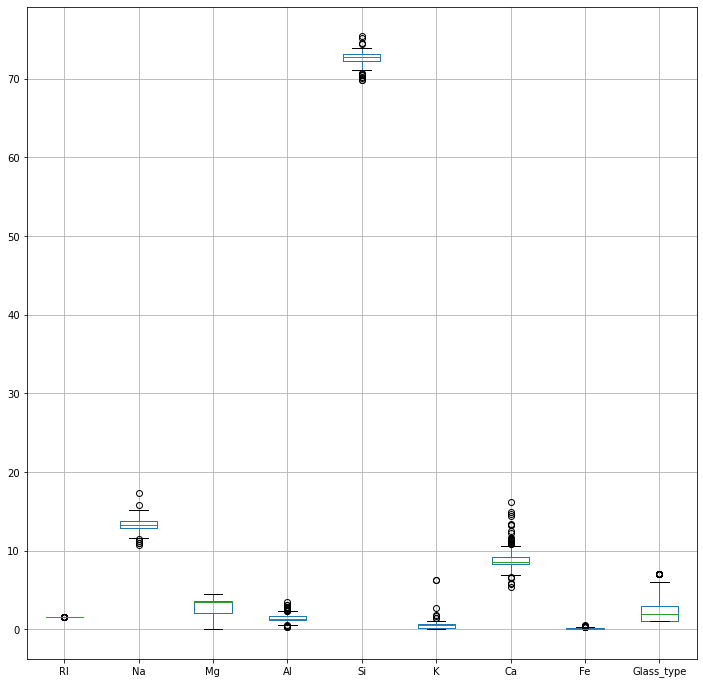
각 컬럼의 스케일을 박스 플롯으로 확인해면 다음과 같다.
DBSCAN의 eps는 거리 기반으로 군집화를 하는데, 아래의 데이터 스케일이 다르기에 scipy의 cdist를 참고하도록 한다.
데이터 분리
먼저 데이터를 분리시킨다.
# 특징과 라벨 분리
X = df.drop(['Glass_type'], axis = 1)
Y = df['Glass_type']
# 학습 데이터와 평가 데이터 분리
from sklearn.model_selection import train_test_split
Train_X, Test_X, Train_Y, Test_Y = train_test_split(X, Y)분리 후 Train_X의 shape를 확인해보자. 군집화 후에 이상치를 제거한 다음의 shape와 비교해 볼 것이다.

샘플간 거리 파악
샘플 간 거리 측정
from scipy.spatial.distance import cdist
from sklearn.cluster import DBSCAN
DM = cdist(Train_X, Train_X) # 거리 행렬 => DBSCAN의 파라미터를 설정하기 위함
np.quantile(DM, 0.1) # 샘플 간 거리의 10% quantile이 0.6622정도임을 확인
print(np.quantile(DM, 0.1))
0.6622273395331424군집화
샘플 간 거리를 바탕으로 군집화를 진행해보자.
# eps = 0.67
cluster_model = DBSCAN(eps = 0.67, min_samples = 3).fit(Train_X)이상치 제거
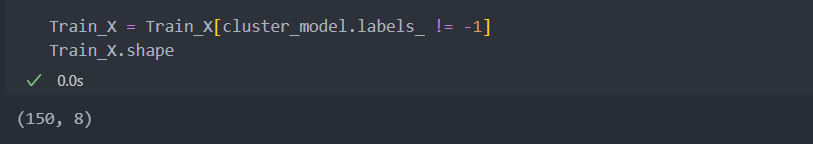
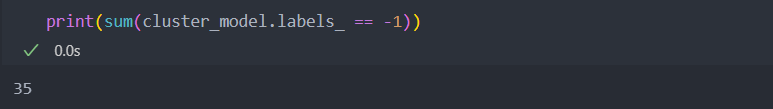
그리고 라벨이 -1인 것, 즉 이상치라 판단한 데이터의 수를 확인해보자.
이상치의 수가 35개로, 35/160은 이상치가 많아서 하이퍼 파라미터를 조절을 해야 할 것이다.
이상치의 개수를 판단하기 위해 eps와 min_samples 바꿔가며 이상치가 10% 미만(대략 15개)인 것들만 보도록 하자.
반복문으로 해서 원하는 이상치의 수치를 파악하고 진행하면 된다.
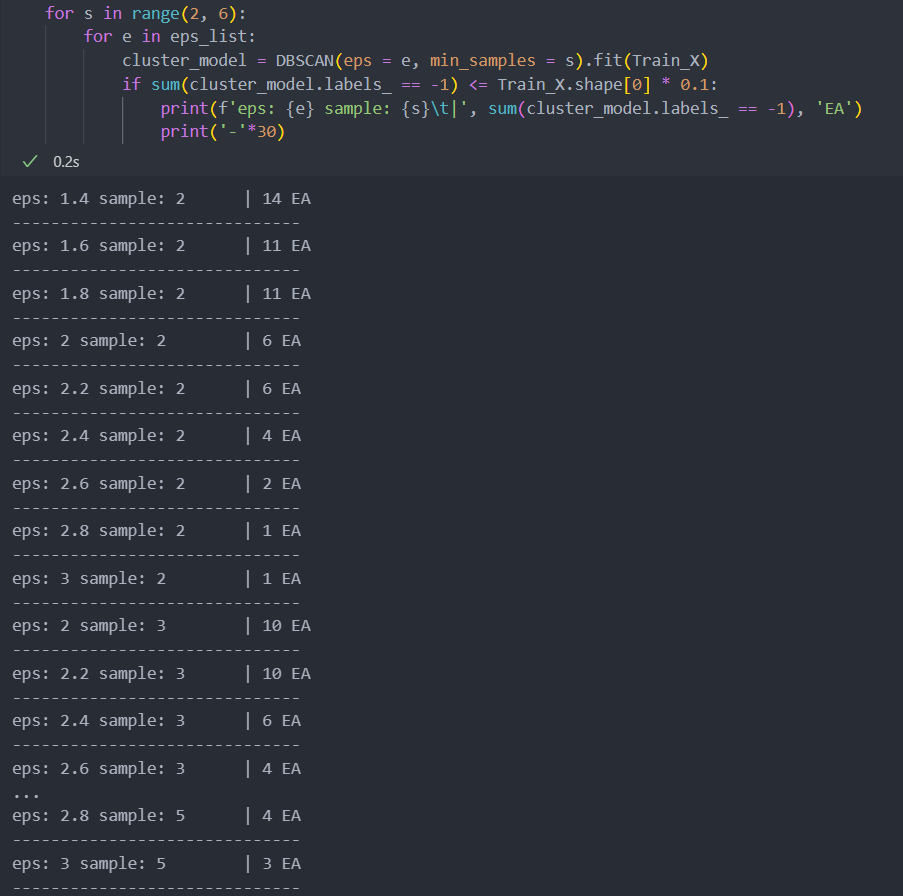
eps = 2, min_samples = 3를 선택하고 이상치를 제거 한 후의 데이터 개수를 보자.