
변수 분포 문제
일반화된 모델을 학습하는데 어려움이 있는 분포를 가지는 변수가 있어, 일반화된 모델을 학습하지 못하는 문제 발생.
문제 정의
회귀, 신경망, SVM과 같이 선형식이 모델에 포함되는 경우, 특징 간 상관성이 높으면 파라미터 추정이 어렵고 추정할 때마다 결과가 달라질 수 있다.
트리 계열의 모델은 모델 예측에는 영향을 받지 않지만, 상관성이 높은 변수 중 소수만 모델에 포함되기 때문에 설명력이 크게 영향을 받는다.
VIF(분산 팽창 계수) 활용
분산 팽창 인수(VIF) 값은 다중회귀분석에서 독립변수가 다중 공산성(Multicollnearity)의 문제를 갖고 있는지 판단하는 기준이며, 주로 10보다 크면 그 독립변수는 다중공산성이 있다고 한다.
Variance Inflation Factors(VIF)는 한 특징을 라벨로 간주하고, 해당 라벨을 예측하는데 다른 특징을 사용한 회귀 모델이 높은 R^2(회귀 모델의 적합성, 0~1 사이)을 보이는 경우 해당 특징이 다른 특징과 상관성이 있다고 판단한다.
그래서 VIF(VIF = 1 / 1 - R^2)가 높은 순으로 삭제하거나 VIF가 10 이상인 경우 삭제한다.
PCA(주성분 분석) 활용
PCA는 데이터 하나 하나에 대한 성분을 분석하는 것이 아니라, 여러 데이터들이 모여 하나의 분포를 이룰 때 이 분포의 주성분(분산이 가장 큰 방향백터)을 분석한다.
2차원이면 2개의 3차원이면 3개의 주성분이 생기며 이들은 서로 수직인데, 설명력이 높은 주성분을 가지고 이를 투영하고 차원을 축소하여 특징 간 상관성을 줄이는 방법을 사용한다.
from sklearn.decomposition import PCA
주요인자 : ncomponents - 사용할 주성분 개수를 나타내며, 이 값은 기존 차원 수(컬럼 수)보다 작아야 함.
주요속성 : explained_variance_ratio - n_components개의 주성분이 원 데이터의 분산의 설명력을 얼만큼 가지는지의 수치
실습
다음과 같은 데이터를 가지고 VIF, PCA를 활용하여 상관성을 제거해보자.
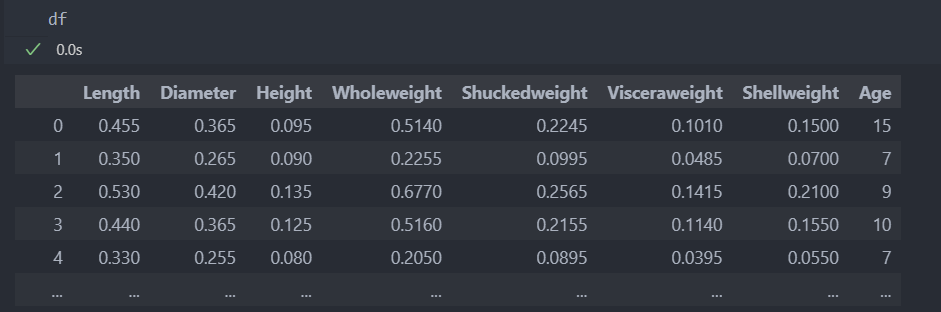
일단 데이터를 data, label로 나누고 학습과 평가 데이터로 나누자.
# 특징과 라벨 분리
X = df.drop(['Age'], axis = 1)
Y = df['Age']
# 학습 데이터와 평가 데이터 분리
from sklearn.model_selection import train_test_split
Train_X, Test_X, Train_Y, Test_Y = train_test_split(X, Y)그리고 데이터들 간의 상관성을 파악해보자.
매우 높은 상관 관계를 가지고 있음을 알 수 있다.
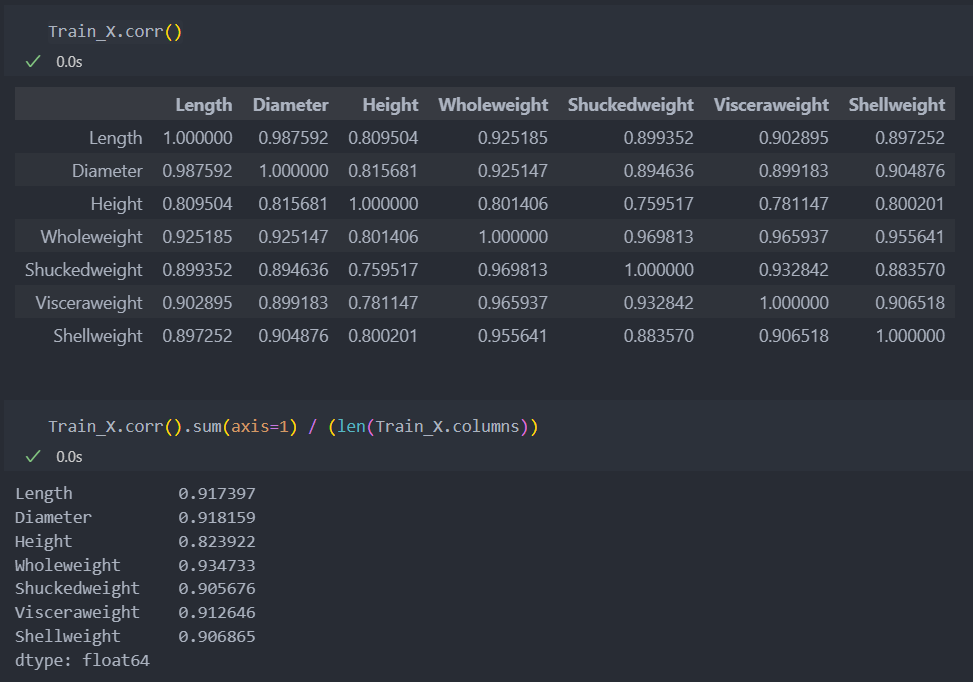
VIF
회귀 모델을 사용하여, 임의의 한 특징을 라벨로 간주하여 다른 특징들로 해당 라벨을 맞추기 위한 모델을 학습한 다음, 그 모델에 R^2을 측정.
# VIF 계산 : LinearRegression 사용
from sklearn.linear_model import LinearRegression as LR
VIF_dict = dict()
for col in Train_X.columns: # 컬럼들을 순회하며 해당 컬럼을 라벨로 간주하여 사용.
model = LR().fit(Train_X.drop([col], axis = 1), Train_X[col]) # fit(data, label)
r2 = model.score(Train_X.drop([col], axis = 1), Train_X[col]) # LinearRegression의 score가 r2 점수임
VIF = 1 / (1 - r2) # VIF 계산
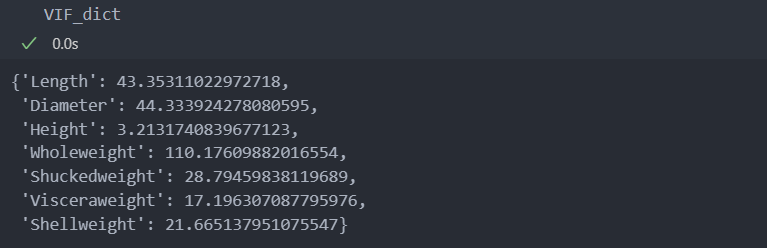
VIF_dict[col] = VIF # 계산한 VIF를 커럼별로 저장 후 확인.VIF_dict를 확인해보자.
'Height' 컬럼을 제외하고 모두 10이상으로 높은 점수를 갖는 것으로 보아 VIF를 활용하긴 어려워 보인다.
실습을 위해 30 미만인 컬럼들을 가지고 진행해 보자.
비교 진행을 위해 '신경망' 모델을 사용하여 VIF를 사용하기 전과 후를 비교해보자.
score를 비교해보면 매우 미세하게 성능이 좋아진 것을 확인할 수 있다.
error score이기에 낮을 수록 좋은 성능이라 판단한다.
from sklearn.neural_network import MLPRegressor as MLP
from sklearn.metrics import mean_absolute_error as MAE
# MLPRegressor에 기존(전체) 데이터 적용
model = MLP(random_state = 29, max_iter = 500)
model.fit(Train_X, Train_Y)
pred_Y = model.predict(Test_X)
score = MAE(Test_Y, pred_Y)
print('score: ', score)
score: 1.6578211710528141
# MLPRegressor에 VIF 적용 데이터
# VIF 점수가 30점 미만인 특징만 사용하였을 때
selected_features = [key for key, val in VIF_dict.items() if val < 30]
model = MLP(random_state = 29, max_iter = 500)
model.fit(Train_X[selected_features], Train_Y)
pred_Y = model.predict(Test_X[selected_features])
score = MAE(Test_Y, pred_Y)
print('VIF score:', score)
VIF score: 1.5962581691050202PCA
PCA는 기존의 컬럼 중에서 n_components의 개수만큼 사용하는 것이 아니라, 새로운 n_components개 만큼의 컬럼을 새롭게 생성(투영하여 차원 축소)하여 사용한다.
n_components를 3개로 생성하여 진행해보자.
from sklearn.decomposition import PCA
# 학습
PCA_model = PCA(n_components = 3).fit(Train_X) # 인스턴스 및 학습 진행
# 적용
Train_Z = PCA_model.transform(Train_X)
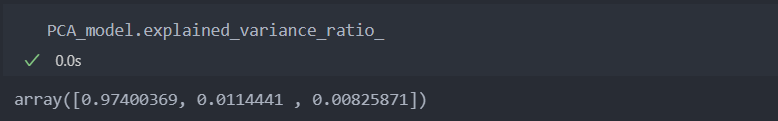
Test_Z = PCA_model.transform(Test_X)그리고 explainedvariance_ratio를 보면 97%, 1.1%, 1%미만으로 하나의 주성분이 97%의 설명력을 갖는 것을 볼 수 있다.
7개의 특성을 가지는 데이터를 5개부터 줄여가며 살펴봐도 되겠지만, 아래와 같이 한 주성분이 모든 설명력을 가지고 있어서 필요가 없어 보인다.
만약 설명력이 골고루 분포되어 있다면 가장 고르게 분포되는 것들만(보통 주성분을 더하여 90%정도 선에서) 사용한다.
위의 기존/VIF 적용과 score를 비교해보자.
조금 더 좋아진 것을 볼 수 있다.
model = MLP(random_state = 29, max_iter = 500)
model.fit(Train_Z, Train_Y)
pred_Y = model.predict(Test_Z)
score = MAE(Test_Y, pred_Y)
print('PCA score:', score)
PCA score: 1.4982701312765596