데이터 전처리
결측치 처리
NaN,Null값은 허용되지 않는다.- 따라서 이러한 결손값은 고정된 다른 값으로 변환해야 한다.
1. 데이터 인코딩
- 사이킷런의 머신러닝 알고리즘은 문자열 값을 허용하지 않는다.
- 따라서 문자열 값을 인코딩해서 숫자형으로 변환하는 과정을 거쳐야한다.
- 카테고리형 피처: 코드 값으로 표현
- 텍스트형 피처: 피처 벡터화(feature vectorization)등의 기법으로 벡터화 하거나 불필요한 피처라고 판단되면 삭제
1-1. 레이블 인코딩
- 사이킷런의 레이블 인코딩(Label Encoding)
LabelEncoder클래스로 구현한다. LabelEncoder를 객체로 생성한 후fit()과transform()을 호출해 인코딩을 수행한다.
from sklearn.preprocessing import LabelEncoder
items = ['TV', '냉장고', '전자레인지', '컴퓨터', '선풍기', '선풍기', '믹서', '믹서']
encoder = LabelEncoder()
encoder.fit(items)
labels = encoder.transform(items)
print('인코딩 변환값: ', labels)인코딩 변환값: [0 1 4 5 3 3 2 2]
TV는 0, 냉장고는 1, 전자레인지는 4, 컴퓨터는 5, 선풍기는 3, 믹서는 2로 변환됐다.
print('인코딩 클래스: ', encoder.classes_)인코딩 클래스: ['TV' '냉장고' '믹서' '선풍기' '전자레인지' '컴퓨터']
문자열 값이 어떤 숫자 값으로 인코딩 됐는지 궁금하다면 classes_속성값으로 확인하면 된다.
레이블 인코딩은 간단하게 문자열 값을 숫자형 카테고리 값으로 변환한다. 하지만 숫자값의 경우 크고 작음에 대한 특성이 작용하여 예측 성능이떨어질 수 있다. (예를 들어서 1+2=3이라는 숫자에 대한 특성이 있지만, 냉장고+믹서=선풍기가 아니다!)
트리계열 알고리즘은 상관이 없으나 선형회귀 알고리즘에서 문제가 발생할 수 있다.
원-핫 인코딩(One-Hot Encoding) 은 레이블 인코딩의 이러한 문제점을 해결하기 위한 인코딩 방식이다.
1-2. 원-핫 인코딩
- 피처 값의 유형에 따라 새로운 피처를 추가해 고유 값에 해당하는 칼럼에만 1을 표시하고 나머지 칼럼에는 0을 표시하는 것이다.
- 원-핫 인코딩은 사이킷런에서
OneHotEncoder클래스로 변환이 가능하다. 단LabelEncoder와 다르게 약간 주의할 점은,- 입력 값으로 2차원 데이터가 필요하다는 것
OneHotEncoder를 이용해 변환한 값이 희소행렬 형태이므로 이를 다시toarray()메서드를 이용해 밀집 행렬로 변환해야 한다는 것이다.
from sklearn.preprocessing import OneHotEncoder
import numpy as np
items = ['TV', '냉장고', '전자레인지', '컴퓨터', '선풍기', '선풍기', '믹서', '믹서']
items = np.array(items)items를 확인해 보면 array(['TV', '냉장고', '전자레인지', '컴퓨터', '선풍기', '선풍기', '믹서', '믹서'], dtype='<U5') 다음과 같은 ndarray가 생성됐음을 확인할 수 있다.
items = np.array(items).reshape(-1, 1)이를 2차원 ndarray로 변환한다.
oh_encoder = OneHotEncoder()
oh_encoder.fit(items)
oh_labels = oh_encoder.transform(items)
print('원-핫 인코딩 데이터')
print(oh_labels.toarray())
print('원-핫 인코딩 데이터 차원')
print(oh_labels.shape)원-핫 인코딩 데이터 [[1. 0. 0. 0. 0. 0.] [0. 1. 0. 0. 0. 0.] [0. 0. 0. 0. 1. 0.] [0. 0. 0. 0. 0. 1.] [0. 0. 0. 1. 0. 0.] [0. 0. 0. 1. 0. 0.] [0. 0. 1. 0. 0. 0.] [0. 0. 1. 0. 0. 0.]] 원-핫 인코딩 데이터 차원 (8, 6)
toarray()를 이용해 밀집 행렬로 변환하고 출력하면 위와 같은 결과가 나온다.
판다스에는 원-핫 인코딩을 더 쉽게 지원하는 API가 있다. get_dummies()를 이용하면 된다.
import pandas as pd
df = pd.DataFrame({'item': ['TV', '냉장고', '전자레인지', '컴퓨터', '선풍기', '선풍기', '믹서', '믹서']})
pd.get_dummies(df)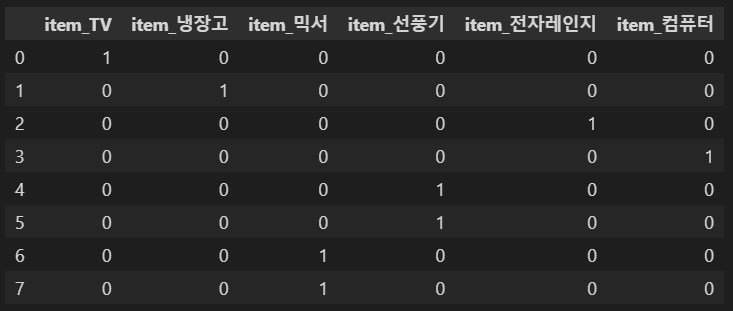
get_dummies()를 이용하면 가변수(dummy varaible)를 생성해(0과 1로만 이루어진 열 생성) 사이킷런의 OneHotEncoder와는 다르게 문자열 카테고리 값을 숫자형으로 변환할 필요 없이 바로 변환할 수 있다.
2. 피처 스케일링과 정규화
- 서로 다른 변수의 값 범위를 일정한 수준으로 맞추는 작업을 피처 스케일링(Feature Scaling)이라고 한다. 대표적인 방법으로 표준화(Standardization)와 정규화(Normalization)가 있다.
- 표준화: 데이터의 피처 각각이 평균이 0이고 분산이 1인 가우시안 정규 분포를 가진 값으로 변환하는 것을 의미
- 정규화: 서로 다른 피처의 크기를 통일하기 위해 크기를 변환해주는 개념
2-1. StandardScaler
- 앞서 말한 표준화를 쉽게 지원하기 위한 클래스
- 몇몇 알고리즘에서는 가우시안 정규 분포를 가질 수 있도록 데이터를 변환하는 것이 매우 중요하다!!
from sklearn.datasets import load_iris
import pandas as pd
iris = load_iris()
iris_data = iris.data
iris_df = pd.DataFrame(data=iris_data, columns=iris.feature_names)
print('feature들의 평균 값')
print(iris_df.mean())
print('\nfeature들의 분산 값')
print(iris_df.var())feature들의 평균 값 sepal length (cm) 5.843333 sepal width (cm) 3.057333 petal length (cm) 3.758000 petal width (cm) 1.199333 dtype: float64 feature들의 분산 값 sepal length (cm) 0.685694 sepal width (cm) 0.189979 petal length (cm) 3.116278 petal width (cm) 0.581006 dtype: float64
이제 StandardScaler를 이용해 각 피처를 한 번에 표준화해 변환한다.
StandardScaler 객체를 생성한 후에 fit()과 transform() 메서드에 변환 대상 피처 데이터 세트를 입력하고 호출하면 간단하게 변환된다.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(iris_df)
iris_scaled = scaler.transform(iris_df)
iris_df_scaled = pd.DataFrame(data=iris_scaled, columns=iris.feature_names)
print('feature들의 평균 값')
print(iris_df_scaled.mean())
print('\nfeature들의 분산 값')
print(iris_df_scaled.var())feature들의 평균 값 sepal length (cm) -1.690315e-15 sepal width (cm) -1.842970e-15 petal length (cm) -1.698641e-15 petal width (cm) -1.409243e-15 dtype: float64 feature들의 분산 값 sepal length (cm) 1.006711 sepal width (cm) 1.006711 petal length (cm) 1.006711 petal width (cm) 1.006711 dtype: float64
모든 칼럼 값의 평균이 0에 아주 가까운 값으로, 그리고 분산은 1에 아주 가까운 값으로 변환됐음을 알 수 있다.
2-2. MinMaxScaler
- 데이터값을 0과 1사이의 범위 값으로 변환한다. (음수가 있으면 -1에서 1값으로 변환)
- 데이터의 분포가 Min, Max Scale을 적용해 볼 수 있다.
from sklearn.preprocessing import MinMaxScaler
scaler = MinMaxScaler()
scaler.fit(iris_df)
iris_scaled = scaler.transform(iris_df)
iris_df_scaled = pd.DataFrame(data=iris_scaled, columns=iris.feature_names)
print('feature들의 최솟값')
print(iris_df_scaled.min())
print('\nfeature들의 최댓값')
print(iris_df_scaled.max())feature들의 최솟값 sepal length (cm) 0.0 sepal width (cm) 0.0 petal length (cm) 0.0 petal width (cm) 0.0 dtype: float64 feature들의 최댓값 sepal length (cm) 1.0 sepal width (cm) 1.0 petal length (cm) 1.0 petal width (cm) 1.0 dtype: float64
모든 피처에 0에서 1 사이 값으로 변환되는 스케일링이 적용됐음을 알 수 있다.
🚨학습 데이터와 테스트 데이터의 스케일링 변환 시 유의점
아래 코드를 통해 테스트 데이터에 fit()을 적용할 때 문제점을 알아보도록 하자.
from sklearn.preprocessing import MinMaxScaler
import numpy as np
# 학습 데이터는 0부터 10까지, 테스트 데이터는 0부터 5까지
train_array = np.arange(0, 11).reshape(-1, 1)
test_array = np.arange(0, 6).reshape(-1, 1)train_array는 0부터 10까지의 학습 데이터이며, test_array는 0부터 5까지의 값을 가지는 테스트 데이터이다.
scaler = MinMaxScaler()
scaler.fit(train_array)
train_scaled = scaler.transform(train_array)
print('원본 train_array 데이터: ', np.round(train_array.reshape(-1), 2))
print('Sclae된 train_array 데이터: ', np.round(train_scaled.reshape(-1), 2))원본 train_array 데이터: [ 0 1 2 3 4 5 6 7 8 9 10] Sclae된 train_array 데이터: [0. 0.1 0.2 0.3 0.4 0.5 0.6 0.7 0.8 0.9 1. ]
위와 같이 학습 데이터에 fit()을 적용하고, 아래에 테스트 데이터에도 fit()을 적용해보자.
scaler.fit(test_array)
test_scaled = scaler.transform(test_array)
print('원본 test_array 데이터: ', np.round(test_array.reshape(-1), 2))
print('Scale된 test_array 데이터: ', np.round(test_scaled.reshape(-1), 2))원본 test_array 데이터: [0 1 2 3 4 5] Scale된 test_array 데이터: [0. 0.2 0.4 0.6 0.8 1. ]
출력 결과를 확인하면 학습 데이터와 테스트 데이터의 스케일링이 맞지 않음을 확인할 수 있다. 스케일링 수준이 다르기 때문에 학습 데이터와 테스트 데이터의 서로 다른 원본값이 동일한 값으로 변환되는 문제점이 발생했다. (학습 데이터의 10과 테스트 데이터의 5가 동일하게 1로 변환됐다.)
따라서 테스트 데이터에 다시 fit()을 적용해서는 안되며, 학습 데이터로 이미 fit()이 적용된 Sclaer객체를 이용해 transform()으로 변환해야 한다.
스케일링 변환 시 유의할 점을 요약하면 다음과 같다.
- 가능하다면 전체 데이터의 스케일링 변환을 적용한 뒤 학습과 테스트 데이터로 분리(전처리를 통으로 한다.)
1이 여의치 않다면 테스트 데이터 변환 시에는 학습 데이터로 이미fit()된 Scaler객체를 이용해transform()으로 변환
