타깃을 모르는 비지도 학습
비지도 학습(unsupervised learning) : 타깃 없을 때 사용하는 머신러닝 알고리즘. 사람이 가르쳐 주지 않아도 데이터 있는 무언가를 학습하는 것.
과일 사진 데이터 준비하기
# 과일 데이터 파일 읽어오기
!wget https://bit.ly/fruits_300_data -O fruits_300.npy+ !는??
'!' 문자로 시작하면 코랩은 이후 명령을 파이썬 코드가 아니라 리눅스 셀(shell) 명령으로 이해한다. wget 명령은 원격 주소에서 데이터 다운로드해 저장함.
# 파일에서 데이터 로드
# 넘파이와 맷플롯립 패키지 임포트
import numpy as np
import matplotlib.pyplot as plt
# 넘파이에서 npy 파일 로드하는 방법
# load() 매서드에 파일 이름 전달
fruits = np.load('fruits_300.npy')
# fruits 배열 크기 확인
print(fruits.shape)
> (300, 100, 100)
# 첫 번째 차원(300)은 샘플의 개수, 두 번째 차원(100)은 이미지 높이, 세 번째 차원(100)은 이미지 너비
# 첫 번째 이미지의 첫 번째 행 출력.
# 3차원 배열이기 때문에 처음 2개의 인덱스를 0으로 지정하고 마지막 인덱스는 지정하지 않거나 슬라이싱 연산자 쓰면 첫 번째 이미지의 첫 번째 행을 모두 선택할 수 있음.
print(fruits[0, 0, :])
> [ 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 1
2 2 2 2 2 2 1 1 1 1 1 1 1 1 2 3 2 1
2 1 1 1 1 2 1 3 2 1 3 1 4 1 2 5 5 5
19 148 192 117 28 1 1 2 1 4 1 1 3 1 1 1 1 1
2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 1 1 1]첫 번째 행에 있는 픽셀 100개에 들어있는 값 출력함. 이 넘파이 배열은 흑백 사진을 담고 있기 때문에 0-255까지의 정숫값 가짐.
맷플롯립의 inshow() 함수를 사용하면 넘파이 배열로 저장된 이미지를 쉽게 그릴 수 있다. 흑 백이미지이므로 camp 매개변수를 'gray'로 지정한다.
plt.imshow(fruits[0], cmap='gray')
plt.show()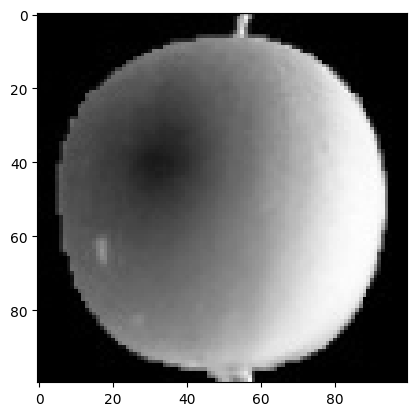
첫번째 이미지는 사과 같음! 첫 번째 행이 위에서 출력한 배열 값에 해당함. 0에 가까울수록 검게 나타나고 높은 값은 밝게 표시됨.
보통 흑백 샘플 이미지는 바탕이 밝고 물체(여기서는 사과)가 짙은 색. 그런데 왜 이렇게 보일까?? 이 흑백 이미지는 사진으로 찍은 이미지 넘파이 배열로 변환할 때 반전시킨 것.사진의 흰 바탕(높은 값)은 검은색(낮은 값)으로 만들고 실제 사과가 있어 짙은 부분(낮은 값)은 밝은색(높은 값)으로 바꾸었다. 다음 그림 참고.
왜 이렇게 바뀌었을까?? 우리의 관심 대상은 바탕이 아니라 사과! 흰색 바탕은 우리에게 중요하지 않지만 컴퓨터는 255에 가까운 바탕에 집중할 것. 따라서 바탕을 검게 만들고 사진에 짙게 나온 사과를 밝은색으로 만듦.
+ 컴퓨터는 왜 255에 가까운 바탕에 집중하는지??
알고리즘이 어떤 출력을 만들기 위해 곱셈, 덧셈을 한다. 픽셀값이 0이면 출력도 0이 되어 의미가 없음. 픽셀값이 높으면 출력값도 커지기 때문에 의미 부여하기 좋음!
우리가 보는 것과 컴퓨터가 처리하는 방식이 다르기 때문에 종종 흑백 이미지를 이렇게 반전하여 사용. 관심 대상의 영역을 높은 값으로 바꾸었지만 맷플롯립으로 출력할 때 바탕이 검게 나오므로 보기에는 썩 좋지 않음. cmap 매개변수를 'gray_r'로 지정하면 다시 반전하여 우리 눈에 보기 좋게 출력됨!
plt.imshow(fruits[0], cmap='gray_r')
plt.show()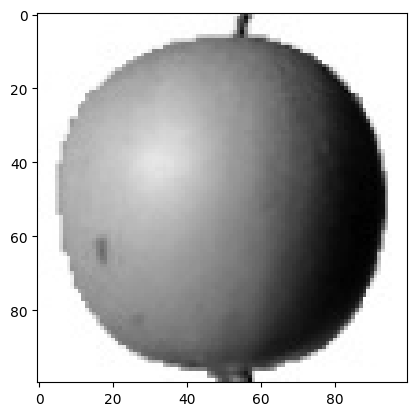
→ 밝은 부분은 0에 가깝고 짚은 부분은 255에 가깝다.
# 사과, 바나나, 파인애플이 각 100개씩 들어 있음. 나머지 과일 이미지도 출력
fig, axs = plt.subplots(1, 2)
axs[0].imshow(fruits[100], cmap='gray_r')
axs[1].imshow(fruits[200], cmap='gray_r')
plt.show()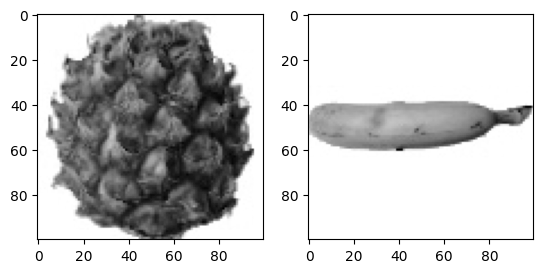
맷플롭립의 subplots() 함수 사용하면 여러 개의 그래프 배열처럼 쌓을 수 있도록 도와줌. subplots() 함수의 두 매개변수는 그래프를 쌓을 향과 열을 지정함. 여기에서는 subplots(1,2)처럼 하나의 행과 2개의 열을 지정함.
반환된 axs는 2개의 서브 그래프를 담고 있는 배열. axs[0]에 파인애플 이미지를 그리고 axs[1]에 바나나 이미지 그림.
픽셀값 분석하기
사용하기 쉽게 fruits 데이터를 사과, 파인애플, 바나나로 각각 나누어 보자. 넘파이 배열을 나눌 때 100 x 100 이미지를 펼쳐서 길이가 10,000인 1차원 배열로 만들자. 펼치면 이미지로 출력하긴 어렵지만 배열을 계산할 땐 편리하다.
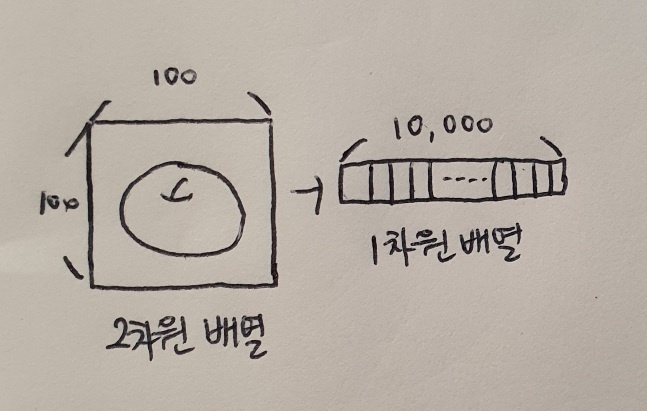
fruits 배열에서 순서대로 100개씩 선택하기 위해 슬라이싱 연산자 사용. 그 다음 reshape() 메서드 사용해 두 번째 차원(100)과 세 번째 차원(100_을 10,000으로 합침. 첫 번째 차원을 -1로 지정하면 자동으로 남은 차원을 할당함. 여기에서는 처 번째 차원이 샘플 개수.
apple = fruits[0:100].reshape(-1, 100*100)
pineapple = fruits[100:200].reshape(-1, 100*100)
banana = fruits[200:300].reshape(-1, 100*100)
# apple, pineapple, banana 배열의 크기는 (100, 10000)
# 사과 확인
print(apple.shape)
>>> (100, 10000)이제 apple, pineapple, banana 배열에 들어 잇는 샘플의 픽셀 평균값 계산해보자! 넘파이 mean() 메서드 사용. axis = 0으로 하면 첫 번째 축인 행을 따라, axis = 1로 지정하면 두 번째 축인 열을 따라 계산함.
우리가 필요한 것은 샘플의 평균값. 샘플은 모두 가로로 값을 나열했으니 axis = 1로 지정. 평균 계산하는 넘파이 np.mean() 함수 사용해도 되지만 넘파이 배열은 이런 함수들을 메서드로도 제공.
# apple 배열의 평균값.
print(apple.mean(axis=1))
>>> [ 88.3346 97.9249 87.3709 98.3703 92.8705 82.6439 94.4244 95.5999
90.681 81.6226 87.0578 95.0745 93.8416 87.017 97.5078 87.2019
88.9827 100.9158 92.7823 100.9184 104.9854 88.674 99.5643 97.2495
94.1179 92.1935 95.1671 93.3322 102.8967 94.6695 90.5285 89.0744
97.7641 97.2938 100.7564 90.5236 100.2542 85.8452 96.4615 97.1492
90.711 102.3193 87.1629 89.8751 86.7327 86.3991 95.2865 89.1709
96.8163 91.6604 96.1065 99.6829 94.9718 87.4812 89.2596 89.5268
93.799 97.3983 87.151 97.825 103.22 94.4239 83.6657 83.5159
102.8453 87.0379 91.2742 100.4848 93.8388 90.8568 97.4616 97.5022
82.446 87.1789 96.9206 90.3135 90.565 97.6538 98.0919 93.6252
87.3867 84.7073 89.1135 86.7646 88.7301 86.643 96.7323 97.2604
81.9424 87.1687 97.2066 83.4712 95.9781 91.8096 98.4086 100.7823
101.556 100.7027 91.6098 88.8976]히스토그램 그려 평균값이 어떻게 분포되어 있는지 확인. 세 과일에 대한 히스토그램 모두 겹쳐 그려보자. 이렇게 하려면 조금 투명하게 해야 겹친 부분 잘 볼 수 있음.
alpha 매개변수를 1보다 작게 하면 투명도 줄 수 있음. 또 legend() 함수 사용해 어떤 과일의 히스토그램인지 범례 만들기.
plt.hist(np.mean(apple, axis=1), alpha=0.8)
plt.hist(np.mean(pineapple, axis=1), alpha=0.8)
plt.hist(np.mean(banana, axis=1), alpha=0.8)
plt.legend(['apple', 'pineapple', 'banana'])
plt.show()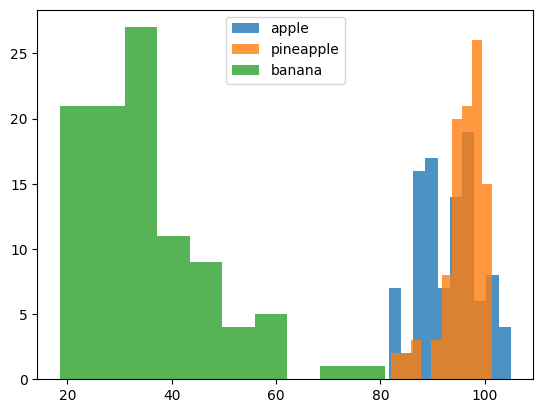
히스토그램 보면 바나나 사진의 펴균은 40 아래에 집중되어 있음. 사과와 파인애플은 90-100 사이에 많이 모여있음. 이 그림 보면 바나나는 픽셀 평균값만으로 사과나 파인애플과 확실히 구분됨. 바나나는 사진에서 차지하는 영역이 작기 때문에 평균값이 작음.
반면 사과와 파인애플은 많이 겹쳐있어 픽셀값만으로는 구분하기 쉽지 않음. 사과나 파인애플은 대체로 형태가 동그랗고 사진에서 차지하는 크기도 비슷하기 때문.
더 나은 방법은 없을까?? 샘플의 평균값이 아닌 픽셀별 평균값을 비교해보자! 전체 샘플에 대해 각 픽셀의 평균을 계산하는 것. 세 과일은 모양이 다르므로 픽셀값이 높은 위치가 조금 다를 것.
픽셀의 평균 계산하는 것도 간단. axis = 0으로 지정하면 됨! 이번에는 맷플롯립의 bar() 함수를 사용해 픽셀 10,000개에 대한 평균값 막대그래프로 그려보자.
# subplots() 함수로 34ㅐ의 서브 그래프 만들어 사과, 파인애플, 바나나에 대한 막대 그래프 그려보기.
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].bar(range(10000), np.mean(apple, axis=0))
axs[1].bar(range(10000), np.mean(pineapple, axis=0))
axs[2].bar(range(10000), np.mean(banana, axis=0))
plt.show()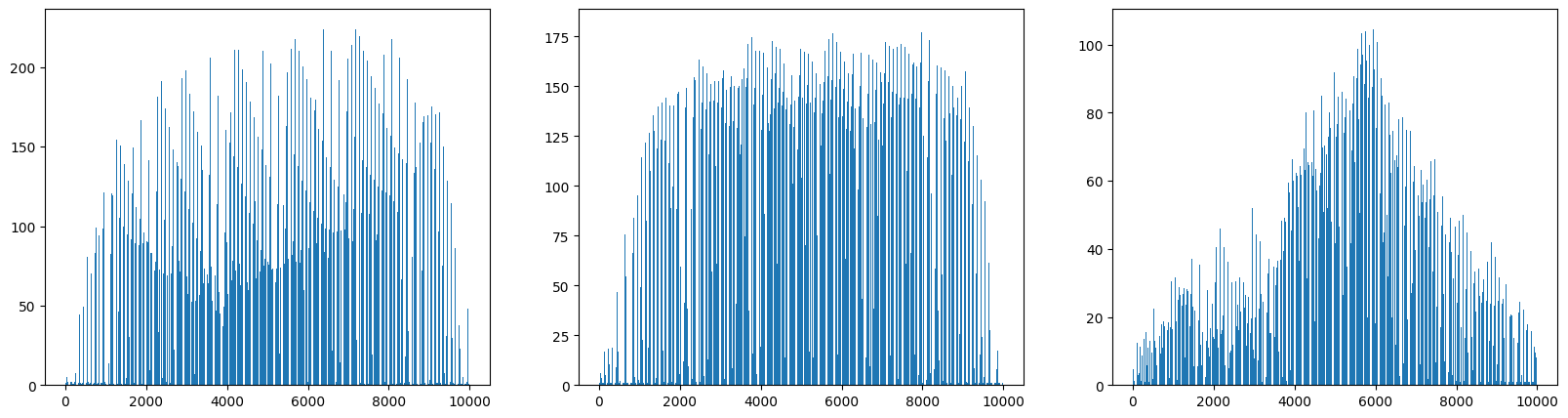
순서대로 사과, 파인애플, 바나나 그래프. 3개의 그래프 보면 과일마다 값이 높은 구간이 다르다. 사과는 사진 아래쪽으로 갈수록 값이 높아지고, 파인애플 그래프는 비교적 고르면서 높음. 바나나는 중앙의 픽셀값이 높음.
픽셀 평균값을 100 x 100 크기로 바꿔서 이미지 출력하여 위 그래프와 비교하면 더 좋다.
apple_mean = np.mean(apple, axis=0).reshape(100, 100)
pineapple_mean = np.mean(pineapple, axis=0).reshape(100, 100)
banana_mean = np.mean(banana, axis=0).reshape(100, 100)
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].imshow(apple_mean, cmap='gray_r')
axs[1].imshow(pineapple_mean, cmap='gray_r')
axs[2].imshow(banana_mean, cmap='gray_r')
plt.show()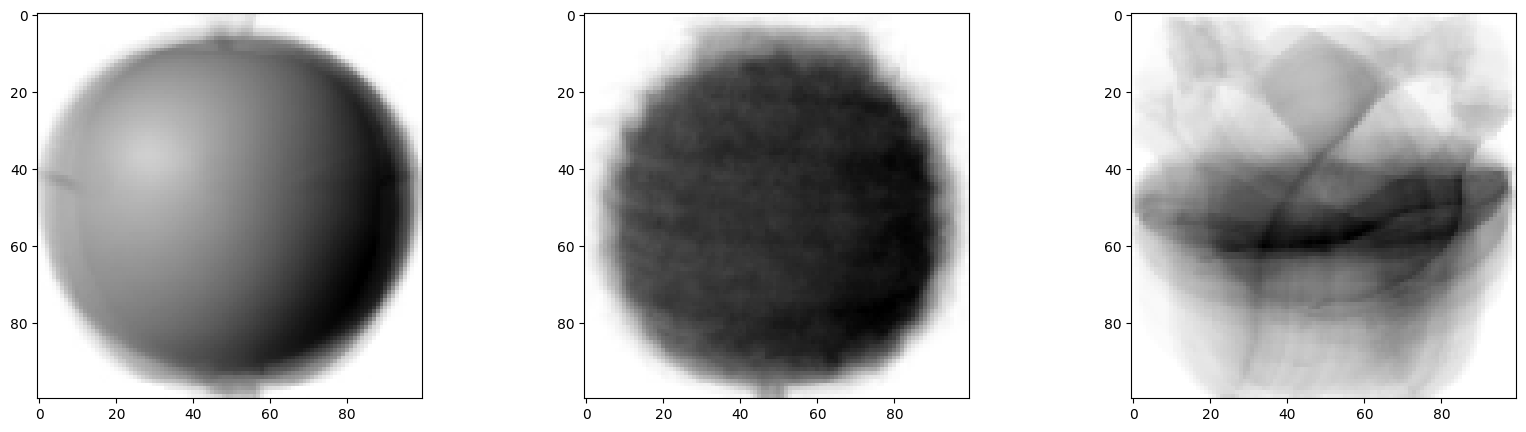
세 과일은 픽셀 위치에 따라 값의 크기가 차이남. 따라서 이 대표 이미지와 가까운 사진을 골라낸다면 사과, 파인애플, 바나나를 구분할 수 있음.
평균값과 가까운 사진 고르기
사과 사진의 평균값인 apple_mean과 가장 가까운 사진 골라보자. 3장에서 봤던 절댓값 오차를 사용. fruits 배열에 있는 모든 샘플에서 apple_mean 뺀 절댓값이 평균을 계산하면 된다.
절댓값부터 계산. 넘파이 abs() 함수는 절댓값을 계산하는 함수. 예를 들어 np.abs(-1)은 1을 반환. 배열 입력하면 모든 원소 절댓값 계산하여 입력과 동일한 크기의 배열 반환함. 이 함수는 np.absolute() 함수의 다른 이름.
다음 코드에서 abs_diff는 (300, 100, 100) 크기의 배열. 따라서 각 샘플에 대한 평균을 구하기위해 axis에 두 번째, 세 번째 차원 모두 지정. 이렇게 계산한 abs_mean은 각 샘플의 오차 평균이므로 크기가 (300,)인 1차원 배열.
abs_diff = np.abs(fruits - apple_mean)
abs_mean = np.mean(abs_diff, axis=(1,2))
print(abs_mean.shape)
>>> (300,)
# 그 다음 값이 가장 작은 수서대로 100개 골라보기. 즉 apple_mean과 오차가 가장 작은 ㅅ매플 100개 고르는 셈.
# np.argsort() 함수는 작은 것에서 큰 순서대로 나열한 abs_mean 배열의 인덱스 반환.
# 이 인덱스 중에서 처음 100개 선택해 10 x 10 격자로 이루어진 그래프 그려보자.
apple_index = np.argsort(abs_mean)[:100]
fig, axs = plt.subplots(10, 10, figsize=(10,10))
for i in range(10):
for j in range(10):
axs[i, j].imshow(fruits[apple_index[i*10 + j]], cmap='gray_r')
axs[i, j].axis('off')
plt.show()
apple_mean과 가장 가까운 사진 100개 골랐더니 모두 사과!
먼저 subplots() 함수로 10 x 10, 총 100개의 서브 그래프를 만든다. 그래프가 많기 땜누에 전체 그래프의 크기를 figsize = (10, 10)으로 조금 크게 지정.
그 다음 2중 for 반복문을 순화하면서 10개의 행과 열에 이미지를 출력. axs는 (10, 10) 크기의 2차원 배열이므로 i, j 두 첨자를 사용하여 서브 그래프 위치를 지정. 또 깔끔하게 이미지만 그리기 위해 axis('off') 사용해 좌표축 그리지 않음.
흑백 사진에 있는 픽셀값을 사용해 과일 사진을 모으는 작업 해보기. 이렇게 비슷한 샘플끼리 그룹으로 모으는 작업을 군집(clustering)이라고 한다. 군집은 대표적인 비지도 학습 작업 중 하나. 군집 알고리즘에서 만든 그룹을 클러스터(cluster)라고 부른다.
하지만 우리는 이미 사과, 파인애플, 바나나가 있다는 점 알고 있음. 즉 타깃값을 알고 있었기 때문에 사과, 파인애플, 바나나의 사진 평균값을 계산해서 가장 가까운 과일을 찾을 수 있음. 실제 비지도 학습에서는 타깃값을 모르기 때문에 이처럼 샘플의 평균값 미리 구할 수 있음.
전체 코드 (출처 : https://bit.ly/hg-06-1)
!wget https://bit.ly/fruits_300_data -O fruits_300.npy
import numpy as np
import matplotlib.pyplot as plt
fruits = np.load('fruits_300.npy')
print(fruits.shape)
print(fruits[0, 0, :])
plt.imshow(fruits[0], cmap='gray')
plt.show()
plt.imshow(fruits[0], cmap='gray_r')
plt.show()
fig, axs = plt.subplots(1, 2)
axs[0].imshow(fruits[100], cmap='gray_r')
axs[1].imshow(fruits[200], cmap='gray_r')
plt.show()
apple = fruits[0:100].reshape(-1, 100*100)
pineapple = fruits[100:200].reshape(-1, 100*100)
banana = fruits[200:300].reshape(-1, 100*100)
print(apple.shape)
print(apple.mean(axis=1))
plt.hist(np.mean(apple, axis=1), alpha=0.8)
plt.hist(np.mean(pineapple, axis=1), alpha=0.8)
plt.hist(np.mean(banana, axis=1), alpha=0.8)
plt.legend(['apple', 'pineapple', 'banana'])
plt.show()
fig, axs = plt.subplots(1, 3, figsize=(20, 5))
axs[0].bar(range(10000), np.mean(apple, axis=0))
axs[1].bar(range(10000), np.mean(pineapple, axis=0))
axs[2].bar(range(10000), np.mean(banana, axis=0))
plt.show()
abs_diff = np.abs(fruits - apple_mean)
abs_mean = np.mean(abs_diff, axis=(1,2))
print(abs_mean.shape)
apple_index = np.argsort(abs_mean)[:100]
fig, axs = plt.subplots(10, 10, figsize=(10,10))
for i in range(10):
for j in range(10):
axs[i, j].imshow(fruits[apple_index[i*10 + j]], cmap='gray_r')
axs[i, j].axis('off')
plt.show()정리
- 비지도 학습은 머신러닝의 한 종류로 훈련 데이터에 타깃이 없음. 타깃이 없기 때문에 외부의 도움 없이 스스로 유용한 무언가를 학습해야 한다. 대표적인 비지도 학습 작업은 군집, 차원 축소 등.
- 히스토그램은 구간별로 값이 발생한 빈도를 그래프로 표시한 것. 보통 x축이 값의 구간(계급)이고 y축은 발생 빈도(도수) 이다.
- 군집은 비슷한 샘플끼리 하나의 그룹으로 모으는 대표적인 비지도 학습 작업. 군집 알고리즘으로 모은 샘플 그룹을 클러스터라고 부른다.