boosting algorithm..!
부스팅(Boosting)은 머신러닝에서 사용되는 앙상블 학습 기법 중 하나로, 약한 학습기(weak learner)를 결합하여 강력한 학습기(strong learner)를 만드는 방법입니다. 부스팅 알고리즘은 성능이 상대적으로 낮은 여러 모델을 순차적으로 학습하고, 이전 모델에서 발생한 오류에 가중치를 부여하여 새로운 모델을 향상시킵니다.
몇 가지 종류에 대해서 설명해 보겠습니다.
1. AdaBoost (Adaptive Boosting):
오분류된 데이터에 가중치를 높여 새로운 분류기를 학습하는 방식입니다. 각 분류기는 이전 분류기의 오류를 보완하는 방식으로 학습됩니다.
2. Gradient Boosting:
잔차(residual)에 대해 새로운 모델을 학습하는 방식입니다. 이전 모델의 예측과 실제 값의 차이에 대한 그래디언트(기울기)를 이용하여 새로운 모델을 학습합니다. 대표적인 구현으로는 XGBoost, LightGBM, CatBoost 등이 있습니다.
3. XGBoost (Extreme Gradient Boosting):
Gradient Boosting의 개선된 버전으로, 효율적인 트리 학습 알고리즘, 정규화 및 병렬 처리 기능 등을 제공합니다. XGBoost는 분류 및 회귀 문제에서 많이 사용됩니다.
4. LightGBM:
마이크로소프트에서 개발한 Gradient Boosting 프레임워크로, 대용량 데이터셋에 대해 효과적으로 처리할 수 있도록 설계되었습니다. Leaf-wise 트리 성장 방식을 사용하여 빠른 속도와 낮은 메모리 사용량을 제공합니다.
5. CatBoost:
Yandex에서 개발한 Gradient Boosting 알고리즘으로, 범주형 데이터 처리에 특화되어 있습니다. 자동적인 범주형 특성 처리 및 안정적인 성능을 제공합니다.
부스팅 알고리즘은 약한 학습기를 순차적으로 학습하고 각각의 오류를 보완하여 강력한 모델을 만들어내므로, 과적합에 강하고 높은 성능을 내는 경우가 많습니다.
[와인데이터 사용]
1. 데이터 불러오기
import pandas as pd
wine_url = '수업내용'
wine = pd.read_csv(wine_url, index_col=0)
wine.head()| fixed acidity | volatile acidity | citric acid | residual sugar | chlorides | free sulfur dioxide | total sulfur dioxide | density | pH | sulphates | alcohol | quality | color | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 | 1 |
| 1 | 7.8 | 0.88 | 0.00 | 2.6 | 0.098 | 25.0 | 67.0 | 0.9968 | 3.20 | 0.68 | 9.8 | 5 | 1 |
| 2 | 7.8 | 0.76 | 0.04 | 2.3 | 0.092 | 15.0 | 54.0 | 0.9970 | 3.26 | 0.65 | 9.8 | 5 | 1 |
| 3 | 11.2 | 0.28 | 0.56 | 1.9 | 0.075 | 17.0 | 60.0 | 0.9980 | 3.16 | 0.58 | 9.8 | 6 | 1 |
| 4 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.9978 | 3.51 | 0.56 | 9.4 | 5 | 1 |
2. taste 범주형 데이터로 변경하기
wine['taste'] = [1. if grade > 5 else 0. for grade in wine['quality']]
X = wine.drop(['taste', 'quality'], axis=1)
y = wine['taste']3. 표준화 시키기
from sklearn.preprocessing import StandardScaler
sc = StandardScaler() # 표준화 스케일러
X_sc = sc.fit_transform(X) # 표준화된 데이터4. 데이터 나누기
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_sc, y, test_size=0.2, random_state=13)
5. 와인데이터 히스토그램 그리기
import matplotlib.pyplot as plt
%matplotlib inline
wine.hist(bins=10, figsize=(15,10));히스토그램에서 "bins(빈)"은 데이터를 나누는 구간을 의미합니다. 히스토그램은 데이터의 분포를 시각적으로 표현하는 그래픽 방법 중 하나이며, 주로 연속된 데이터의 빈도 분포를 보여줍니다.
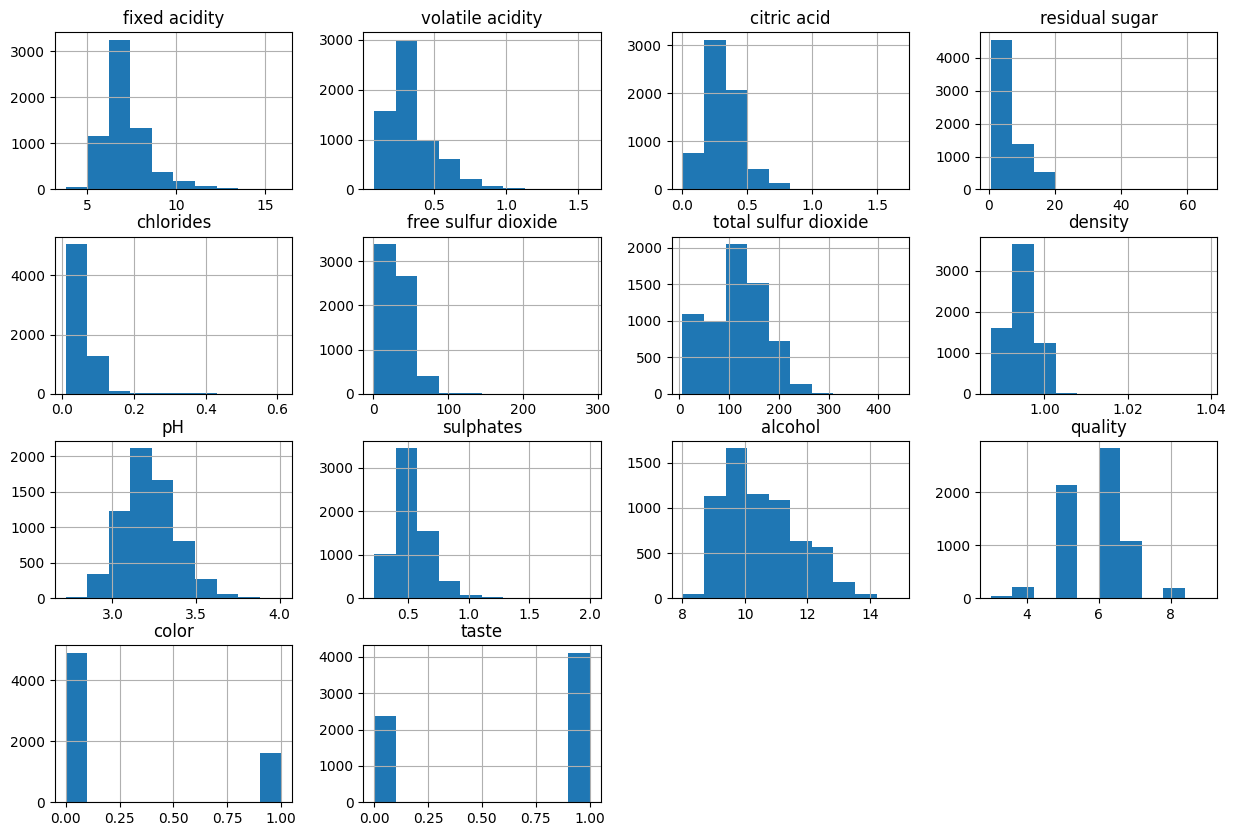
6. 와인 컬럼 확인
wine.columnsIndex(['fixed acidity', 'volatile acidity', 'citric acid', 'residual sugar',
'chlorides', 'free sulfur dioxide', 'total sulfur dioxide', 'density',
'pH', 'sulphates', 'alcohol', 'quality', 'color', 'taste'],
dtype='object')
7. 와인 컬럼으로 피벗테이블 만들기 (인덱스는 퀄리티)
column_names = ['fixed acidity', 'volatile acidity', 'citric acid', 'residual sugar',
'chlorides', 'free sulfur dioxide', 'total sulfur dioxide', 'density',
'pH', 'sulphates', 'alcohol']
df_pivot_table = wine.pivot_table(column_names, ['quality'], aggfunc='median')
df_pivot_table피벗 테이블에서 aggfunc은 "aggregation function(집계 함수)"을 나타냅니다. aggfunc은 데이터를 그룹화하고 집계하는 데 사용되는 함수를 지정하는 매개변수입니다. 피벗 테이블은 데이터를 다양한 축에 따라 정리하고, 그룹화된 데이터에 대해 특정 집계 함수를 적용하여 새로운 형태의 테이블을 생성하는 데 사용됩니다.
피벗 테이블에서 aggfunc으로 사용되는 일반적인 집계 함수에는 다음과 같은 것들이 있습니다:
Sum (np.sum 또는 sum):
그룹화된 데이터의 합을 계산합니다.
Mean (np.mean 또는 mean):
그룹화된 데이터의 평균을 계산합니다.
Count (np.count_nonzero 또는 count):
그룹화된 데이터의 개수를 계산합니다.
Max (np.max 또는 max):
그룹화된 데이터 중 최댓값을 계산합니다.
Min (np.min 또는 min):
그룹화된 데이터 중 최솟값을 계산합니다.
Median (np.median 또는 median):
그룹화된 데이터의 중앙값을 계산합니다.
Standard Deviation (np.std 또는 std):
그룹화된 데이터의 표준 편차를 계산합니다.
Frequency (lambda x: len(x) / len(x.dropna()) 또는 count와 len을 조합):
그룹화된 데이터의 빈도를 계산합니다.
| alcohol | chlorides | citric acid | density | fixed acidity | free sulfur dioxide | pH | residual sugar | sulphates | total sulfur dioxide | volatile acidity | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| quality | |||||||||||
| 3 | 10.15 | 0.0550 | 0.33 | 0.995900 | 7.45 | 17.0 | 3.245 | 3.15 | 0.505 | 102.5 | 0.415 |
| 4 | 10.00 | 0.0505 | 0.26 | 0.994995 | 7.00 | 15.0 | 3.220 | 2.20 | 0.485 | 102.0 | 0.380 |
| 5 | 9.60 | 0.0530 | 0.30 | 0.996100 | 7.10 | 27.0 | 3.190 | 3.00 | 0.500 | 127.0 | 0.330 |
| 6 | 10.50 | 0.0460 | 0.31 | 0.994700 | 6.90 | 29.0 | 3.210 | 3.10 | 0.510 | 117.0 | 0.270 |
| 7 | 11.40 | 0.0390 | 0.32 | 0.992400 | 6.90 | 30.0 | 3.220 | 2.80 | 0.520 | 114.0 | 0.270 |
| 8 | 12.00 | 0.0370 | 0.32 | 0.991890 | 6.80 | 34.0 | 3.230 | 4.10 | 0.480 | 118.0 | 0.280 |
| 9 | 12.50 | 0.0310 | 0.36 | 0.990300 | 7.10 | 28.0 | 3.280 | 2.20 | 0.460 | 119.0 | 0.270 |
8. 상관계수 확인하기
corr_matrix = wine.corr()corr_matrix| fixed acidity | volatile acidity | citric acid | residual sugar | chlorides | free sulfur dioxide | total sulfur dioxide | density | pH | sulphates | alcohol | quality | color | taste | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| fixed acidity | 1.000000 | 0.219008 | 0.324436 | -0.111981 | 0.298195 | -0.282735 | -0.329054 | 0.458910 | -0.252700 | 0.299568 | -0.095452 | -0.076743 | 0.486740 | -0.067354 |
| volatile acidity | 0.219008 | 1.000000 | -0.377981 | -0.196011 | 0.377124 | -0.352557 | -0.414476 | 0.271296 | 0.261454 | 0.225984 | -0.037640 | -0.265699 | 0.653036 | -0.267046 |
| citric acid | 0.324436 | -0.377981 | 1.000000 | 0.142451 | 0.038998 | 0.133126 | 0.195242 | 0.096154 | -0.329808 | 0.056197 | -0.010493 | 0.085532 | -0.187397 | 0.075739 |
| residual sugar | -0.111981 | -0.196011 | 0.142451 | 1.000000 | -0.128940 | 0.402871 | 0.495482 | 0.552517 | -0.267320 | -0.185927 | -0.359415 | -0.036980 | -0.348821 | -0.032484 |
| chlorides | 0.298195 | 0.377124 | 0.038998 | -0.128940 | 1.000000 | -0.195045 | -0.279630 | 0.362615 | 0.044708 | 0.395593 | -0.256916 | -0.200666 | 0.512678 | -0.181908 |
| free sulfur dioxide | -0.282735 | -0.352557 | 0.133126 | 0.402871 | -0.195045 | 1.000000 | 0.720934 | 0.025717 | -0.145854 | -0.188457 | -0.179838 | 0.055463 | -0.471644 | 0.044819 |
| total sulfur dioxide | -0.329054 | -0.414476 | 0.195242 | 0.495482 | -0.279630 | 0.720934 | 1.000000 | 0.032395 | -0.238413 | -0.275727 | -0.265740 | -0.041385 | -0.700357 | -0.047585 |
| density | 0.458910 | 0.271296 | 0.096154 | 0.552517 | 0.362615 | 0.025717 | 0.032395 | 1.000000 | 0.011686 | 0.259478 | -0.686745 | -0.305858 | 0.390645 | -0.268876 |
| pH | -0.252700 | 0.261454 | -0.329808 | -0.267320 | 0.044708 | -0.145854 | -0.238413 | 0.011686 | 1.000000 | 0.192123 | 0.121248 | 0.019506 | 0.329129 | 0.018842 |
| sulphates | 0.299568 | 0.225984 | 0.056197 | -0.185927 | 0.395593 | -0.188457 | -0.275727 | 0.259478 | 0.192123 | 1.000000 | -0.003029 | 0.038485 | 0.487218 | 0.035807 |
| alcohol | -0.095452 | -0.037640 | -0.010493 | -0.359415 | -0.256916 | -0.179838 | -0.265740 | -0.686745 | 0.121248 | -0.003029 | 1.000000 | 0.444319 | -0.032970 | 0.394676 |
| quality | -0.076743 | -0.265699 | 0.085532 | -0.036980 | -0.200666 | 0.055463 | -0.041385 | -0.305858 | 0.019506 | 0.038485 | 0.444319 | 1.000000 | -0.119323 | 0.814484 |
| color | 0.486740 | 0.653036 | -0.187397 | -0.348821 | 0.512678 | -0.471644 | -0.700357 | 0.390645 | 0.329129 | 0.487218 | -0.032970 | -0.119323 | 1.000000 | -0.116595 |
| taste | -0.067354 | -0.267046 | 0.075739 | -0.032484 | -0.181908 | 0.044819 | -0.047585 | -0.268876 | 0.018842 | 0.035807 | 0.394676 | 0.814484 | -0.116595 | 1.000000 |
['quality'] 상관계수 내림차순하기
corr_matrix['quality'].sort_values(ascending=False)quality 1.000000
taste 0.814484
alcohol 0.444319
citric acid 0.085532
free sulfur dioxide 0.055463
sulphates 0.038485
pH 0.019506
residual sugar -0.036980
total sulfur dioxide -0.041385
fixed acidity -0.076743
color -0.119323
chlorides -0.200666
volatile acidity -0.265699
density -0.305858
Name: quality, dtype: float64
9. taste 변수 카운트플랏
import seaborn as sns
sns.countplot(wine['taste'])
plt.show()
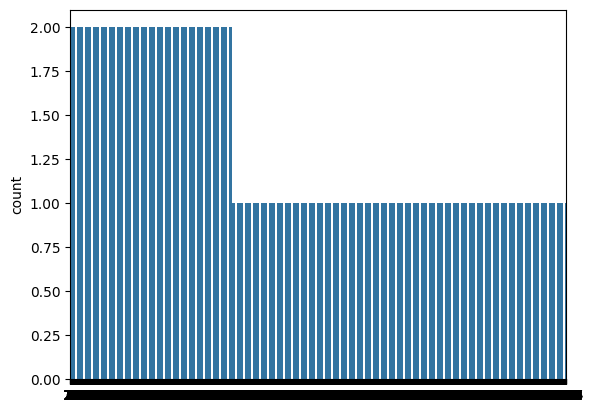
10. 모델 한 번에 정리하기
from sklearn.ensemble import (AdaBoostClassifier, GradientBoostingClassifier, RandomForestClassifier)
from sklearn.tree import DecisionTreeClassifier
from sklearn.linear_model import LogisticRegression
models = []
models.append(('RandomForest:' , RandomForestClassifier()))
models.append(('AdaBoost: ', AdaBoostClassifier()))
models.append(('GradientBoosting: ', GradientBoostingClassifier()))
models.append(('DecisionTree: ', DecisionTreeClassifier()))
models.append(('LogisticRegression: ', LogisticRegression()))
11. 결과를 저장하기 위한 작업
%%time
from sklearn.model_selection import KFold, cross_val_score
results = []
names = []
for name, model in models: # name: 모델 이름, model: 모델 객체 ex) name : RandomForestClassifier , model : RandomForestClassifier()
kfold = KFold(n_splits=5, random_state=13, shuffle=True)
cv_result = cross_val_score(model, X_train, y_train, cv=kfold,
scoring='accuracy')
results.append(cv_result)
names.append(name)
print(name, cv_result.mean(), cv_result.std()) # 모델 이름, 평균 정확도, 표준편차 출력
RandomForest: 0.8216215665950989 0.019082935401416767
AdaBoost: 0.7533103205745169 0.02644765901536818
GradientBoosting: 0.7663959428444511 0.021596556352125432
DecisionTree: 0.7535096616569186 0.006582230062181481
LogisticRegression: 0.74273191678389 0.015548839626296565
CPU times: user 12.5 s, sys: 522 ms, total: 13 s
Wall time: 20.8 s
12. cross-validation 결과를 일목요연하게 확인하기
flg = plt.figure(figsize=(14,8))
flg.suptitle('Algorithm Comparison') # 그래프 제목
ax = flg.add_subplot(111) # 그래프의 위치
plt.boxplot(results) # 결과를 박스플롯으로 출력
ax.set_xticklabels(names) # x축에 모델 이름 출력
plt.show()# 그래프 출력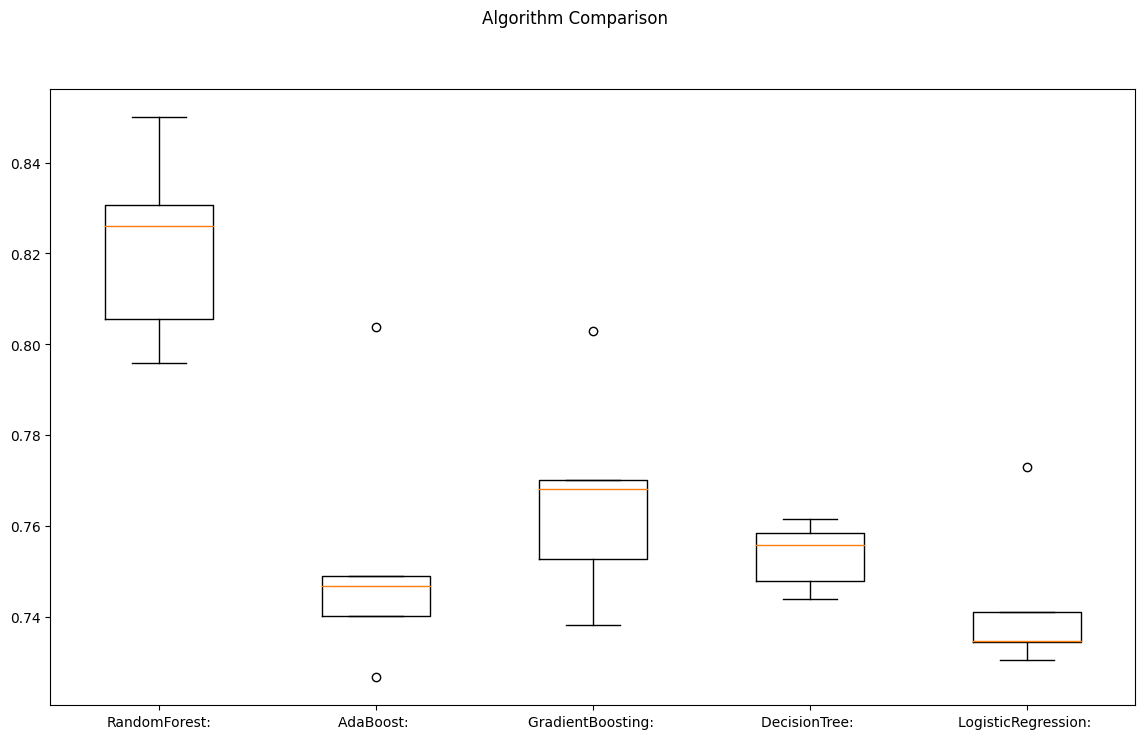
13. 테스트 데이터에도 모델 적용하여 정확도 측정
from sklearn.metrics import accuracy_score
for name, model in models:
model.fit(X_train, y_train)
pred = model.predict(X_test)
print(name, accuracy_score(y_test, pred))
RandomForest: 0.8315384615384616
AdaBoost: 0.7553846153846154
GradientBoosting: 0.7876923076923077
DecisionTree: 0.7792307692307693
LogisticRegression: 0.7469230769230769
14. 결론
테스트 데이터에서도 랜덤포레스트가 가장 좋은 성능을 내고 있다.
