Molecular Representation이란? : The language of chmistry is the 2D structure diagram
하지만, 컴퓨터가 읽고 이해하고, 저장하고 교환할 때 2D 구조이기 때문에 쉽지 않음
따라서, One Code ↔ One Structure
In the idal representation, there is only one 'code' for a given structure and any one code can be interpreteed to give only one structure.
This unique representation(one code, one structure) is essential for chemical registration systems in which the novelty of a structure is determined before it is recoreded in a database
💡 들어가기 전에 ! 화학구조 표현의 기본적인 사항들을 짚고 넘어가야한다-
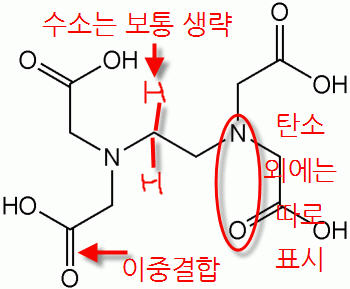
보통 분자 구조식은 유기화합물을 나타내는데 주로 사용된다.
-
따라서 탄소가 주요 구성요소이기 때문에 구조식을 그릴때도 탄소를 의미하는 "C"는 생략해주며, 구조식에서 별다른 표시 없이 꺾인 꼭지점은 탄소가 있는 부분이라 생각하면 된다.
-
결합 수? : 탄소-4, 질소-3, 산소-2 ⇒ 원자들은 할당된 결합 수를 모두 사용해야 하며, 생략된 결합(-)은 수소를 나타낸다.
Molecular structures representation method
- Linear notation : 한 줄에 표현하는 것,
- SMILES
- InChI, InChIKey
- Connection table method : 2D 방법, 하나의 file로 저장
- Molfile
- SDF
- MOL2
*ChEMBL : Biological active compound에 대한 DB 사이트 → 분자 구조에 대한 SMIELS, InChI, molefile 등과 같은 표현 방식을 제공한다.
*PubChem : 모든 화합물에 대해 deposit되어있는 사이트
Linear Notation
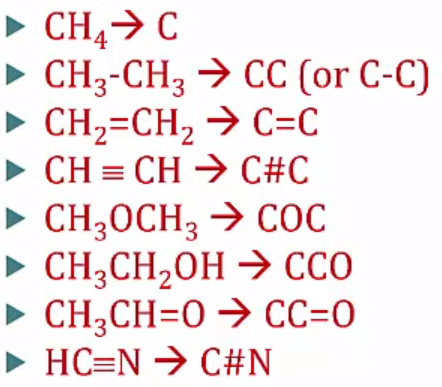
Linear notation represent structure as a linear string of alphanumeric symbols.
Cheminfomratics 초기에는 과거 저장공간이 크지 않았기 때문에 이 방법이 compactness하여 효율적이라는 장점이 있었다.
현재까지도, 이 방법은 빠르기 때문에 자주 사용된다. 특히 SMILES
Even nowadays, it can be faster to enter a structure as a notation instead of using a chemical structure drawing program
SMILES (Simplified Molecular Input Line Entry System)
주어진 chemical structure 하나에 다양한 수의 SMILES 코드를 만들어 낼 수 있다.
- Not unique representation
- have many valid and unambiguous representations
*canonical SMILES : unique representation
- Atoms : 원자 표현, 하나의 대문자 알파벳
- non-hydrogen atom is in square brackets [] : [Au], or [Fe]
- 유기화합물 (Organic~)은 보통 브라켓 생략함 (B,C,N,O,P,S,F,Cl,Br,I)
- If the proper number of "implicit" hydrogen atoms is assumed: BH_3 → B, CH_4→C,,,
- Bonds : 결합
-
Single bond → "-" (can be omitted)
-
Doungle bond → "="
-
Triple bond → "#"
-
Aromatic bond → ":" (can be omitted)
[Example]
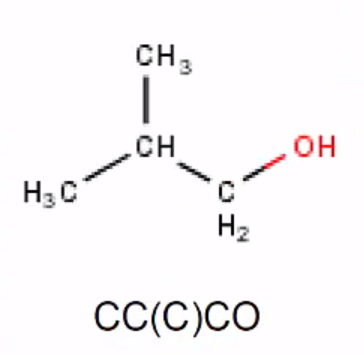
-
- Branches : 괄호로 표현
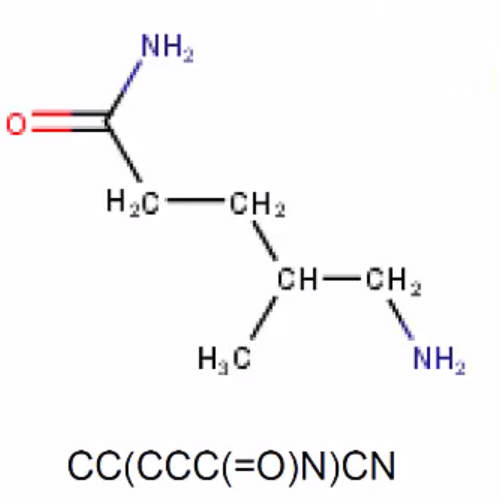
- Rings represented by breaking one single or aromitc bond in ehac rihg, designating this ring-closure point witha d digit *Benzene (aromitic)링과 Cyclohexane 링 표현 법이 약간 다르다.
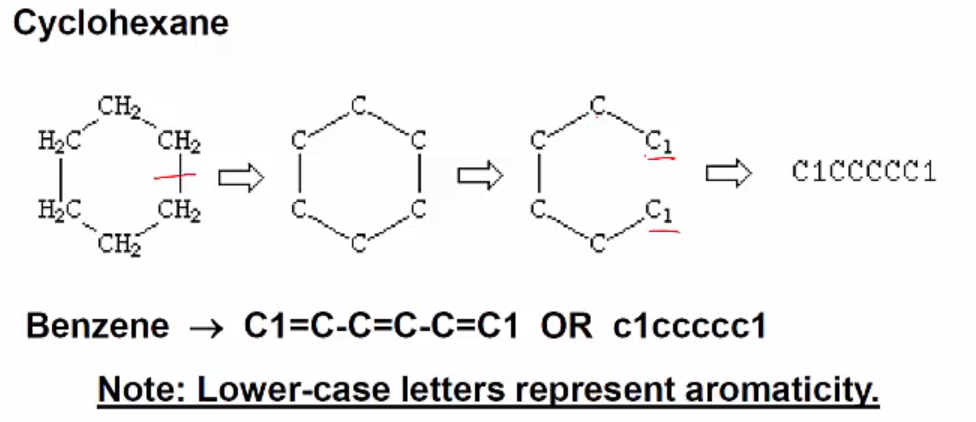
Canonical SMIELS
One "canonical" SMILES is selected among them: Morgan algorithm (어떤 부분을 첫번째 원소로 쓸 지 선택하는 알고리즘)
-
Morgan Algorithm
-
각 원소에 invariant값인 1을 할당하고, 연결된 neighboring value를 총합하여 값을 결정한다.
*#values : 서로 다른 숫자의 갯수
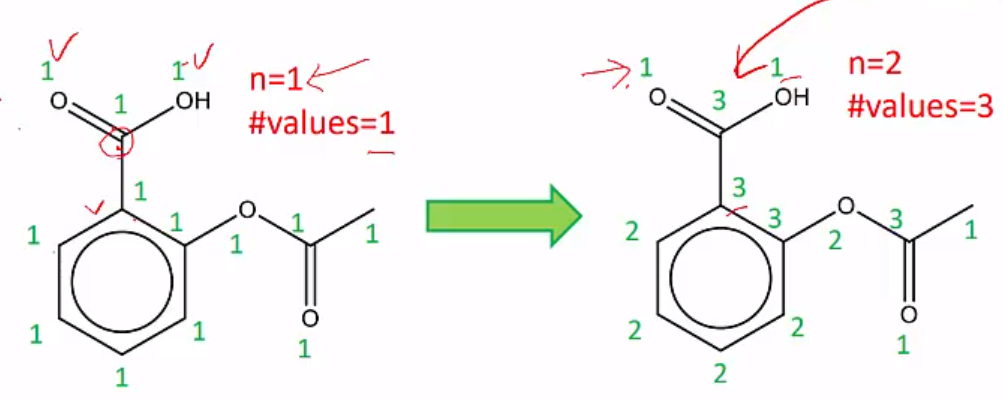
-
repreat the suming of neighboring values
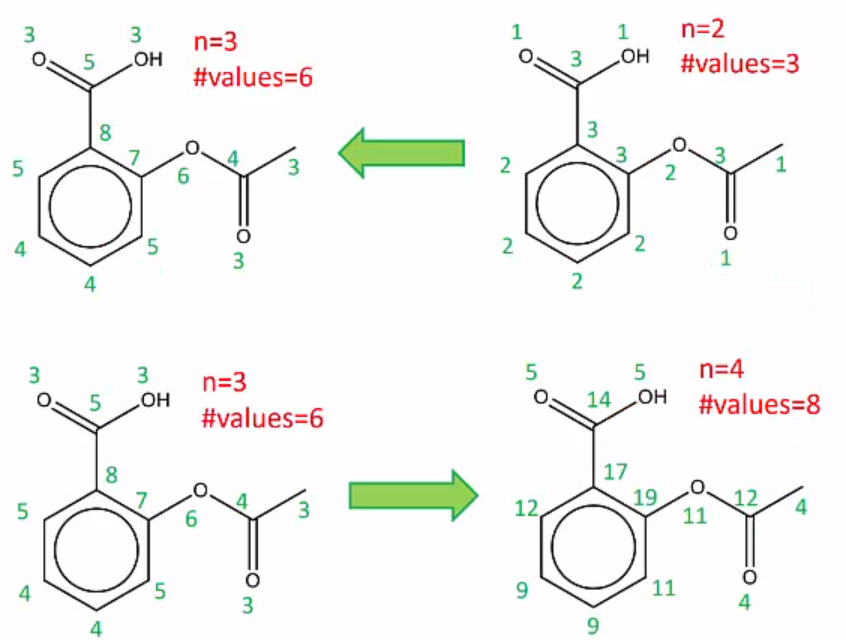
-
Until number of values does not increase anymore
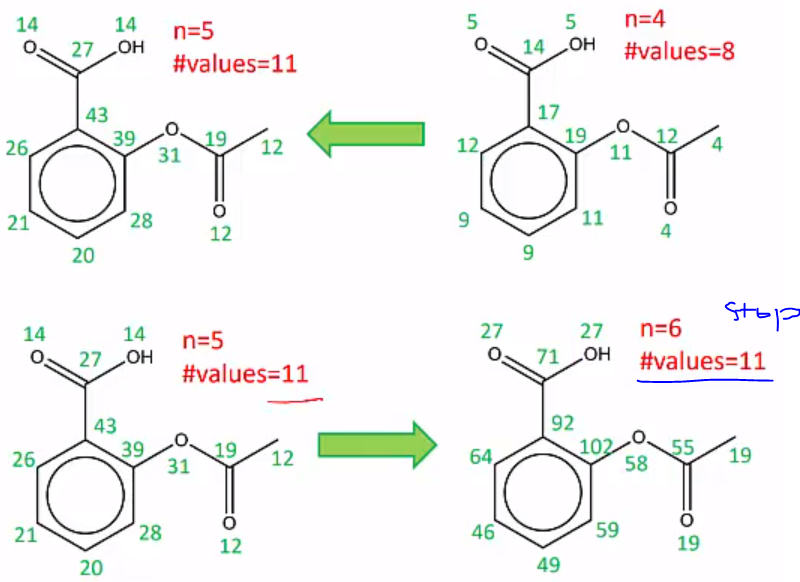
-
Assign priorites according to invariants
Disambiguate ties by atom type and bond order (single bond의 우선순위가 더 높다 등등,, rule에 따라)
Construct Smiles according to invariants
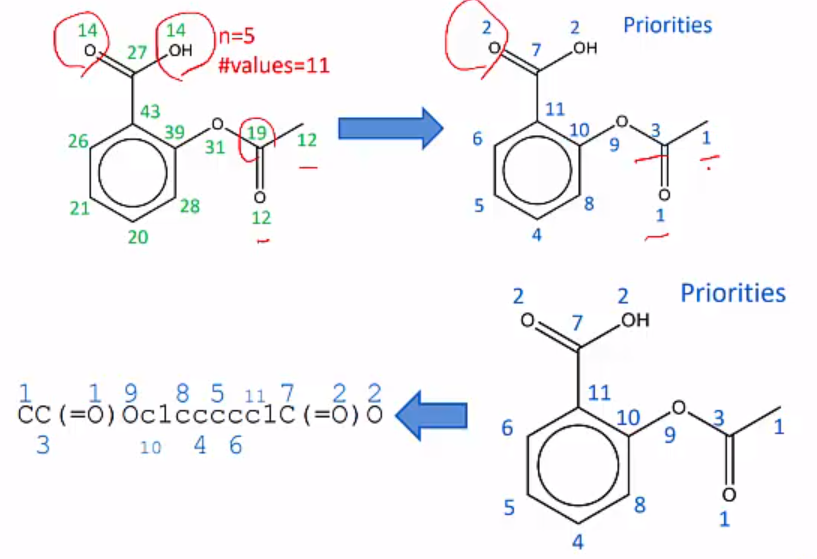
-
Isomeric SMILES
Isotope: the integral atomic mass preceding the atomic symbol: → [13CH4]
*통상적이지 않는 Isotope를 갖는 경우를 표현함
보통 Carbon (C)는 12가 일반적이지만, Carbon13를 가지고 있을 경우 C앞에 13을 기재
Stereochemistry : 입체 화학, 분자의 3차원적인 구조 또는 그를 밝히는 방법론
Atom stereo center (분자 입체중심)
@@ - L, @ - D

- Limitation of SMILES
- Most SMILES encoders/decoders are proprietary
- 회사마다 약간씩 다른 impletation
- Not interchable between databased unless the smae software is used
- Doesn;t have 2D, 3D coordinates retained, so need to changes to other format like MOL, SDF, ...
- Multiple SMILES for one compound
- Most SMILES encoders/decoders are proprietary
InCHI (Interantional Chemical Identifier)
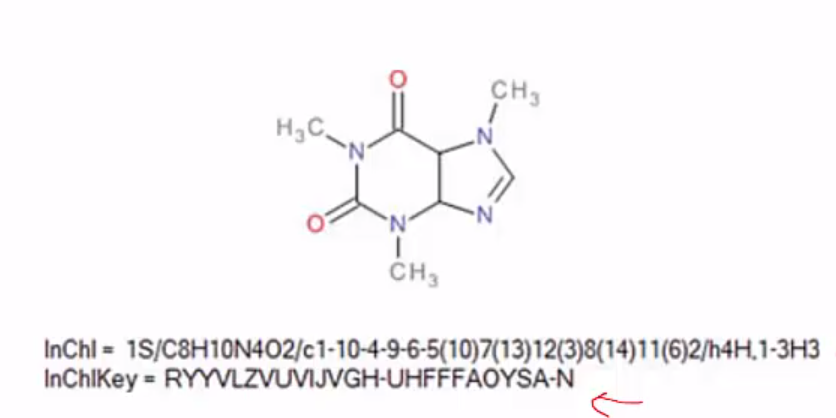
⇒ 표현식이 너무 길고 복잡하다 & 인터넷 search에 적합하지 않다.
**InChIKey
: 인터넷 search/db indexing에 적합하도록 만든 것
Connection Table
: The MDL (now Symyx) connection table, has become the in fact standard for exchange of datasets.
It seperates atoms and bonds into separate blocks.
A molecule file, or 'molefile,' describes a single modelcular structure that can contain disjoint fragments
A more file consists of a header block and connection table.
Structure-data files (SDFiles) contain structures and data for any number of molecules
> 하나의 분자를 표현할 땐 molefile, 여러개의 분자를 표현할 때는 SDFile
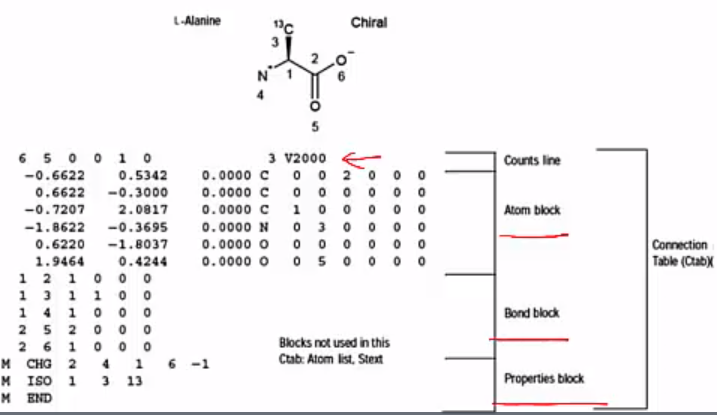
출처 : LAIDD 강의
