1. 와인데이터분석
(1) 데이터 불러오기
import pandas as pd
red_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-red.csv'
white_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-white.csv'
red_wine = pd.read_csv(red_url, sep=';')
white_wine = pd.read_csv(white_url, sep=';')
(2) 컬럼확인
(3) 데이터합치기
- 레드와인과 화이트 와인 구분을 위한 color 컬럼추가
red_wine['color'] = 1.
white_wine['color'] = 0.- 합치기
wine = pd.concat([red_wine,white_wine])
wine.info()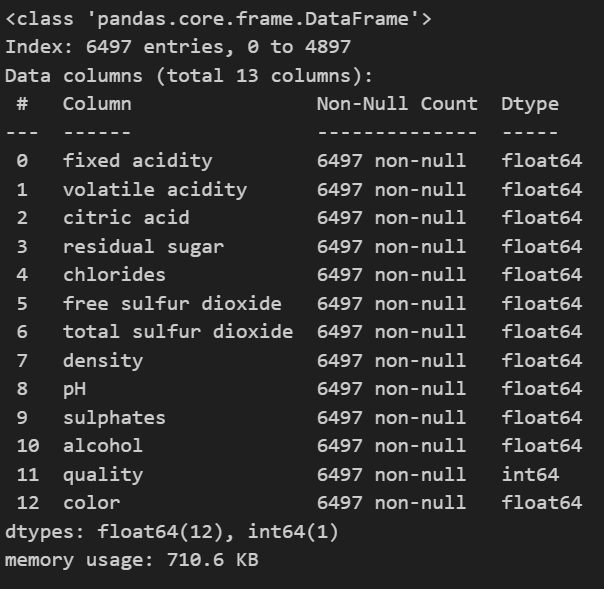
(4) histogram
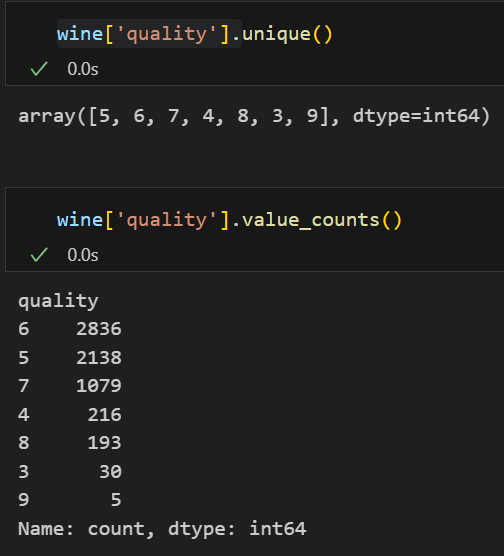
- 등급별 와인 수
import plotly.express as px
fig = px.histogram(wine, x='quality')
fig.show()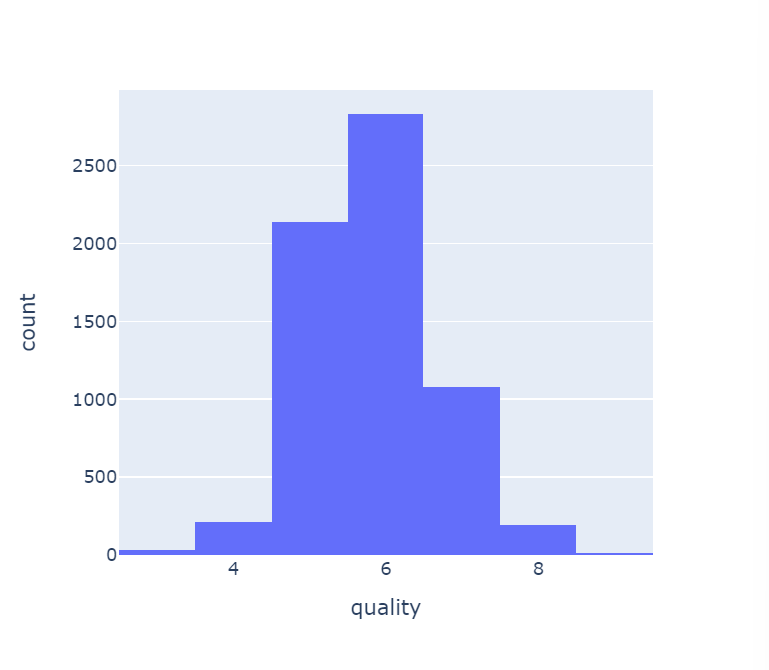
- 와인color별 등급별 수
fig = px.histogram(wine, x='quality', color = 'color')
fig.show()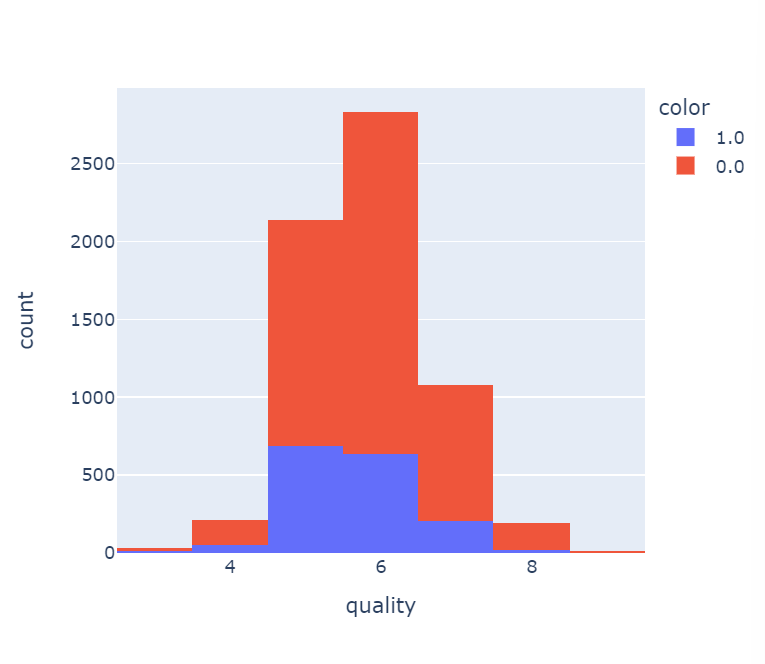
(5) red/white 분류 - DecisionTree
- 특성데이터, 타겟 분리
X = wine.drop(['color'], axis=1)
y = wine['color']X : 특성데이터 / y : 타겟
- 훈련 / 테스트 데이터 분리
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.2, random_state=2
)- 분리데이터 확인
import numpy as np
np.unique(y_train, return_counts=True)➡️ (array([0., 1.]), array([3930, 1267], dtype=int64))
📌 0(화이트) 3930개, 1(레드) 1267개
import plotly.graph_objects as go
fig = go.Figure()
fig.add_trace(go.Histogram(x=X_train['quality'], name = 'Train'))
fig.add_trace(go.Histogram(x=X_test['quality'], name = 'Test'))
fig.update_layout(barmode = 'overlay') #겹쳐보기
fig.update_traces(opacity=0.75) #투명도
fig.show()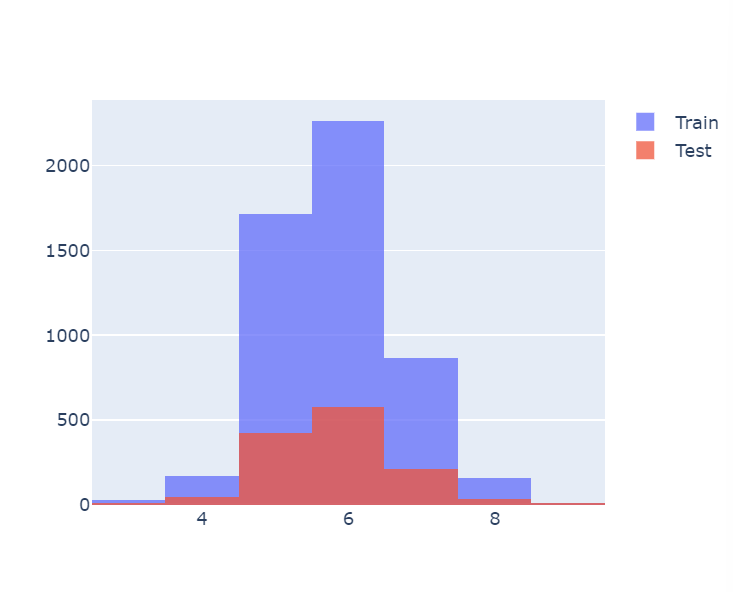
- 학습
from sklearn.tree import DecisionTreeClassifier
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=2)
wine_tree.fit(X_train,y_train)- train, test accuracy 확인
from sklearn.metrics import accuracy_score
# train 정확도
y_pred_tr = wine_tree.predict(X_train)
# test 정확도
y_pred_test = wine_tree.predict(X_test)accuracy_score(y_train,y_pred_tr)➡️ 0.9667115643640561
accuracy_score(y_test,y_pred_test)➡️ 0.9538461538461539
(6) 데이터 전처리
- 고정산도, 염화물, 품질 boxplot
fig = go.Figure()
fig.add_trace(go.Box(y=X['fixed acidity'], name = 'fixed acidity'))
fig.add_trace(go.Box(y=X['chlorides'], name = 'chlorides'))
fig.add_trace(go.Box(y=X['quality'], name = 'quality'))
fig.show()
-
컬럼들의 최대/최소 범위가 각각 다르고, 평균과 분산이 각각 다르다.
-
특성(feature)의 편향 문제는 최적의 모델을 찾는데 방해가 될 수도 있다
➡️ MinMaxScaler와 StandardScaler
- MinMaxScaler, StandardScaler 학습
from sklearn.preprocessing import MinMaxScaler, StandardScaler
MMS = MinMaxScaler()
SS = StandardScaler()
X_mms = MMS.fit_transform(X)
X_ss = SS.fit_transform(X)
X_ss_pd = pd.DataFrame(X_ss, columns=X.columns)
X_mms_pd = pd.DataFrame(X_mms, columns=X.columns)- MinMaxScalar
최대 최소값을 1과 0으로 강제로 맞추는 것
fig = go.Figure()
fig.add_trace(go.Box(y=X_mms_pd['fixed acidity'], name = 'fixed acidity'))
fig.add_trace(go.Box(y=X_mms_pd['chlorides'], name = 'chlorides'))
fig.add_trace(go.Box(y=X_mms_pd['quality'], name = 'quality'))
fig.show()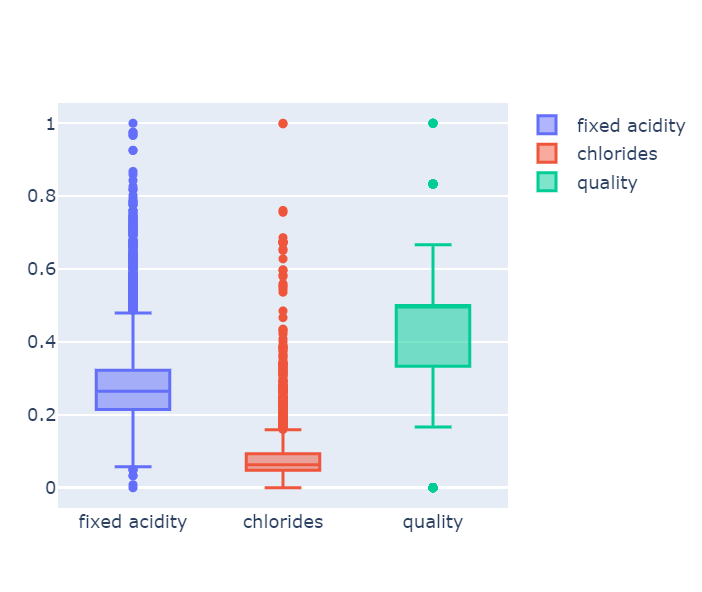
: 학습
X_train, X_test, y_train, y_test = train_test_split(
X_mms_pd, y, test_size=0.2, random_state=2
)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=2)
# 학습
wine_tree.fit(X_train, y_train)
# 학습데이터 예측
y_pred_tr = wine_tree.predict(X_train)
# 테스트데이터 예측
y_pred_test = wine_tree.predict(X_test)
# 학습데이터 정확도
print(accuracy_score(y_train, y_pred_tr))
# 테스트데이터 정확도
print(accuracy_score(y_test, y_pred_test))⬇️
0.9653646334423706
0.9576923076923077
- StandardScaler
평균을 0으로 표준편차를 1로 맞추는 것
fig = go.Figure()
fig.add_trace(go.Box(y=X_ss_pd['fixed acidity'], name = 'fixed acidity'))
fig.add_trace(go.Box(y=X_ss_pd['chlorides'], name = 'chlorides'))
fig.add_trace(go.Box(y=X_ss_pd['quality'], name = 'quality'))
fig.show()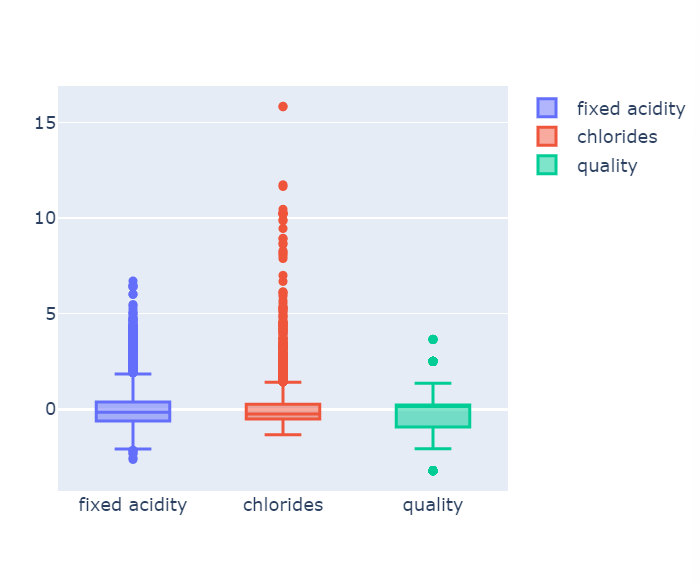
: 학습
X_train, X_test, y_train, y_test = train_test_split(
X_ss_pd, y, test_size=0.2, random_state=2
)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=2)
# 학습
wine_tree.fit(X_train, y_train)
# 학습데이터 예측
y_pred_tr = wine_tree.predict(X_train)
# 테스트데이터 예측
y_pred_test = wine_tree.predict(X_test)
# 학습데이터 정확도
print(accuracy_score(y_train, y_pred_tr))
# 테스트데이터 정확도
print(accuracy_score(y_test, y_pred_test))⬇️
0.9653646334423706
0.9576923076923077
- 🚩 레드/화이트 구분에 영향도
dict(zip(X_train.columns, wine_tree.feature_importances_))⬇️
{'fixed acidity': 0.0,
'volatile acidity': 0.0,
'citric acid': 0.0,
'residual sugar': 0.0,
'chlorides': 0.765109853101628,
'free sulfur dioxide': 0.0,
'total sulfur dioxide': 0.23489014689837195,
'density': 0.0,
'pH': 0.0,
'sulphates': 0.0,
'alcohol': 0.0,
'quality': 0.0}📌 chlorides의 영향이 가장 크다
(7) 이진분류
와인 맛(quality)에 대한 분류
- 와인 quality : 맛 이진화
wine['taste'] = [1 if grade > 5 else 0 for grade in wine['quality']]📌 5등급초과 1 / 이하 0
- 훈련 / 테스트 데이터 분리
X = wine.drop(['taste','quality'], axis=1)
y = wine['taste']
X_train, X_test, y_train, y_test = train_test_split(
X,y, test_size= 0.2, random_state=2
)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=2)
wine_tree.fit(X_train, y_train)📌 taste는 quality로 만든 컬럼이므로,
quality도 drop 시켜줘야함
- train, test accuracy 확인
y_pred_tr = wine_tree.predict(X_train)
y_pred_test = wine_tree.predict(X_test)
print(accuracy_score(y_train,y_pred_tr))
print(accuracy_score(y_test,y_pred_test))0.7394650760053877
0.7307692307692307
- decision tree 확인
from sklearn.tree import plot_tree
import matplotlib.pyplot as plt
plt.figure(figsize=(12,8))
plot_tree(wine_tree, feature_names=X_train.columns.to_list()
,filled=True);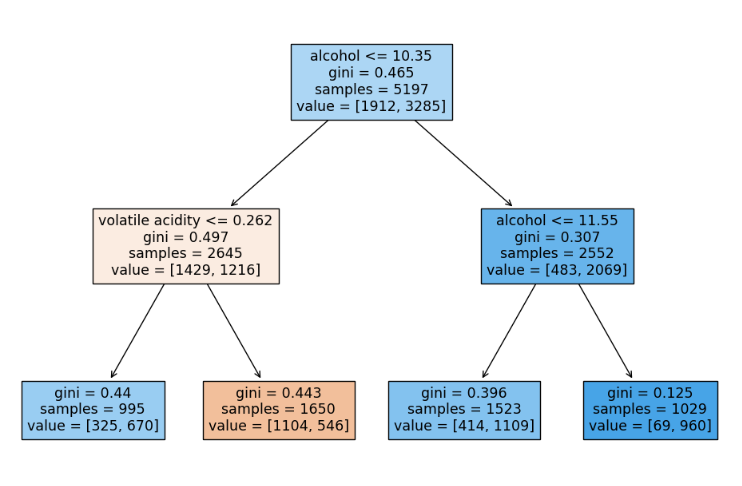
📌 알코올 도수가 높으면 맛있다..? ㅋㅋㅋ(ㅇㅈ!)
2. Pipeline
- 데이터의 전처리와 여러 알고리즘의 반복 실행, 하이퍼 파라미터의 튜닝 과정을 번갈아 하다 보면 코드의 실행 순서에 혼돈이 있을 수 있다.
- 이런 경우 클래스(class)로 만들어서 진행해도 되지만, sklearn 유저에게는 꼭 그럴 필요없이 준비된 기능인 Pipeline이 있다.
- 데이터준비
import pandas as pd
red_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-red.csv'
white_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-white.csv'
red_wine = pd.read_csv(red_url, sep=';')
white_wine = pd.read_csv(white_url, sep=';')
red_wine['color'] = 1.
white_wine['color'] = 0.
wine = pd.concat([red_wine, white_wine])
X = wine.drop(['color'], axis=1)
y = wine['color']- 레드/화이트 와인 분류기의 동작 Process
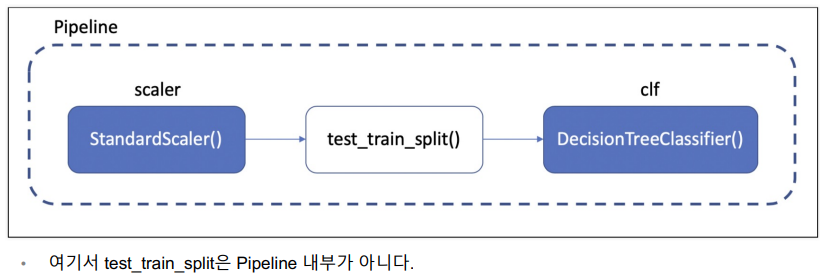
(기존)
StandardScaler 변환 -> 학습 -> 정확도체크
(pipelime)
StandardScaler 변환 & 학습 -> 정확도체크
(1) pipeline 실행
from sklearn.pipeline import Pipeline
from sklearn.tree import DecisionTreeClassifier
from sklearn.preprocessing import StandardScaler
estimators = [
('scaler', StandardScaler()), # 첫번째 단계의 (객체 이름, 객체)
('clf', DecisionTreeClassifier()) # 두번째 단계의 (객체 이름, 객체)
]
pipe = Pipeline(estimators) # 파이프라인 생성📌estimators에 수행내용을 적어줌
(2)steps 확인
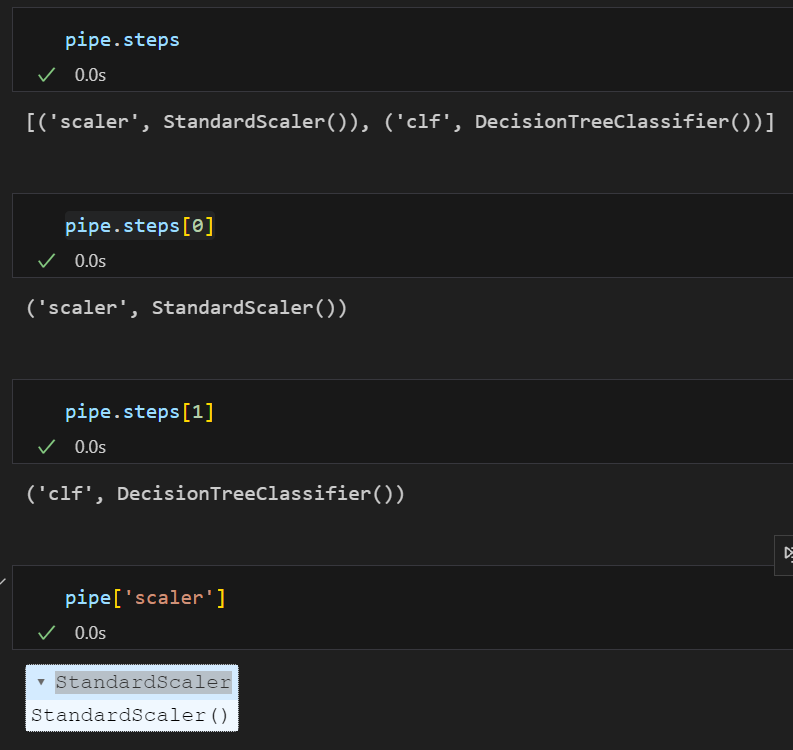
(3) 속성 파라미터 입력
파라미터 지정은 pipe.setparams(객체명__파라미터명 = 값) 을 이용하면 된다. (언더바가 2개인 것에 주의⭐)
간단하게 DecisionTree의 max_depth와 random_state 파라미터만 조정한다고 하면 아래와 같이 작성할 수 있다.
pipe.set_params(clf__max_depth = 2)
pipe.set_params(clf__random_state= 2)
(4) 데이터 분리 & 학습
과적합 방지를 위해 훈련용/테스트용 데이터를 분리해주고 학습(fit)시킨 후 성능을 평가
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.2, random_state=2, stratify=y
)
# 이전에는 스케일러를 통과시키고 분류기에 학습시켜두었는데
# 그 과정이 pipe 변수에 저장이 되어있으므로 간단하게 아래 코드로 실행 가능하다.
pipe.fit(X_train,y_train)📌 pipe에 바로 fit
(5) 정확도예측
from sklearn.metrics import accuracy_score
y_pred_tr = pipe.predict(X_train)
y_pred_test = pipe.predict(X_test)
print('Train : ', accuracy_score(y_train, y_pred_tr))
print('Test : ', accuracy_score(y_test, y_pred_test))Train : 0.9667115643640561
Test : 0.9538461538461539
3. 교차검증
참고링크 : https://blog.naver.com/sjy5448/222427780700
(1) kfold
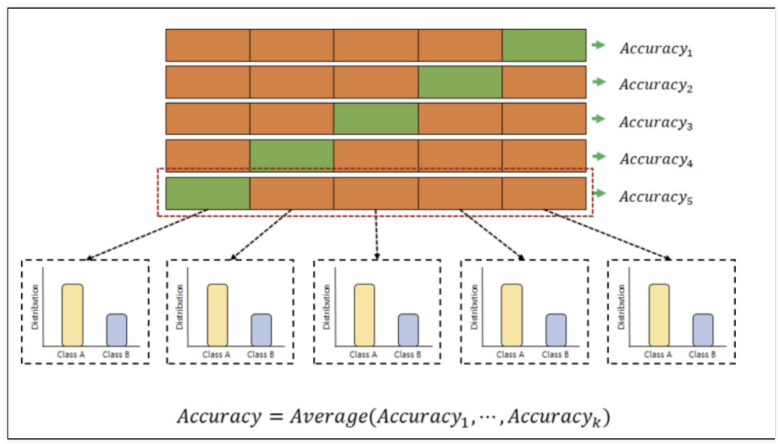
훈련용 데이터를 또 훈련용 / 검증용 데이터로 분리하여 검증을 좀 더 여러 번 한 작업
모델의 알반화 성능을 평가하기 위해 사용
- 기존 학습 및 정확도
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.2, random_state=2
)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=2)
# 학습
wine_tree.fit(X_train, y_train)
# 학습데이터 예측
y_pred_tr = wine_tree.predict(X_train)
# 테스트데이터 예측
y_pred_test = wine_tree.predict(X_test)
# 학습데이터 정확도
print('Train Acc : ', accuracy_score(y_train, y_pred_tr))
# 테스트데이터 정확도
print('Test Acc : ', accuracy_score(y_test, y_pred_test))⬇️
Train Acc : 0.9653646334423706
Test Acc : 0.9576923076923077- 데이터준비
# 1) 데이터 불러오기 & concat
import pandas as pd
red_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-red.csv'
white_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-white.csv'
red_wine = pd.read_csv(red_url, sep=';')
white_wine = pd.read_csv(white_url, sep=';')
red_wine['color'] = 1.
white_wine['color'] = 0.
wine = pd.concat([red_wine, white_wine])
# 2) 맛 분류를 위한 데이터 정리
wine['taste'] = [1. if grade > 5 else 0 for grade in wine['quality']]
X = wine.drop(['taste','quality'], axis= 1)
y = wine['taste']
# -> 데이터와 라벨- KFold
검증하는 것을 3번이든, 5번이든, 여러번 한다는 의미로 교차검증을 k-fold croos validation이라 부른다.
또한 각 구분된 데이터 셋에 타켓 데이터의 비중을 비슷하게 맞추어 분리하는 것을 stratified k-fold cross validation이라 한다.
# 모듈
from sklearn.model_selection import KFold
# n_splits는 몇 개의 폴드(fold)로 나눌 것인지를 의미하는 매개변수
# 5겹 교차 검증이 가장 일반적
kfold = KFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
# 5) 모델링 (학습 & 결과 확인)
# 기록 보관을 위해 '빈리스트'생성
cv_accuracy =[]
# ->kfold는 idx를 반환한다.
for train_idx, test_idx in kfold.split(X):
# 데이터 구성
X_train, X_test = X.iloc[train_idx], X.iloc[test_idx]
y_train, y_test = y.iloc[train_idx], y.iloc[test_idx]
# 학습
wine_tree_cv.fit(X_train, y_train)
# 모델링
pred = wine_tree_cv.predict(X_test)
# 리스트 저장 (결과값)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy⬇️
[0.6007692307692307,
0.6884615384615385,
0.7090069284064665,
0.7628945342571208,
0.7867590454195535]- 평균 Acc
np.mean(cv_accuracy)⬇️
0.709578255462782다중 클래스 분류 문제에서 클래스 비율이 불균형한 경우에서 각 폴드가 대표적인 샘플들로 이루어져 있지 않고 편향될 가능성이 있다. 만약 전체 데이터 셋의 90%가 양성 레이블을 가지고 있다면 일반적인 K-Fold 교차 검증에서는 각 fold에서 레이블의 분포가 90%, 10%로 나누어지기 때문에 학습셋과 검증셋 모두 음성 레이블이 거의 포함되지 않아 양성 레이블에 편향되게 학습될 가능성이 있다.
(2) Stratified kfold
- Stratified KFold
K-Fold와 달리, Stratified k-fold 교차 검증은 각 fold에서 양성, 음성 레이블의 비율이 전체 데이터셋의 비율과 유사하게 유지되므로, 각 폴드가 대표적인 샘플로 이루어지며, 모델이 레이블의 편향을 학습하는 것을 방지할 수 있다.
from sklearn.model_selection import StratifiedKFold
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cv_accuracy =[]
for train_idx, test_idx in skfold.split(X,y):
# 데이터 구성
X_train, X_test = X.iloc[train_idx], X.iloc[test_idx]
y_train, y_test = y.iloc[train_idx], y.iloc[test_idx]
# 학습
wine_tree_cv.fit(X_train, y_train)
# 모델링
pred = wine_tree_cv.predict(X_test)
# 리스트 저장 (결과값)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy⬇️
[0.5523076923076923,
0.6884615384615385,
0.7143956889915319,
0.7321016166281755,
0.7567359507313318]- 평균 Acc
np.mean(cv_accuracy)⬇️
0.6888004974240539📌 Stratified kfold의 acc가 더 나쁘다
(3) cross_val_score
위 코드를 보다 간편하게 할 수 있다.
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cross_val_score(wine_tree_cv, X, y, scoring=None, cv= skfold)➡️ array([0.55230769, 0.68846154, 0.71439569, 0.73210162, 0.75673595])
- depth 함수
depth를 바꿔가며 acc를 확인하고싶을 때
def skfold_dt(depth) :
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=depth, random_state=13)
print(cross_val_score(wine_tree_cv, X, y, scoring=None, cv= skfold))- train score 확인
from sklearn.model_selection import cross_validate
cross_validate(wine_tree_cv, X, y, scoring=None, cv=skfold, return_train_score=True)⬇️
{'fit_time': array([0.01210546, 0.01591825, 0.01565552, 0.0159409 , 0.02544117]),
'score_time': array([0. , 0. , 0. , 0.00654483, 0. ]),
'test_score': array([0.55230769, 0.68846154, 0.71439569, 0.73210162, 0.75673595]),
'train_score': array([0.74773908, 0.74696941, 0.74317045, 0.73509042, 0.73258946])}📌 train보다 test의 acc가 더 낮다 : 과적합
4. 하이퍼파라미터 튜닝
모델의 성능을 확보하기 위해 조절하는 설정값
이는 일일이 다 조절해보는 수 밖에 없는데, 그나마 GridSearchCV가 우리에게 도움을 준다.
- 데이터준비
# 1) 데이터 불러오기 & concat
import pandas as pd
red_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-red.csv'
white_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-white.csv'
red_wine = pd.read_csv(red_url, sep=';')
white_wine = pd.read_csv(white_url, sep=';')
red_wine['color'] = 1.
white_wine['color'] = 0.
wine = pd.concat([red_wine, white_wine])
# 2) 맛 분류를 위한 데이터 정리
wine['taste'] = [1. if grade > 5 else 0 for grade in wine['quality']]
X = wine.drop(['taste','quality'], axis= 1)
y = wine['taste']
# X = 데이터 / y = 맛 라벨- GridSearchCV
from sklearn.model_selection import GridSearchCV
우리가 조절해야하는 파라미터의 수가 엄청 많을 것이다. 파라미터가 5개라고 해도 각 파라미터에 들어갈 값들과 그 파라미터들을 조절해서 만들 수 있는 분류기의 경우의 수는 엄청 많다.
이를 좀 더 간단히 나타낼 수 있게 해주는 것이 GridSearchCV이다.
결과를 확인하고 싶은 파라미터를 정의하고 이를 GridSearchCV에 넣어주면 된다.
from sklearn.model_selection import GridSearchCV
from sklearn.tree import DecisionTreeClassifier
params = {'max_depth' : [2,4,7,10]}
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=42)
# estimator = 분류기
# param_grid = 파라미터
# cv = (cross validation)5겹
gridsearch = GridSearchCV(estimator=wine_tree, param_grid=params, cv = 5)
gridsearch.fit(X,y)
# train_test_split을 GridSeach가 알아서함
- GridSearchCV 결과
(1) rank
import pprint
import pprint
pp = pprint.PrettyPrinter(indent=4)
pp.pprint(gridsearch.cv_results_)⬇️
{ 'mean_fit_time': array([0.00667 , 0.01027937, 0.0137569 , 0.02942438]),
'mean_score_time': array([0.00140371, 0.00208001, 0.00692248, 0.00071783]),
'mean_test_score': array([0.6888005 , 0.66356523, 0.65463895, 0.64432392]),
'param_max_depth': masked_array(data=[2, 4, 7, 10],
mask=[False, False, False, False],
fill_value='?',
dtype=object),
📌'params': [ {'max_depth': 2},
{'max_depth': 4},
{'max_depth': 7},
{'max_depth': 10}],
📌'rank_test_score': array([1, 2, 3, 4]),
'split0_test_score': array([0.55230769, 0.51230769, 0.51692308, 0.51076923]),
'split1_test_score': array([0.68846154, 0.63153846, 0.60307692, 0.60538462]),
'split2_test_score': array([0.71439569, 0.72363356, 0.6812933 , 0.66974596]),
'split3_test_score': array([0.73210162, 0.73210162, 0.73595073, 0.71439569]),
'split4_test_score': array([0.75673595, 0.7182448 , 0.73595073, 0.7213241 ]),
'std_fit_time': array([0.00362313, 0.00290087, 0.00716285, 0.00634075]),
'std_score_time': array([0.00140127, 0.00301846, 0.00689966, 0.00063393]),
'std_test_score': array([0.07179934, 0.08390453, 0.08433027, 0.07853469])}2,4,7,10 각각 순위가 1,2,3,4등
max_depth가 높을수록 성능이 좋지 않았다는 것을 확인할 수 있다.
(2) 최적의 성능을 가진 모델과 점수는?
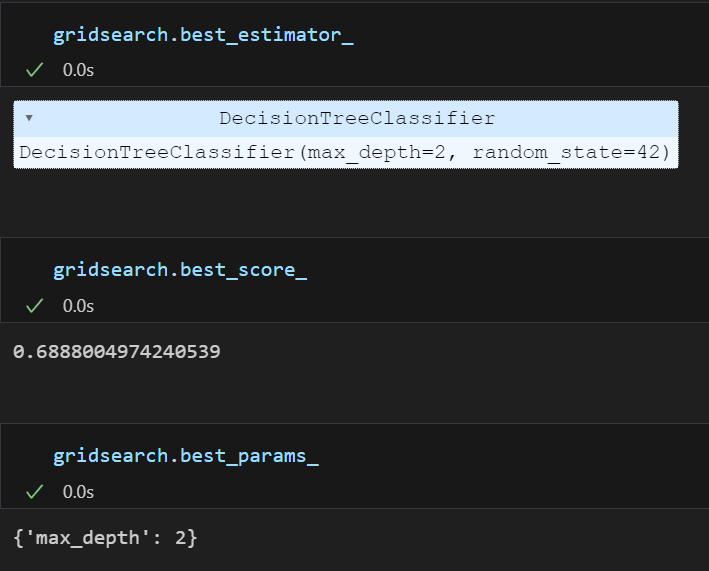
max_depth가 2일 때 정확도 0.68..의 정확도로 가장 성능이 높다.
- GridSearchCV + pipeline
만약 pipeline에 GridSearch를 적용하고 싶다면???
params = {'clf__max_depth' : [2,4,7,10]}
# 분류기(estimator)에 pipe할당
gridsearch = GridSearchCV(estimator=pipe, param_grid=params, cv = 5)
gridsearch.fit(X,y)앞서 파이프라인에서 파라미터 값을 clf__파라미터명으로 작성했던 것처럼 작성해주고 이를 GridSearchCV에 넣어주면 된다.
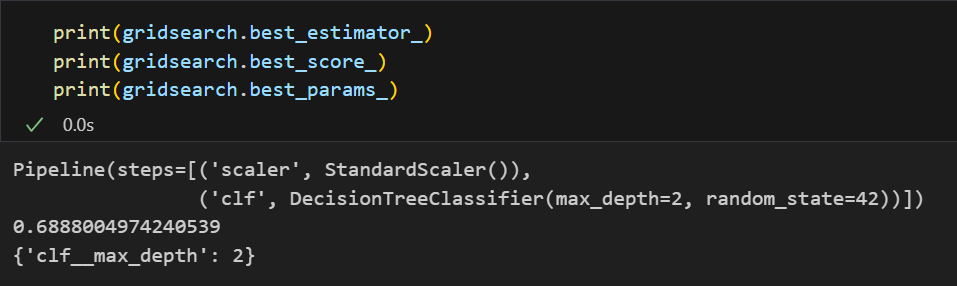
결과확인도 동일하다
import pandas as pd
score_df = pd.DataFrame(gridsearch.cv_results_)
score_df = score_df[['params','rank_test_score','mean_test_score','std_test_score']]
score_df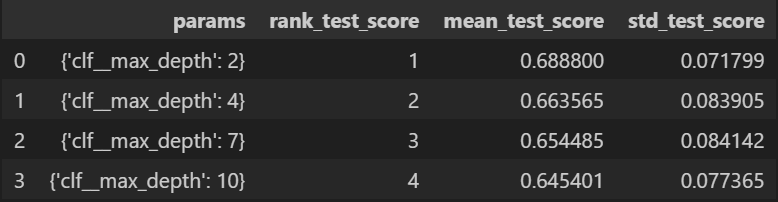
데이터프레임으로 깔끔하게 정리도 가능하다.
