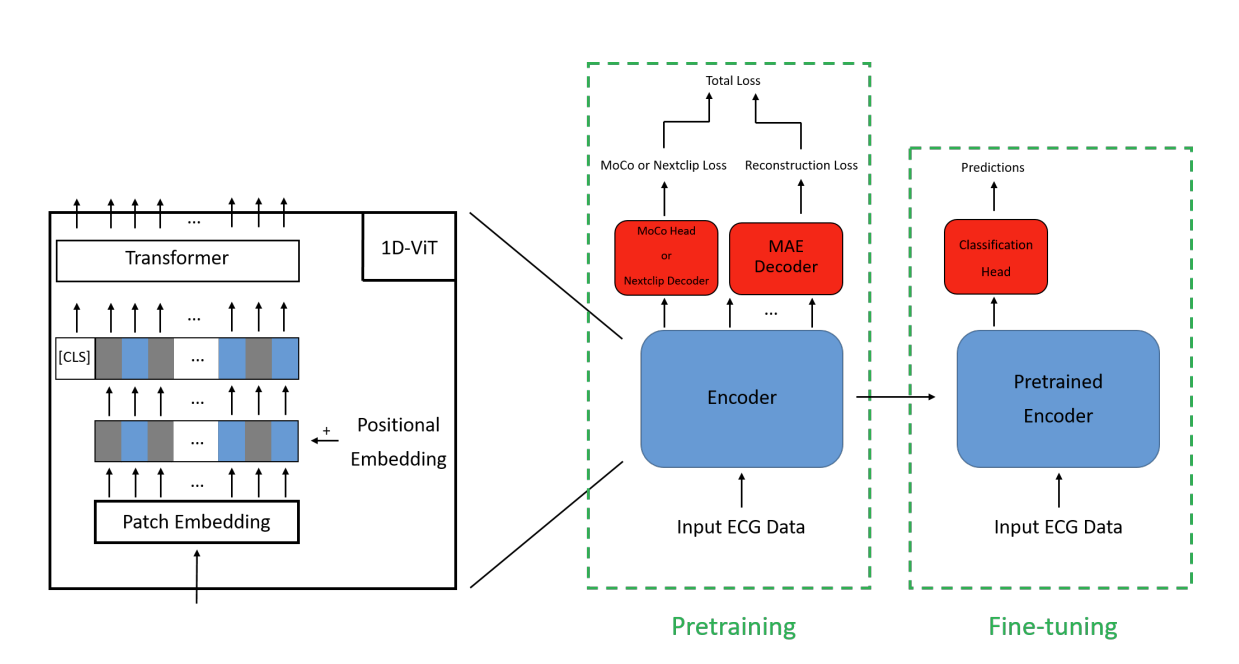
MAE-MoCo
https://openreview.net/pdf?id=iTjqAWYw2K
요약
- Goal : ECG 해석 (심혈관 질환)
- Dataset : ECG 데이터
- Architecture : Hybrid Self-Supervised Learning(SSL) 학습 프레임워크
- MAE-MoCo : masked autoencoder와 momentum encoder를 결합하여 생성+대조 동시 학습
- MAE-Nextclip : Nextclip + [CLS] 토큰으로 마스킹된 패치뿐만 아니라 전체 구조까지 재구성
Introduction
- 12 Lead ECG 신호는 시계열(time series) 및 2차원 의료 이미지로 간주될 수 있음
- ECG 분야에서는 SSL의 contrastive learning(CL) 및 generative learning(GL) 프레임워크가 뛰어난 성능을 보여준다.
- CL 사전 연구
ISL(Inter-Subject Learning)는 ECG에 특화된 대조적 학습 방법 중 하나는, 이는 다변량 심장 신호(multivariate cardiac signals)에 맞춰 설계되었다. 이 대조적 학습 방법은 피험자 내 학습(intra-subject learning)과 피험자 간 학습(inter-subject learning) 기법을 결합한다. 피험자 내 학습은 동일한 환자의 ECG 신호에서 시간적 종속성(temporal dependency)을 분석하는 반면, 피험자 간 학습은 같은 환자의 증강된 신호를 양성 쌍(positive pairs) 으로, 다른 환자의 신호를 음성 쌍(negative pairs) 으로 취급하여 대조 손실(contrastive loss)을 계산한다. - GL 사전 연구
ECG 분야에서 제안된 모델들이 주로 마스킹된 오토인코딩(masked autoencoding, MAE) 기법을 기반으로 하고 있다. 마스킹된 시간축 오토인코더(MTAE)는 시간 축에서 마스킹된 ECG 패치를 찾도록 사전 학습되며, 마스킹된 리드 축 오토인코더(MLAE)는 리드 축을 따라 마스킹된 패치를 재구성한다. 이러한 모델들은 현재 ECG 분류(classification) 분야에서 최첨단(state-of-the-art) 성능을 기록하고 있다. 추가적으로, MassMIB는 마스킹 기법을 활용한 또 다른 방법으로, 인코더-디코더 쌍을 포함하여 시간축에서 마스킹된 신호를 인코딩하고 해당 마스킹된 패치를 복원하는 방법이다. 또한, Nextclip은 ECG 데이터를 다음 반심장 주기(semi-cardiac cycle)까지 예측하는 방식으로 재구성하는 사전 학습 과제를 수행한다. - 최신 연구 경향
Transformer 기반 아키텍처 가 MAE와 결합되면서 GL에서 최첨단 성능을 달성하게 되었다. ViT 가 ECG 관련 사전 학습 작업에서 ResNet을 능가하는 성능을 보이고 있음. ViT의 주요 특징 중 하나는 [CLS] 토큰 을 포함한다는 점이며, ECG 사전 학습 단계에서의 최적화 가능성이 존재한다. - 연구 목표
[CLS] 토큰을 포함한 강력한 사전 학습 작업을 설계하여 ECG 신호 표현을 위한 하이브리드 학습 프레임워크 를 구축하는 것. 이를 위해 두 가지 접근법을 탐구하였다.
1. MAE-MoCo
1D-ViT 기반 MAE 프레임워크의 [CLS] 토큰 출력에 대조적 투영 헤드를 추가하는 방식.
ECG 신호 재구성과 positive-negative pairs 분류를 동시에 수행하는 인코더 개발.
2. MAE-Nextclip
Nextclip 디코더를 통합하여 생성적 자기지도 학습 프레임워크를 구성하는 방식.
마스킹된 패치 및 ECG의 다음 반심장 주기(semi-cardiac cycle)를 재구성
Method
MAE-MoCo 모델은 생성적(generative) 및 대조적(contrastive) 학습 관점을 통합하며, MAE-Nextclip은 두 가지 독립적인 생성적 학습 과제를 결합한다.
A. Model Architecture
1) MAE-MoCo
MAE-MoCo 모델은 인코더(encoder), 디코더(decoder), 모멘텀 인코더(momentum encoder) 의 세 가지 주요 블록으로 구성된다. 인코더는 1D-ViT(1D Vision Transformer) 구조를 기반으로 하며, MAE와 MoCo의 두 부분으로 구성된다.
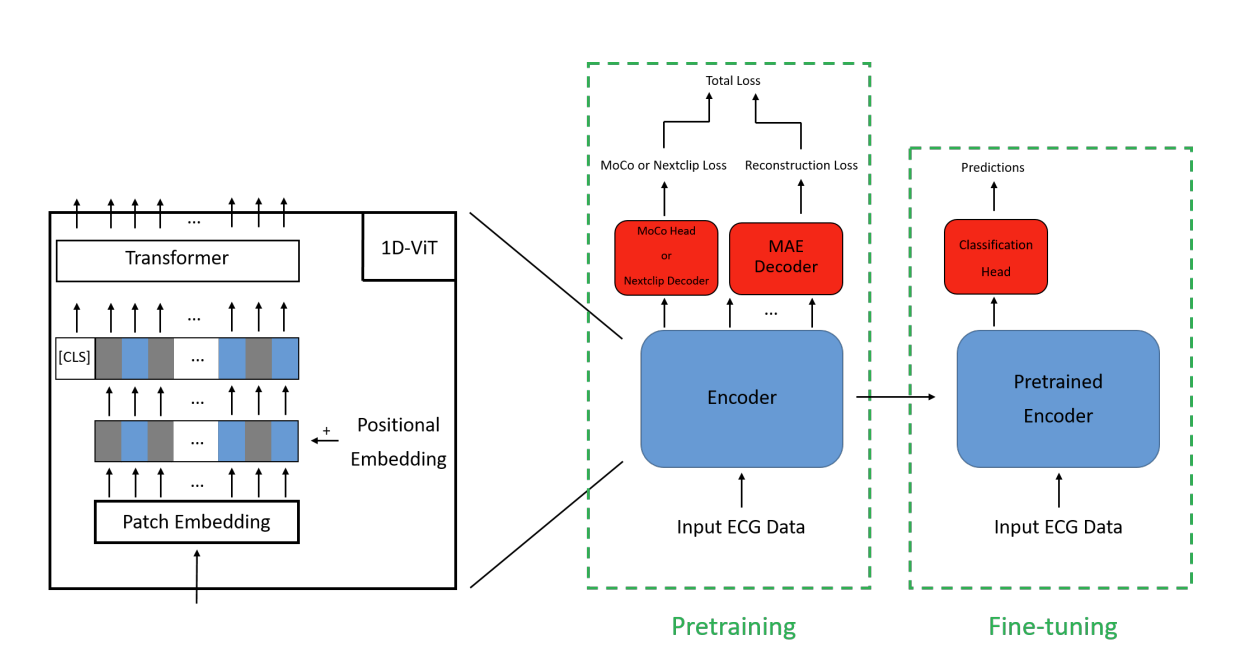
Fig 1. 파란색 : unmasked patches, 회색 : masked patches
- MAE Encoder
- 원본 ECG 데이터 입력 (증강 없이 입력)
- Patch Embedding 통과
- 이 블록은 1D CNN 커널 및 스트라이드 크기가 패치 크기와 동일하며, ECG 데이터를 지정된 크기의 패치로 나누고 이를 숨겨진(hidden) 크기에 매핑한다.
- positional embedding 추가
- 기존 연구에서 고정된 사인(sine) 임베딩을 사용하지만 [18], 본 연구에서는 학습 가능한 위치 임베딩(learnable positional embedding) 을 사용하여 MAE 및 MoCo 모두에 적용하였다.
- 이후, 일부 패치는 랜덤 마스킹(random masking) 되어 숨겨진 상태로 유지되며, 마스킹되지 않은 패치만 디코더에서 사용된다.
- 최종적으로, 마스킹된 ECG 신호는 여섯 개의 연속적인 transformer 블록과 layer normalization 블록을 통과한다.
- MoCo Encoder
- 랜덤하게 증강된 데이터 입력
- 이 신호는 패치 임베딩 및 위치 임베딩 레이어를 거치며, MAE 부분과 달리 마스킹을 적용하지 않는다. 대신, 학습 가능한 [CLS] 토큰 을 추가하여 ECG 신호의 전체 구조를 표현한다.
- 이후, 신호는 트랜스포머 블록(transformer block) 과 레이어 정규화(layer normalization) 블록을 거친다.
- 마지막으로, [CLS] 토큰만 추출하여 Linear-Batch Normalization-ReLU 블록을 통과해 최종 MoCo 출력을 얻는다.
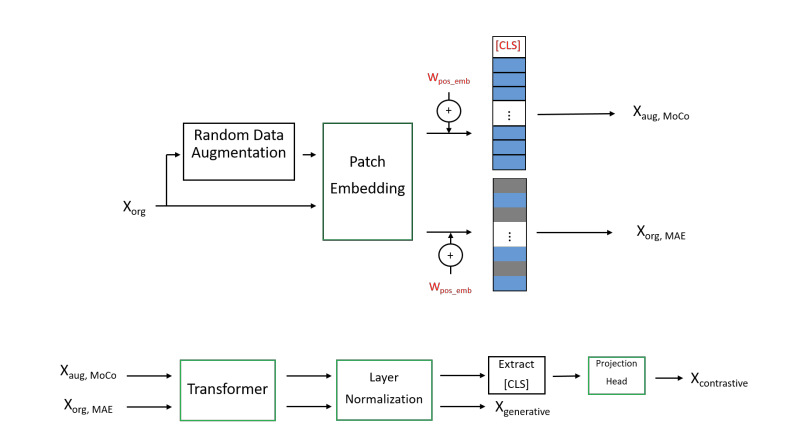
Fig 2. Encoder Block. X_org : 원본 ECG, X_generative : MAE에 사용된 masked latent representation, X_contrastive : MoCo에 사용된 latent representation.
- Decoder
- 마스킹된 패치 재구성 단계. 마스킹된 패치는 생성적 학습 출력을 기반으로 하여 최소한의 오류로 복원되며, 이 과정에서 새로운 학습 가능한 파라미터가 적용된다.
- 디코더는 x_generative 입력을 받아 패치 임베딩 블록을 통과한다.
- 이는 인코더의 패치 임베딩 블록과 구조적으로 다르며, 패치를 개별적으로 처리하지 않고 선형 레이어를 사용하여 직접 매핑한다.
- 이후, 학습 가능한 [mask] 토큰 을 통해 마스킹된 패치를 디코딩한다.
- 디코더는 트랜스포머 블록과 레이어 정규화 블록을 거쳐 최종적으로 마스킹된 패치를 원래 ECG 신호에 가깝게 복원한다.
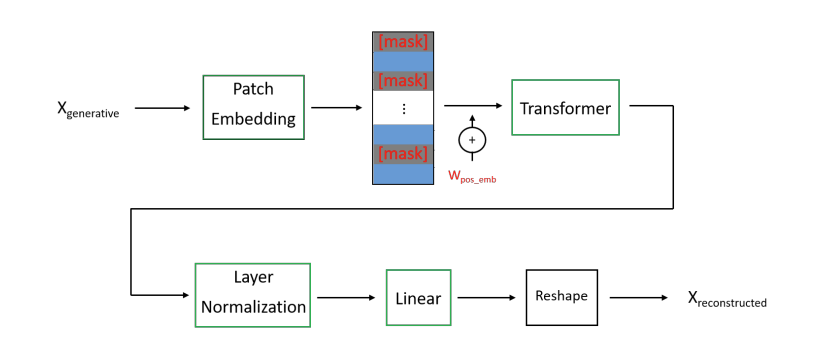
Fig 3. Decoder Block. masked patches를 재구성하기 위한 학습 가능한 [mask] 토큰
- Momentum Encoder
- MAE-MoCo의 모멘텀 인코더는 X_generative 출력을 무시하면서 동일한 인코더 아키텍처를 사용한다.
- 데이터 증강 기법은 MoCo와 다르게 설정되며, MoCo에서 사용되는 파라미터와 동일하지만 모멘텀 업데이트가 적용된다.
- 각 배치마다 새로운 데이터가 추가되고, 가장 오래된 배치는 제거되며, 새로운 배치가 추가되는 방식으로 MoCo 학습 원칙을 따른다.
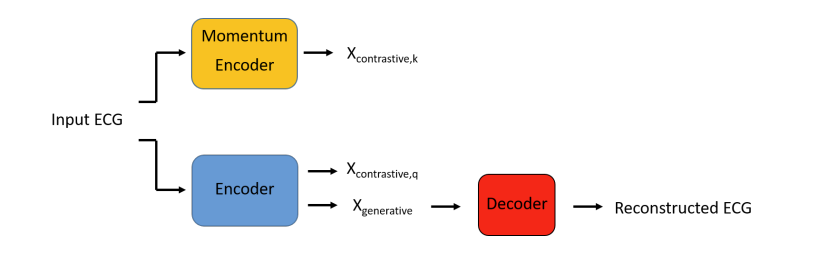
Fig 4. MAE-MoCo 프레임워크의 전체적인 흐름
2) MAE-Nextclip
MAE-Nextclip 프레임워크는 공유된 인코더(shared encoder) 와 MAE 및 Nextclip 복원을 위한 두 개의 개별 디코더(separate decoders) 로 구성된다. 아키텍처적으로나 기능적으로 이 모델은 MAE-MoCo 모델과 상당한 유사성을 갖는다.
- MAE 인코더 구성 요소 : 앞서 섹션 II-A1에서 설명한 내용과 동일하다.
- Nextclip Endoer : 원본 ECG 데이터를 Nextclip 데이터로 변환하는 과정이 포함된다.
- ECG 데이터의 일부 패치 내에서 랜덤한 시작 지점(random starting point) 을 선택한다.
- 선택된 랜덤 지점을 기준으로 반심장 주기(semi-cardiac cycle) 를 추출하여 정답 데이터(ground truth data) 로 사용한다.
- 이 랜덤 지점 이후의 ECG 데이터는 인코더에서 모두 0으로 설정된다.
- 변환된 ECG 데이터는 패치 임베딩(patch embedding) 및 위치 임베딩(positional encoding) 을 적용한 후 Nextclip 디코더로 전달된다.
- 또한, [CLS] 토큰 도 Nextclip 디코더에서 활용할 수 있도록 추가된다.
- 최종 신호는 transformer 블록과 layer normalization 블록을 연속적으로 통과하여 변환된다.
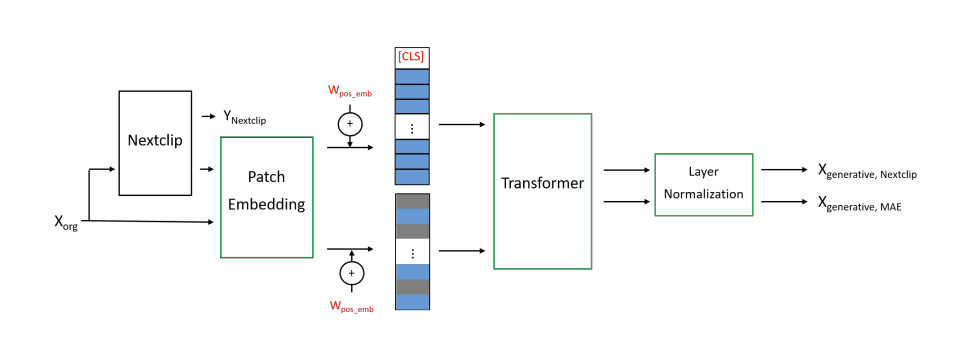
Fig 5. MAE-Nextclip Encoder.
Y_Nextclip : Nextclip 디코더를 거쳐 복원될 ECG 신호의 정답 데이터(ground truth ECG cycle)
X_generative,Nextclip & X_generative,MAE : 각각 Nextclip 및 MAE에 대한 인코딩된 신호
- Nextclip 디코더 구성:
- MAE 디코더는 앞서 제시된 Fig 3과 동일한 구조를 갖는다.
- Nextclip 디코더는 [CLS] 토큰을 먼저 추출한 후, 패치 임베딩 단계를 거친다.
- 이후, [mask] 토큰을 사용하지 않고, transformer block, layer normalization, linear layer을 순차적으로 통과하면서 Nextclip 신호를 재구성한다.
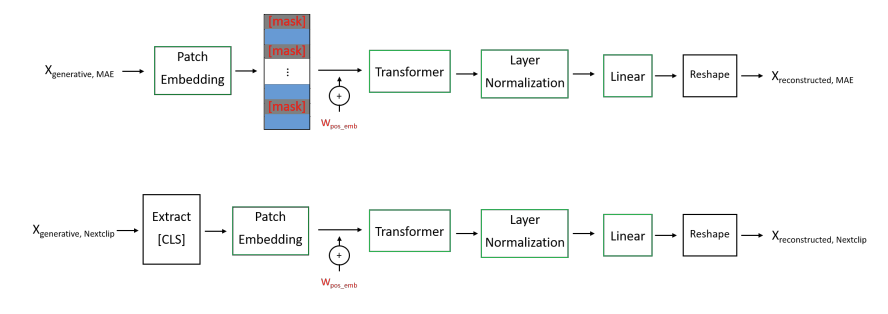
Fig 6. MAE-Nextclip Decoders
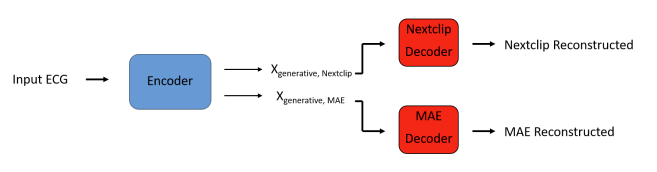
Fig 7. MAE-Nextclip 프레임워크의 전체적인 흐름
B. Training Strategies
1) Pretraining
- 데이터셋
- China Physiological Signal Challenge 2018 (CPSC2018)
- Physikalisch-Technische Bundesanstalt XL (PTB-XL)
- Chapman dataset
- 전처리 : Z-score normalization
- 손실 함수 : 두 가지 손실 함수를 결합하여 사용
- Generative Loss
- 원본 데이터와 마스킹된 패치의 복원된 데이터를 비교
- MSE Loss :
- Contrastive Loss
- 동일한 ECG에서 인코딩된 키와 쿼리는 positive pairs 으로 간주되며, 그렇지 않으면 negative pairs 로 처리
- positive sample :
- negative sample :
- Cross-Entropy Loss :
- MAE-MoCo
- MAE-Nextclip
- α 조정
- MAE-MoCo: α = 40(default) , 20, 10
- MAE-Nextclip: α = 2(default), 1, 0.5
- Generative Loss
- 학습 과정 및 하이퍼파라미터 설정
- 모델은 학습 중 배치별 손실을 최소화하도록 가중치를 업데이트함.
- warm-up training : 초반 특정 횟수의 epoch 동안 학습률을 선형적으로 증가시켜 학습 안정성을 높이고 수렴 속도를 가속화함.
- 학습률 스케줄링 : 학습이 진행될수록 학습률을 점진적으로 감소시킴. validation loss 가 일정 에폭 이후 개선되지 않으면 학습률을 특정 비율로 감소시킴.
2) Fine-tuning
- 데이터셋 : ****Chapman dataset
- 모델 구조 변경 : 인코더만 유지, 나머지 구성 요소(모멘텀 인코더 및 디코더)는 제거
- MAE-MoCo 모델의 인코더에서 사용된 projection head는 파인튜닝에서 사용되지 않음.
- 학습 방법론 : 두 가지 방법 사용
- [CLS] 토큰과 연결된 선형 분류기(linear network)를 추가하여 최적의 분류를 수행하도록 설계됨.
- Linear Probe 방식
- 사전 학습된 인코더의 모든 학습 가능한 파라미터를 고정(동결) 한다.
- [CLS] 토큰과 연결된 새로운 선형 네트워크의 파라미터만 업데이트됨.
- 이 방법은 인코더를 feature extractor 로 사용하여,
- 선형 분류기가 성능을 얼마나 잘 발휘하는지 평가할 수 있도록 함.
- 즉, 사전 학습의 성공 여부를 더 정확하게 평가할 수 있음.
- End-to-End Fine-tuning 방식
- 사전 학습된 인코더 파라미터를 초기화한 상태에서 학습을 시작함.
- 선형 계층과 인코더의 모든 파라미터를 end-to-end 방식으로 함께 학습
- 목표
- 크로스 엔트로피 손실(Cross-Entropy Loss) 최소화
- 모델이 예측한 클래스 확률과 원-핫 인코딩된 정답 레이블 간의 차이를 줄이는 것.
- 전략
- 사전 학습과 동일하게 웜업 학습 및 학습률 스케줄링을 적용.
- 조기 종료(Early Stopping) 적용:
- 검증 손실(validation loss)이 특정 epoch 동안 개선되지 않으면 학습을 중단.
- 최적의 성능을 보인 모델을 테스트 단계에서 사용.
- 미세 조정에 대한 상세한 구성 정보는 표 VII(Table VII)에 제공됨.
C. Definition of the Metrics
- 본 연구에서는 다중 레이블(multi-label) 분류기를 사용하기 때문에 "One-vs-One AUC" 방법을 적용함.
- One-vs-One AUC: 모든 가능한 클래스 쌍(class pairs) 간의 AUC 값을 평균 내어 최종 AUC 값을 계산.
DISCUSSION
A. Limitations
- 주요 한계점:
- 높은 계산 비용
- 최적의 하이퍼파라미터를 찾기 어려움
- MAE-MoCo 및 MAE-Nextclip 프레임워크는 두 가지 서로 다른 사전 학습(pretext) 작업을 결합한 구조이므로,
- 개별 작업보다 더 높은 계산 비용이 요구됨.
- 특히, 대조적 학습(contrastive learning) 모델은 생성적 학습(generative learning) 모델보다 계산 비용이 더 많이 소모됨. 이는 이중 순전파(double forward pass) 가 필요하기 때문임.
- 결과적으로, MAE-MoCo 및 MAE-Nextclip은 다른 모델보다 더 높은 계산 비용을 요구하는 모델로 간주될 수 있음.
- 또 다른 한계점:
- 학습 불가능한(non-trainable) 파라미터 증가
- 두 가지 다른 모델을 결합하면서 학습되지 않는(non-trainable) 파라미터의 개수가 증가함.
- 이러한 증가로 인해, 개별 모델보다 최적의 파라미터 세트를 찾는 작업이 더욱 복잡해짐.
B. Future Work
- 백본 네트워크 개선
- Convolutional Vision Transformer(CvT) 를 도입한 ViT 구조가 ImageNet등 에서 강력한 성능을 보임.
- 다양한 태스크 조합 실험
- 예를 들어, Centerclip 태스크 는 현재 구간이 아닌 랜덤하게 선택된 ECG 세그먼트를 재구성하는 방식으로 작동함.
- 또한, SimCLR, BYOL, DINO [29]와 같은 대체 태스크를 활용하여 조합 가능함.
- 또 다른 전략: 자기지도 학습(self-supervised learning) 태스크의 수를 증가시키는 방법.
- 다양한 생리학적 신호(Physiological Signals) 활용
- ECG와 같은 특성을 가진 다른 생리 신호(Physiological Signals) 모델 실험
- 예: 시간 시계열(time series) + 2D 이미지 를 결합하여 조합 전략의 효과를 더욱 잘 입증 가능
- 하이퍼파라미터 튜닝 강화
- MAE-MoCo 및 MAE-Nextclip에는 많은 고정 파라미터(fixed parameters) 가 포함됨.
- 기존 프레임워크에서 최적의 값으로 설정된 파라미터가 반드시 MAE-MoCo 및 MAE-Nextclip에 최적이라고 보장할 수 없음.
- 따라서, 제안된 모델들은 더욱 최적화된 파라미터를 가질 가능성이 있음.
Hyperparameters
- Encoder
- Data augmentation techniques: Erase, time out, partial noise, drop, and RRC
- Type of patch embedding: 1D Convolution
- Output channels of patch embedding: 512
- Kernel size of patch embedding: 25
- Stride of patch embedding: 25
- Size of positional embedding: (145, 512)
- Size of [CLS] token: (1, 512)
- Type of transformer block: Attention and linear
- Dimension of transformer: 512
- Number of transformer blocks: 6
- Number of attention heads: 16
- MLP ratio of transformer: 2
- Activation of transformer: GELU
- Output features of layer normalization: 512
- Type of projection head: Linear and 1D BatchNorm
- Number of blocks in projection head: 3
- Output features of MLP in projection head: 128
- Output features of projection head: 128
- Decoder
- Type of patch embedding: Linear
- Output channels of patch embedding: 256
- Size of [mask] token: (1,256)
- Size of positional embedding: (144, 256)
- Type of transformer block: Attention and linear
- Dimension of transformer: 256
- Number of transformer blocks: 1
- Number of attention heads: 8
- MLP ratio of transformer: 2
- Activation of transformer: GELU
- Output features of layer normalization: 256
- Type of size matcher network: Linear
- Output channels of size matcher network: 25×1225×12
- Momentum Encoder
- Momentum constant: 0.99
- Temperature of logits: 0.2
- Queue size: 65536
- Pre-Training
- Epochs of MAE-MoCo: 710
- Batch size: 64
- Optimizer: Adam
- Learning rate: 2×10−42×10−4
- Warm-up epochs: 20
- Learning rate scheduler (reduce rate): 0.9
- Learning rate scheduler (number of bad epochs): 15
