U-Net: Convolutional Networks for Biomedical Image Segmentation
Paper: U-Net: Convolutional Networks for Biomedical Image Segmentation
0. Abstract
이 논문에서는 segmentation task에 대해 주어진 training dataset을 더욱 효과적으로 활용할 수 있는 네트워크와 training 전략을 소개한다. contracting path를 통해 물체에 대한 정보 (context)를 포착하고, 대칭 구조인 expanding path를 통해 정확한 위치정보를 얻을 수 있다. 위의 구조를 사용하면 ㅁ우 적은 이미지로도 end-to-end training을 통해 성능을 매우 높일 수 있다.
1. Introduction
Convolutional network은 오래 전부터 주목받아 visual recognition task 분야에서 많이 쓰이고 있지만, 충분한 training set과 충분히 큰 network를 이용해야 한다는 제한이 있었다. (ex-ImageNet)
또한, CNN은 classification task에 주로 쓰이는 반면, biomedical 분야에서는 각 픽셀별로 class를 분류하는 작업이 필요하고, (Segmentation) training dataset이 부족하다는 문제점을 가지고 있다.
이전 연구에서(Ciresan et al) sliding window를 이용해 local region(patch)를 input으로 받아 각각의 픽셀의 class label을 예측하려는 시도가 있었다. 이 방식은 (1)localize가 가능하고, (2)patch로 나누어 사용하기 때문에 training data의 양이 꽤 늘어난다는 장점을 가지고 있다. 하지만, 각각의 patch마다 겹치는 부분이 많아 작업 속도가 느리고, localization과 context의 정확도에 tradeoff가 존재한다는 단점이 있다.
2. Network Architecture
이 논문에서는 FCN에 영향을 받아, 매우 적은 training image를 이용해 segmentation을 정확하게 수행하는 모델을 소개한다.
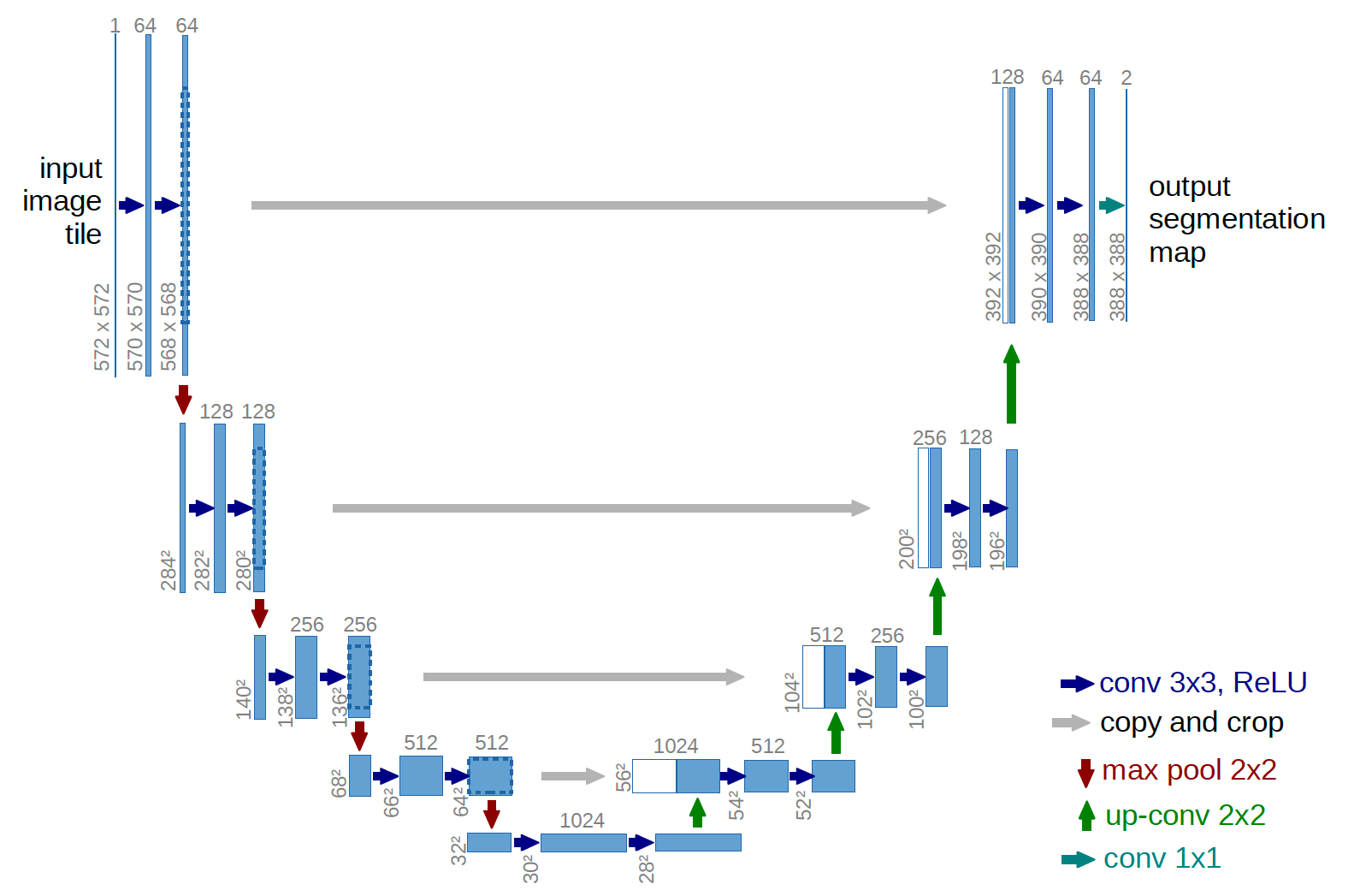
이 모델은 크게 downsampling하는 부분과 upsampling하는 부분 두개로 나누어져 있다. downsampling과정은 이미지의 context를 추출하는 부분이고, upsampling 과정은 localization을 수행한다. 모델을 자세히 살펴보면, 다음과 같이 구성되어 있다:
- conv 3x3, ReLU: downsampling, upsampling 모두 한 단계에 3x3 filter conv와 ReLU layer을 각각 두번씩 거친다. 이때, 따로 padding을 해주지 않기 때문에 최종 output은 input image보다 크기가 작다.
- max pooling 2x2: convolution layer을 두번 거친 후 max pooling방식으로 resolution을 줄여 나간다.
- up-conv 2x2: 마찬가지로 upsampling과정에서 up-convolution을 통해 resolution을 늘인다.
- copy-and-crop: FCN의 skip architecture와 유사한 기법인데, 매 up-conv layer 이후에 같은 단계의 downsampling한 feature map과 concatenate하면서 진행한다.
- conv 1x1: 마지막 layer로 1x1 convolution 연산을 수행한다.
또한, 이 모델은 FCN의 영향을 받아 Fully connected layer(FC Layer)가 없다.
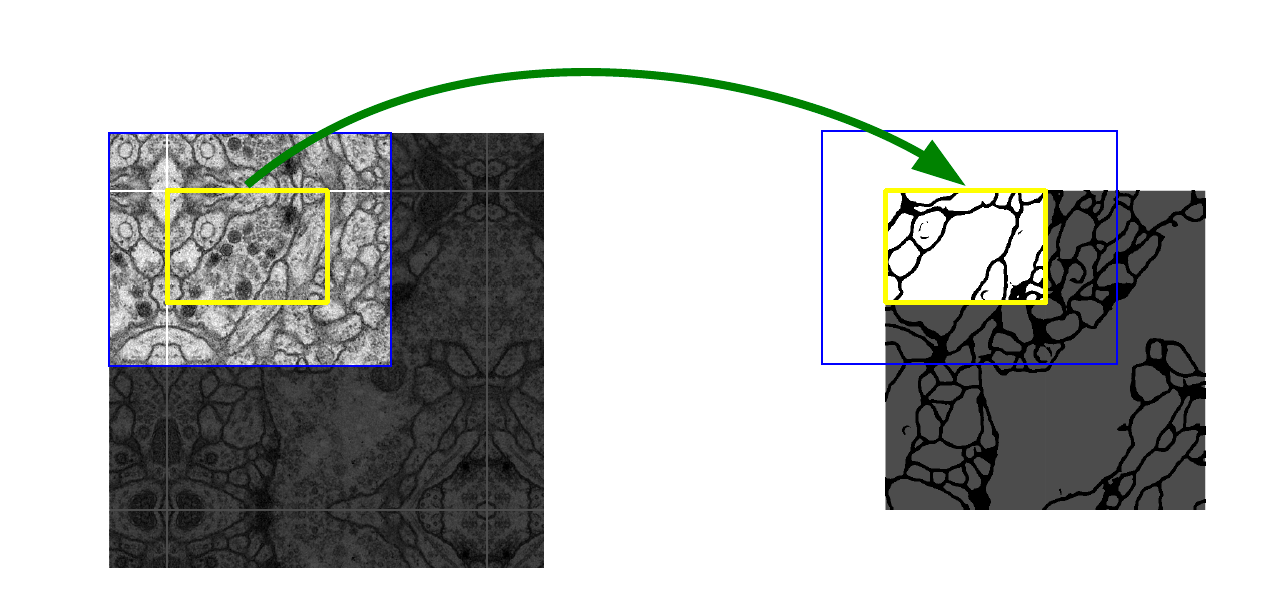
Overlap-tile
이미지가 큰 경우에는, 다음과 같이 이미지를 잘라서 patch를 input으로 사용했다. 파란색 영역의 input을 통해 노란색 영역의 output을 얻을 수 있다.
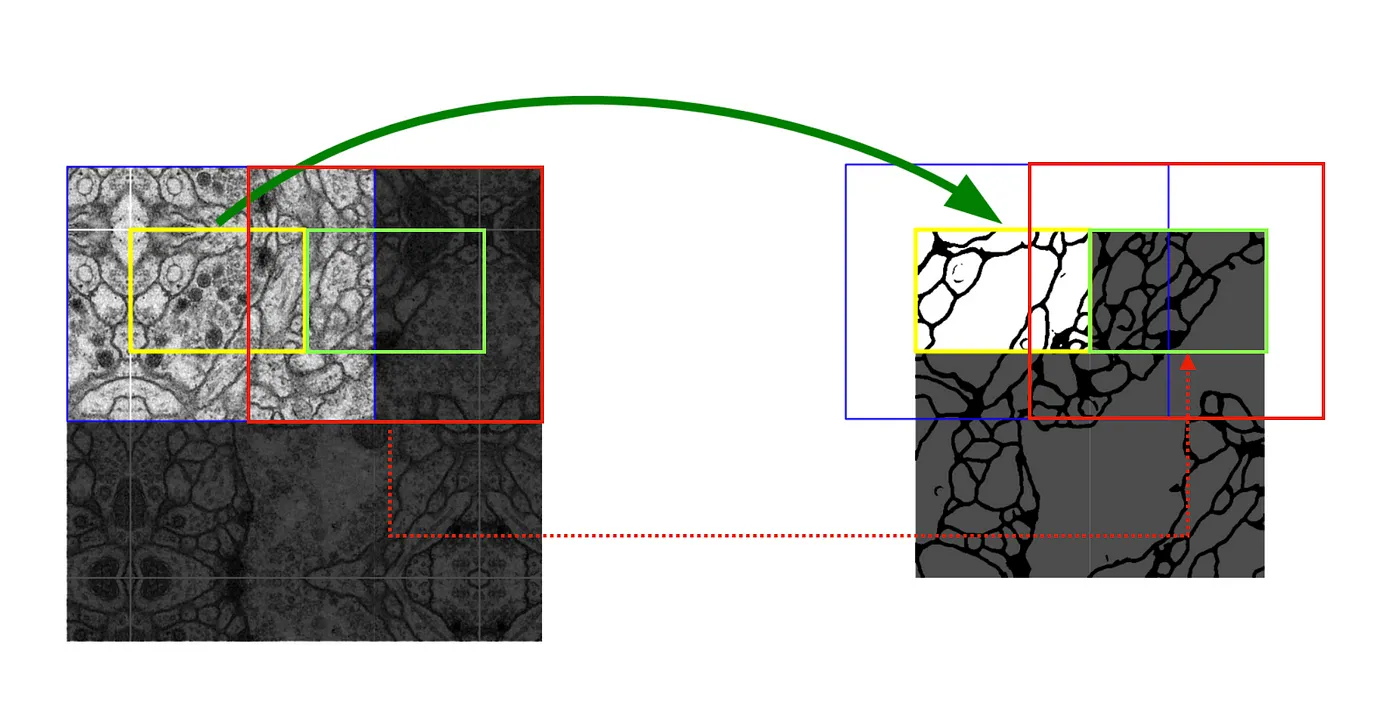
다음의 그림과 같이, 이미지 전체의 segmentation 결과를 얻기 위해서는 이전 input과 겹치는 부분을 다시 input으로 넣어주어야 한다. 이때, 각 이미지의 모서리에는 reflect padding을 해준다.
3. Training
energy function은 다음과 같이 픽셀 단위의 soft-max값으로 예측한다.
- : 위치 (2d)에서의 feature channel 의 activation값
- K: class의 갯수
loss function으로는 cross-entropy 함수가 사용된다.
Touching cells separation
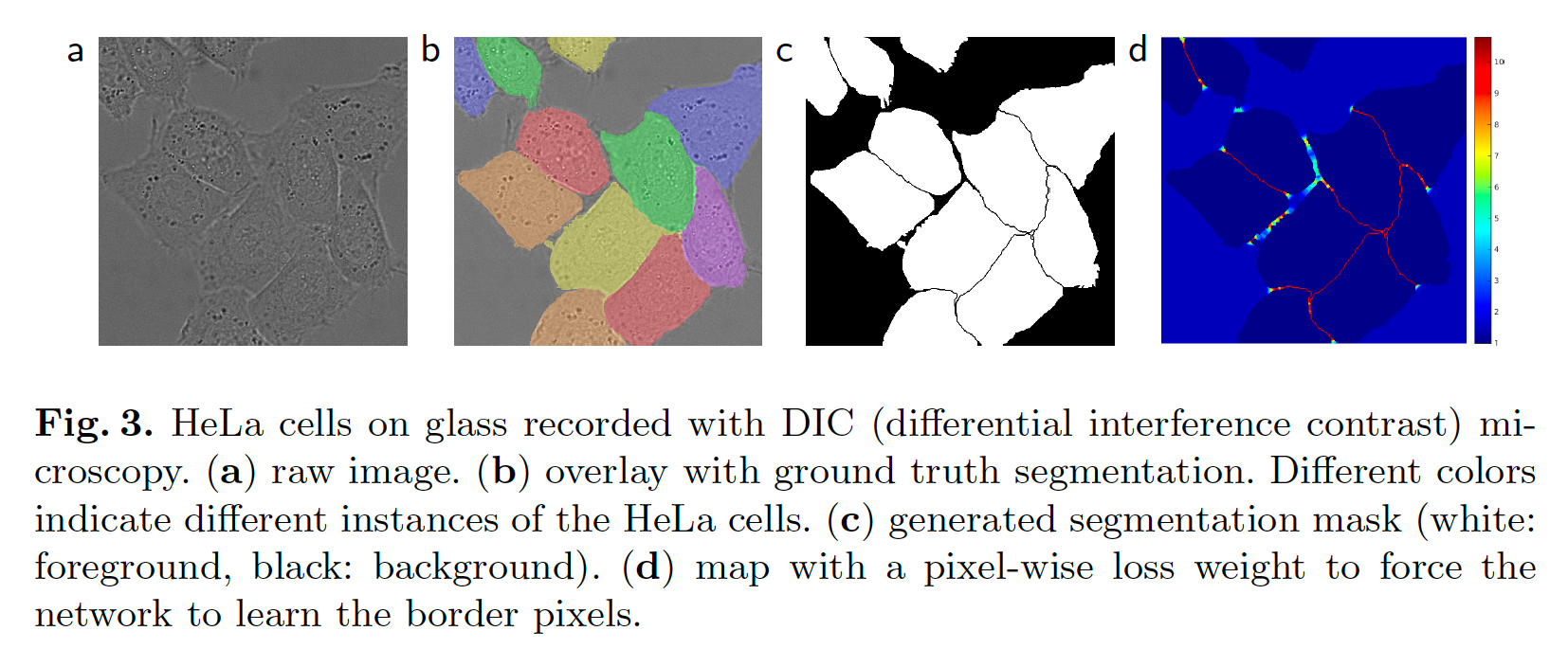
세포 사진에 대한 segmentation 작업에서 주요한 과제 중 하나는 동일한 클래스의 접촉 개체를 분리하는 것이다. 위의 이미지에서 c, d와 같이 각 세포 사이의 경계를 찾아내는 것이 중요하다.
다음과 같이 weight map을 계산해 사용한다:
- : weight map for balancing class frequencies
- : 가장 가까운 세포의 경계면까지의 거리
- : 두번째로 가까운 세포의 경계면까지의 거리
- $w_0 = 10, \sigma \sim $5 pixels
4. Experiments
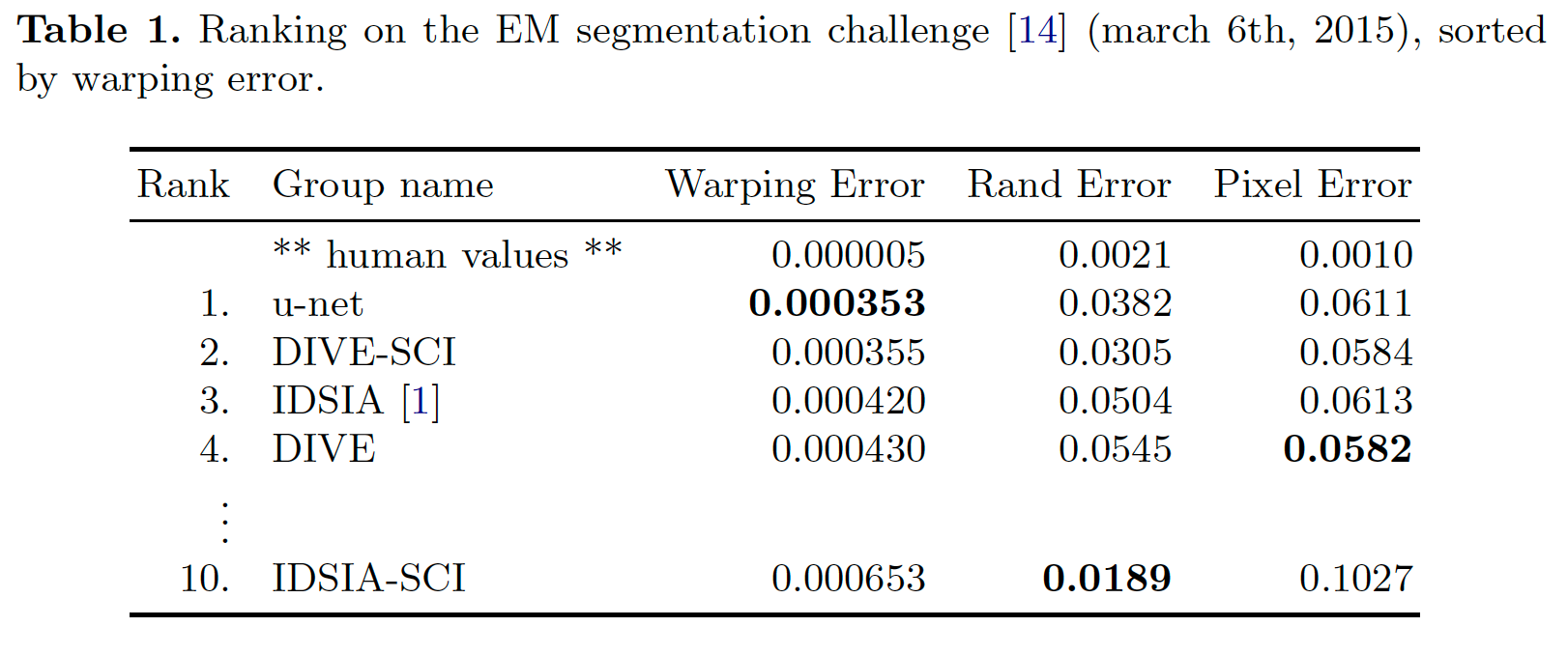
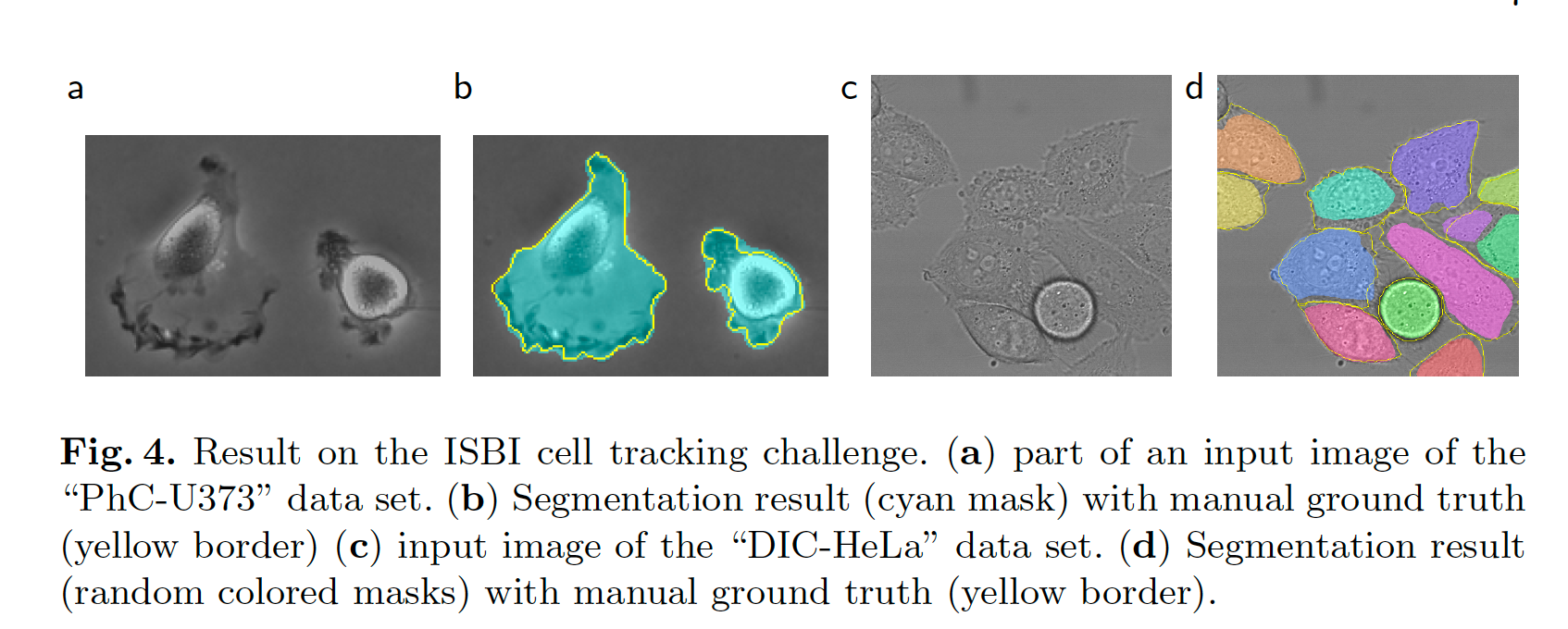
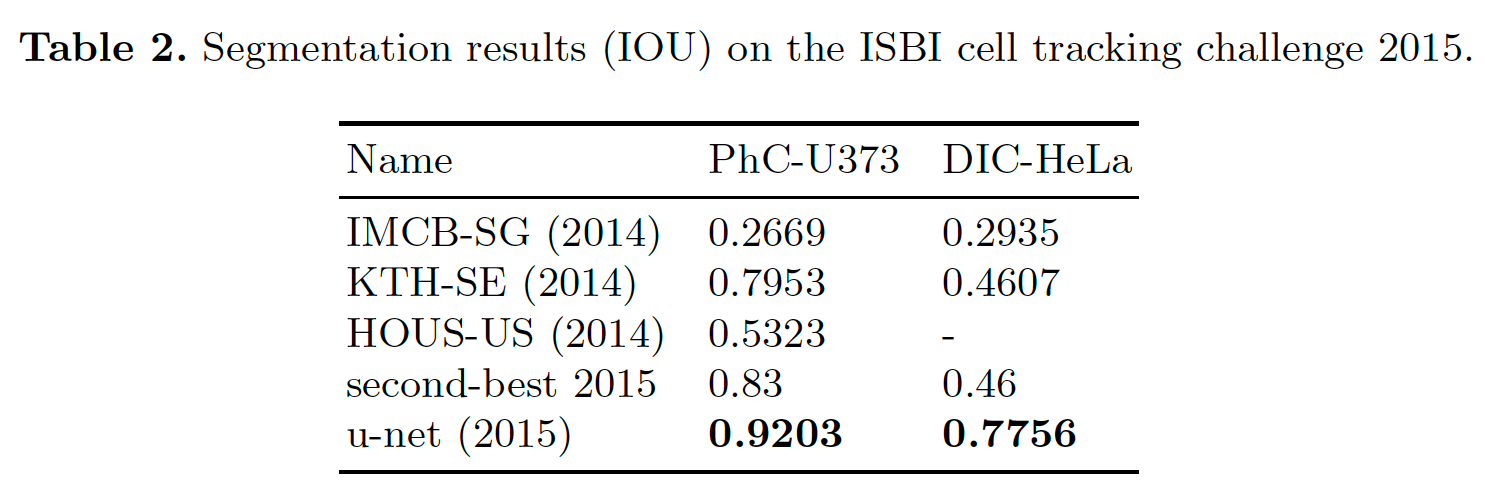
5. Conclusion
U-Net은 elastic deformation data augmentation 방식을 이용하여 biomedical segmentation 분야에서 매우 좋은 성능을 보여주고 있다.
Reference
https://medium.com/@msmapark2/u-net-논문-리뷰-u-net-convolutional-networks-for-biomedical-image-segmentation-456d6901b28a
https://gaussian37.github.io/vision-segmentation-unet/