1. 프로젝트 개요
1-1 프러젝트 주제
뼈는 신체의 구조와 기능을 유지하는 데 중요한 역할을 하며, 뼈의 정확한 분할은 의료 진단과 치료 계획을 수립하는 데 필수적이다. 특히, 손뼈는 손등뼈의 겹치는 구조로 돼 있어 분할이 더욱 까다롭다. 본 프로젝트에서는 Hand bone segmentation 모델을 개발하여 의료 영상에서 손뼈를 정확히 분할함으로써 의료 진단, 수술 계획 수립, 의료 장비 제작, 의료 교육 등 다양한 의료 응용 분야에 기여하고자 한다.
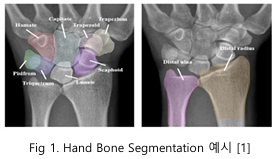
1-2 데이터 및 평가 방법
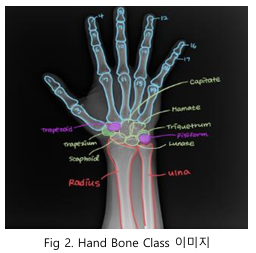
이미지 크기는 모두 2048 x 2048로 학습 이미지는 800 장, 테스트 이미지는 288장으로 이루어져 있다. 클래스는 손가락, 손등, 팔목 뼈로 구분된다. 손가락 관련 클래스는 f1부터 f19까지 총 19개가 있으며, 손등 관련 클래스는 Trapezium, Trapezoid, Capitate, Hamate, Scaphoid, Lunate, Triquetrum, Pisiform 으로 이루어진다. 팔목 뼈는 Radius 와 Ulna 로 나뉘어, 총 29개의 클래스로 구성된다.
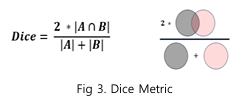
평가는 Dice Coefficient 를 사용했다. Dice Coefficient 는 두 집합 간의 유사성을 측정하는 지표로, 주로 분할된 이미지의 정확도를 평가할 때 사용된다. 이 지표의 값은 0과 1 사이를 가지며 1은 두 집합이 완벽히 일치함을, 0 은 전혀 일치하지 않음을 나타낸다.
2. 프로젝트 수행 결과
2-1. 통계 분석
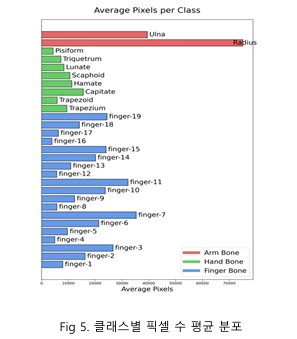
클래스별 픽셀 수 분포를 분석하여, 각 클래스의 크기를 확인했다. 이미지 내에서 팔뼈는 큰 비중을 차지하는 반면, 손등뼈와 손가락은 상대적으로 작은 비중을 보여 작은 객체로 분류했다. 작은 객체의 예측 성능 저하 문제를 해결하기 위해 다양한 해상도로 이미지를 입력하여 작은 객체를 효과적으로 구분할 방법을 고려했다.
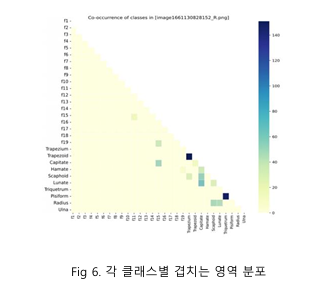
Fig 6 을 보면 손등뼈 영역에서 객체 간 겹침이 많이 발생하는 것을 알 수 있다. 이러한 겹침 영역은 예측하기 어려운 부분으로 작용한며, multi-label segmentation task 에서 성능 저하의 원인이 된다. 이를 해결하기 위해 다양한 증강 기법을 적용하여 성능 개선을 도모했다.
2-2-1. Data Cleaning
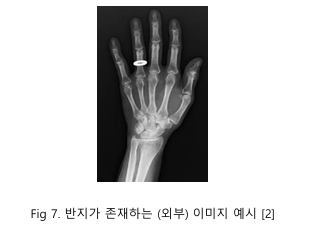
이미지 데이터를 시각화한 결과, 몇 가지 문제점을 발견하고 이를 해결했다. 먼저, Fig 7에서와 같이 Xray 이미지에 반지가 포함된 사례가 관찰되었으며, 이를 노이즈로 간주하고 해당 이미지를 제거했다. 또한, 데이터의 일관성을 유지하기 위해 동일한 쌍을 이루는 이미지를 함께 삭제하였다.
훈련 데이터의 일부에서 영역 마스킹은 적절히 수행되었으나, 잘못된 라벨이 포함된 이미지를 발견했다. 이러한 오류를 올바르게 수정했고, 이를 통해 데이터의 품질을 개선하고 클렌징하는 과정을 마무리했다.
2-2-2. Stratified K Fold with MetaData
주어진 데이터와 함께 제공된 메타 데이터 (성별, 키, 몸무게, 나이)를 활용하여 EDA를 진행한 결과, 키와 성별이 이미지 내에서 뼈가 차지하는 픽셀 수와 가장 큰 상관관계를 가지는 것으로 나타났다. 이를 바탕으로 validation set 을 구성할 때, 키와 성별에 따라 다양한 손뼈 크기를 포함하도록 조정하여 데이터 분포의 다양성을 확보했다.
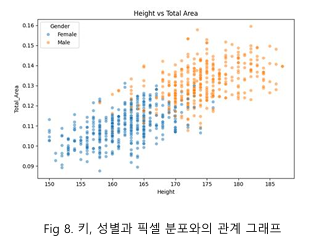
Fig 8 의 EDA 결과를 반영하여 기존 group KFold 보다 개선된 validation 데이터 세트를 생성하기 위해 stratified group K-Fold [3] 방식을 도입하였다. 성별은 두 그룹으로, 키는 4분위 수로 나누어 총 8 개의 그룹을 생성한 뒤, 각 그룹의 비율이 train set 과 validation set 에서 최대한 유사하게 유지되도록 데이터를 분할했다.
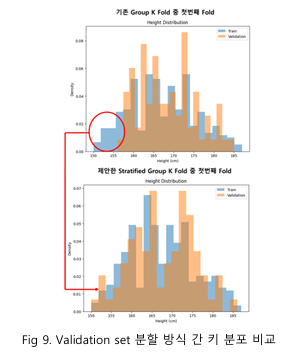
Fig 9를 보면 키 분포가 stratified group KFold에서 상당히 개선된 것을 알 수 있다. 특히, 8그룹 비율의 제곱 오차 합은 기존 group KFold에서 0.015였으나, stratified group KFold에서는 0.005로 감소하며 데이터 분포 차이가 완화되었음을 확인했다.
2-3. Baseline 모델 선정 및 분석
Table 1. Baseline 모델 성능비교
| Model | Encoder | Size | Dice |
|---|---|---|---|
| DeepLabV3+ | Xception71 | 512 | 0.9478 |
| DeepLabV3+ | EfficientNet-B4 | 512 | 0.9443 |
| YOLO11x | 512 | 0.7754 | |
| nnUNet | 512 | 0.9076 | |
| UNet | EfficientNet-B0 | 512 | 0.8808 |
| UNet3+ | HRNet-W64 | 512 | 0.9492 |
| UNet3+ | HRNet-W64 | 1536 | 0.9615 |
| UNet++ | EfficientNet-B4 | 512 | 0.9443 |
| UNet++ | ResNet101 | 512 | 0.9483 |
| UNet++ | HRNet-W64 | 1024 | 0.9692 |
Table 1을 보면 UNet++ [7]과 HRNet-W64 [8] encoder를 사용한 모델이 가장 높은 성능을 보이고, image size를 키울수록 성능이 좋아짐을 알 수 있다.
최종적으로 UNet++ 모델을 baseline 모델로 선정하였으며, HRNet-W64 encoder를 선택하였다. 빠르게 성능 관찰하기 위해 Image size 512 x 512에 대해서 실험을 진행하고, 최종적으로 1536 x 1536에 대해서 성능을 평가하도록 계획을 수립했다.
2-4. Data Augmentation
2-4-1. 기본 증강
의료 데이터는 데이터셋이 제한적인 경우가 많아, 데이터 증강을 통해 데이터 양을 확장하고 모델 성능을 향상시키는 접근법이 여러 연구에서 효과적으로 활용되고 있다 [9]. 본 프로젝트에서도 이러한 방법론을 적용하여 모델의 일반화 성능을 개선하고자 하였다.
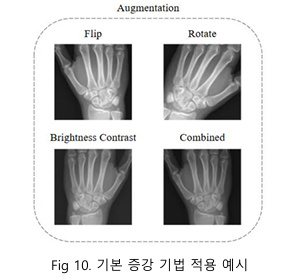
Train set에서는 원본 데이터에 데이터 증강을 통해 생성된 이미지를 더하여 데이터의 양을 두 배로 확장하였다. 증강 기법은 test set의 데이터 분포를 반영하도록 설계되었으며, 증강 데이터에는 좌우 반전을 위한 Horizontal Flip(적용 확률: 100%), Rotate(적용 확률: 80%, 적용 범위: -15° ~ 15°), Random Brightness and Contrast(적용 확률: 80%, 적용 범위: 밝기, 대비 각각 -25° ~ 25° )를 적용하였다. 이를 통해 데이터의 다양성을 확보하여 모델이 다양한 조명, 시점, 및 영상 품질에서도 강건하게 작동할 수 있도록 학습을 지원했다.
Table 2. Input data에 따른 성능 비교
| Model | Encoder | Input data | Dice |
|---|---|---|---|
| UNet++ | HRNet-W64 | Raw | 0.9533 |
| Raw + Aug* | 0.9550 |
*Aug: Augmented input data
Table 2를 보면train set에 원본 이미지만을 사용한 경우보다 증강 이미지를 추가하여 데이터셋을 확장했을 때, 모델 성능이 향상되는 것을 알 수 있다. 이는 증강된 데이터가 학습 과정에서 더 다양한 특성을 반영하도록 돕고, 모델의 일반화 성능을 강화하는 데 기여했음을 보여준다.
2-4-2. Gamma Correction
기본 증강을 통해 모델의 성능을 개선한 이후, 겹치는 손목뼈 영역에서 성능을 더욱 향상시키기 위해 GC(gamma correction) 증강 기법을 적용했다. [10] 감마 보정은 의료 영상 분할 분야에서 효과적인 데이터 증강 기법으로 널리 사용되고 있으며 [11] 본 프로젝트에서도 이를 활용하여 dice 점수 향상을 도모하였다. 특히, 성능이 상대적으로 낮았던 손목뼈 영역의 검출 정확도를 높이기 위해 추가적으로 CLAHE 증강 기법[12]을 적용했다.
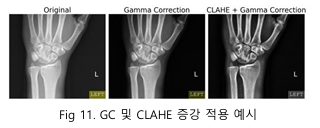
초기 실험에서는 별도의 증강 없이 ResNet101을 encode로 사용하는 UNet++ 모델을 학습시켰으며, 이때의 Dice 점수는 0.9483이었다. 이는 데이터 증강 없이도 모델이 일정 수준의 학습 성능을 발휘할 수 있음을 보여주었다.
Table 3. 증강 기법에 따른 성능 비교
| Model | Encoder | Augmentation | Dice |
|---|---|---|---|
| x | 0.9483 | ||
| UNet++ | ResNet101 | GC + CLAHE | 0.9513 |
| GC | 0.9530 |
*GC: Random gamma correction
Table 3을 보면 gamma correction만을 단독으로 적용한 실험에서는 gamma_limit=(80, 200)과 p=0.3의 확률로 명암을 조정한 결과 Dice 점수가 0.9530으로 증가한 것을 알 수 있다. 하지만 CLAHE와 gamma correction을 함께 적용하여 명암 대비를 조정한 결과, dice 점수가 0.9513 으로 gamma Correction 만을 단독으로 적용했을 때보다 소폭 하락하였다. 이는 CLAHE 적용 과정에서 이미지 윤곽선이 과도하게 강조되며 발생한 노이즈 증가로 인해 모델 학습이 방해받았을 가능성이 있음을 시사한다. Gamma correction 단독 사용은 데이터의 명암 분포를 효과적으로 조정하여 모델 성능을 개선하는 데 기여하였다. 이후 실험에서 감마 보정의 범위를 넓게 설정하고 적용 확률을 낮추는 방식으로 추가 조정을 진행한 결과, 가장 안정적이고 효과적인 성능 향상을 확인할 수 있었다.
2-5. Loss
Hand Bone Segmentation 에서 성능을 최적화 하기 위해 다양한 손실 함수에 대해 실험했다.
본 실험에서는 UNet++ 모델을 사용하였으며, 입력 이미지 크기는 512 x 512로 고정하였다. 각 손실 함수가 모델 성능에 미치는 영향을 비교하고, 최적의 손실 함수를 선정하기 위해 다른 조건을 고정하고 분석을 진행했다.
Table 4. 손실함수에 따른 성능 비교
| Model | Size | Loss | Dice |
|---|---|---|---|
| 512 | BCE | 0.9533 | |
| Focal | 0.9539 | ||
| UNet++ | BCE + IOU | 0.9544 | |
| Hybrid* | 0.9548 | ||
| 1536 | BCE | 0.9713 | |
| BCE + IOU | 0.9733 |
*Hybrid: Focal + MS-SSIM + IOU
먼저, 기본적인 이진 교차 엔트로피(BCE) 손실함수를 사용하여 실험을 진행하였다. BCE는 예측 확률과 실제 라벨 간의 픽셀별 차이를 측정하는 방식으로, 이진 분류 작업에 적합한 특성을 가지고 있다. 이 손실 함수를 사용한 결과, dice score는
0.9533 으로 나타나 양호한 성능을 보였다.
다음으로, Focal Loss 를 적용하였다. Focal Loss는 어려운 예측에 더 높은 가중치를 부여하는 방식으로, 주로 불균형 데이터셋에서 효과적이다. 그러나, 이 손실 함수를 적용한 결과 dice score는 0.9539 로 BCE 보다 미세하게 높은 수준에 그쳤다. focal loss 가 어려운 예측에 집중할 수 있도록 도와주었지만, 전체적인 성능 향상에는 큰 차이를 보이지 않았다.
이후, BCE Loss와 IOU Loss를 결합한 손실 함수를 적용하였다. IOU Loss는 겹치는 구조를 다루는 데 효과적이며, 특히 손등뼈와 같은 겹치는 부분을 분할하는 데 유리할 것으로 예상되었다. 실험 결과, dice score 는 0.9544 로 BCE 와 Focal Loss 보다
높은 성능을 기록하였다. 이는 IOU Loss가 겹치는 영역을 더 잘 처리하여 성능을 향상시켰음을 보여준다.
마지막으로, Focal Loss, IOU Loss, MS-SSIM 을 결합한 하이브리드 손실 함수(Hybrid Loss)를 실험에 적용하였다. MS-SSIM은 이미지의 구조적 유사도를 측정하는 손실 함수로, 시각적으로 더 자연스러운 결과를 도출할 수 있도록 돕는다[5]. 이 하이브리드 손실 함수를 적용한 결과, dice score는 0.9548 로 가장 높은 성능을 기록하였다.
그러나 이 방법은 메모리 부족 문제와 학습 시간 증가를 초래하였다. 특히, 이미지 크기를 1536 x 1536 으로 확대했을 때 메모리 부족 현상이발생하였으며, MS-SSIM 이 많은 메모리와 연산 자원을 소모하는 것으로 분석되었다.
결국, 해상도를 유지하는 것이 중요한 상황에서 BCE + IOU 조합이 가장 안정적이고 효율적인 성능을 발휘하였다. BCE 는 초기 학습에서 수렴 속도가 빠르며, IOU 는 손등 뼈와 같은 겹치는 구조를 처리하는 데 유리한 특성을 보인다. 이러한 이유로, BCE + IOU 손실 함수 조합이 최종적으로 가장 적합한 손실 함수로 선택되었다.
2-7. 최종 모델 선정 및 분석
본 프로젝트에서는 손뼈 세그멘테이션을 위한 최적의 딥러닝 모델을 개발하고, 이를 고해상도 의료 영상에 적용하여 실용적인 결과를 도출하였다. 손뼈 구조는 복잡하고 세밀한 특징을 가지며, 이를 효과적으로 분할하기 위해 여러 모델과 학습 전략을 비교한 결과, U-Net++ 모델이 가장 우수한 성능을 보였다. 고해상도 이미지를 처리하는 과정에서 원본 해상도인 2048 × 2048 이미지를 1536 × 1536 으로 리사이즈하여 처리함으로써, 세부 정보를 충분히 유지하면서도 학습 속도와 메모리 사용량 간의 균형을 잘 맞출 수 있었다.
데이터 증강 기법으로는 원본 데이터셋에 감마 보정을 적용하여 조명 조건의 변화에 강건한 모델을 학습했다. 원본 데이터셋을 복사한 후, 감마 보정뿐만 아니라 다양한 데이터 증강 기법을 추가로 적용하여 두 개의 데이터셋을 결합하였다. 이를 통해 한 에포크에 사용되는 데이터를 두 배로 확장할 수 있었으며, 다양한 조명 조건과 영상 품질에서 모델이 강건하게 동작할 수 있도록 학습시킬 수 있었다.
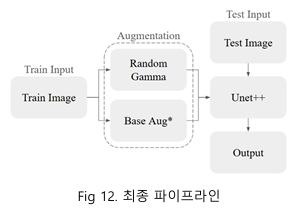
손실 함수로는 Binary Cross Entropy(BCE)와 Log IoU 를 결합하여 사용하였다. BCE는 분할된 결과와 실제 레이블 간의 오차를 측정하는 데 유용하며, IoU 는 세그멘테이션 마스크와 실제 레이블 간의 겹치는 부분에 대한 정확도를 높이는 데 효과적이었다. 이 손실 함수 조합은 뼈 구조와 같은 복잡한 패턴을 잘 학습하는 데 적합한 성능을 보여주었다.
모델 평가 결과, 제안된 방법은 기존의 기본 UNet 모델에 비해 고해상도 이미지를 처리할 때 더 정밀한 세그멘테이션 성능을 보였으며, 손 뼈의 주요 구조를 효과적으로 분할할 수 있음을 확인하였다.
Table 5. 최종 성능표
| Model | Size | Aug | Loss | Dice |
|---|---|---|---|---|
| UNet++ (baseline) | 512 | raw | BCE | 0.9533 |
| UNet++ (ours) | 1536 | GC + Base Aug | BCE + IOU | 0.9733 |
위 테이블를 보면 최종적으로 baseline 에서 우리의 증강 기법과 새로운 loss 등을 적용하였을 때, dice score 0.9533 에서 0.9733 으로 2.10% 성능 향상을 이뤄냈음을 알 수 있다.
추가 시도
추가로 나는 nnU-Net를 연구해 보았다.
nnU-Net 는 Papers with Code 의 'Medical Segmentation Decathlon' 리더보드에서 dice 점수를 기준으로 3 위를 차지한 모델이다. 또한 2024 년 4 월까지 업데이트가 이루어진 최신 모델이다.
nnUNet 은 SMP 라이브러리에서 직접적으로 지원되지 않기 때문에 nnUNet 라이브러리를 설치하여 구현했다. 초기 구성에서는 Dice Loss와 Cross-Entropy Loss 의 조합과 함께 2D U-Net 을 백본으로 사용했다. dice score 는 0.9072 로 UNet++보다 낮았기 때문에 baseline 모델로 활용되기엔 부족했다.
성능이 낮았던 주된 이유는 softmax 를 활성화 함수로 사용하는 multi-class segmentation방식을 사용했기 때문이다. 이 방식은 각 픽셀이 단일 클래스에만 속할 수 있어, 각 픽셀이 여러 클래스를 가질 수 있는 multi-label segmentation task인 이번 프로젝트에선 dice score 가 낮게 나온것으로 판단된다.
이러한 제한 사항을 해결하기 위해 활성화 함수가 softmax 에서 sigmoid 로 수정하여 multi-label segmentation 이 가능하도록 개선했다. 또한 CrossEntropyLoss 대신 BCE Loss 를 활용 하여 각 클래스에 대한 독립적인 손실 계산이 가능해졌다.
최종적으로 성능이 개선 되었지만, UNet 을 backbone 으로 사용하는 한계점으로 UNet++보다 낮은 성능을 기록해 baseline 모델로 선정되진 못했다. 시간이 있었다면, backbone을 최신 모델로 변경하고, 모델 구조를 개선하여 baseline 모델로 사용해도 좋았을 것 같다.
Reference
[1] Bo-Kyeong Kang, et al., "Automatic Segmentation for Favourable Delineation of Ten Wrist Bones on Wrist Radiographs Using Convolutional Neural Network", Journal of Personalized Medicine, MDPI, 2022.
[2] Keisuke Izumi, et al., “Ensemble detection of hand joint ankylosis and subluxation in radiographic images using deep neural networks”, scientific reports, 2024.
[3] StratifiedGroupKFold https://scikitlearn.org/dev/modules/generated/sklearn.model_selection.StratifiedGroupKFold.html
[4] Segmentation- Models-PyTorch(SMP) https://smp.readthedocs.io/en/latest/
[5] Huimin Huang, et al., “UNet 3+: A Full-Scale Connected UNet for Medical Image Segmentation”, Computer Vision and Pattern Recognition, 2020
[6] Fabian Isensee, et al., “nnU-Net Revisited: A Call for Rigorous Validation in 3D Medical Image Segmentation”, Lecture Notes in Computer Science, 2024
[7] Zongwei Zhou, et al., “UNet++: A Nested U-Net Architecture for Medical Image Segmentation”, Computer Vision and Pattern Recognition, 2018
[8] Jingdong Wang, et al., “Deep High-Resolution Representation Learning for Visual Recognition Publisher: IEEE Cite This PDF”, IEEE, 2021
[9] Mingyu Kim, et al., “Data Augmentation Techniques for Deep Learning-Based Medical Image Analyses”, Journal of the Korean Society of Radiology, 2020
[10] Shanto Rahman, et al., “An adaptive gamma correction for image enhancement”, EURASIP Journal on Image and Video Processing, 2016
[11] Ili Ayuni Mohd Ikhsan, et al., “An analysis of xray image enhancement methods for vertebral bone segmentation”, IEEE, 2014
[12] Khan, Sajid Ali, et al., “Contrast Enhancement of Low-Contrast Medical Images Using Modified Contrast Limited Adaptive Histogram Equalization”, American Scientific Publishers,
