데이터셋 출처
- Pima Indians Diabetes Database | Kaggle
- https://scikit-learn.org/stable/modules/generated/sklearn.datasets.load_diabetes.html
Feature Engineering
다양한 옵션을 지정하여 모델의 성능을 개선해볼 수도 있지만 feature engineering을 해본다.
수치의 범위가 넓으면 수치형 변수의 조건이 너무 세분화되어 tree가 깊어진다.
Pregnancies
df["Pregnancies_high"] = df["Pregnancies"] > 6
df[["Pregnancies", "Pregnancies_high"]].head()
pregnancies_high 변수만 추가해 줬는데도 과적합이 나타나지 않고 정확도가 상승.
Age
# One-Hot-Encoding
# 수치 => 범주 => 수치
df["Age_low"] = df["Age"] < 30
df["Age_middle"] = (df["Age"] >= 30) & (df["Age"] <= 60)
df["Age_high"] = df["Age"] > 60
df[["Age", "Age_low", "Age_middle", "Age_high"]].head()
feature engineering을 한다고 해서 성능이 무조건 오르는건 아니다. 하나씩 해보면서 평가해보자.
결측치
당뇨병예측인데 Insulin, BMI, Glucose 등 0이 나올수 없는 값들은 결측치가 된다.
df["Insulin_nan"] = df["Insulin"].replace(0, np.nan)
df[["Insulin", "Insulin_nan"]].head()
df["Insulin_nan"].isnull().sum() # 374# 결측치 비율
df["Insulin_nan"].isnull().mean() # 0.4869791666666667평균값으로 대체
df.groupby(["Outcome"])[["Insulin", "Insulin_nan"]].agg(["mean", "median"])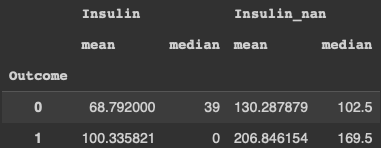
# 결측치 채우기
df.loc[(df["Outcome"] == 0) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 68.7
df.loc[(df["Outcome"] == 1) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 100.3중앙값으로 대체
# 결측치 채우기
df.loc[(df["Outcome"] == 0) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 102.5
df.loc[(df["Outcome"] == 1) & (df["Insulin_nan"].isnull()), "Insulin_nan"] = 169.5정규분포 만들기
한 쪽에 데이터가 몰려있으면 학습을 할 때 어려움이 있을 수 있다.
sns.distplot(df.loc[df["Insulin"] > 0, "Insulin"])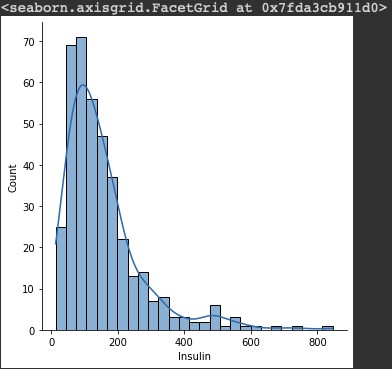
그래프를 그려보면, 현재 데이터는 한쪽에 치우쳐져 있고 뾰족한 모양이다.
왜도는 치우친 정도, 첨도는 뾰족한 정도를 나타냄.
Insulin_log = np.log(df.loc[df["Insulin"] > 0, "Insulin"] + 1)
# sns.distplot(Insulin_log)
# seaborn 0.11.0 이상
sns.displot(Insulin_log, kde=True)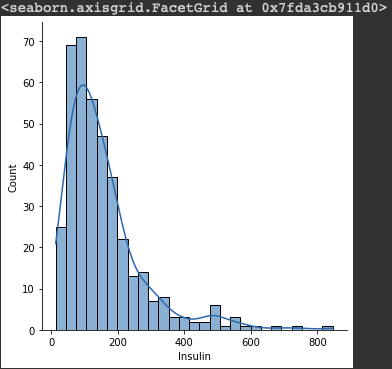
log변환을 하면 정규분포와 비슷한 모양을 띈다.
0 이하에서는 음의 무한대 값에 수렴하므로 +1 를 해준다.
sns.displot(df, x="Insulin_nan", kde=True)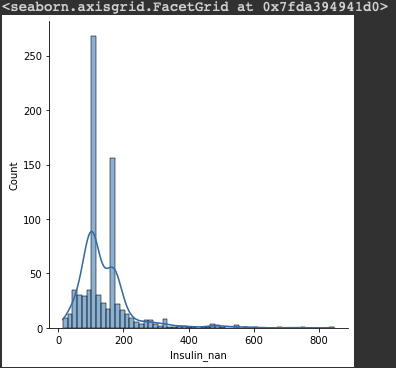
중앙값으로 채워줬기 때문에 값이 뾰족하게 튀어나와 있다.
df["Insulin_log"] = np.log(df["Insulin_nan"] + 1)
sns.displot(df, x="Insulin_log", kde=True)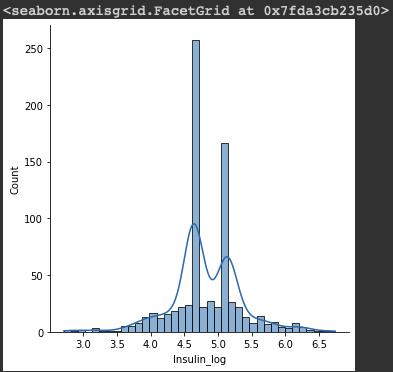
마찬가지로 log 변환해주기
파생변수 만들기
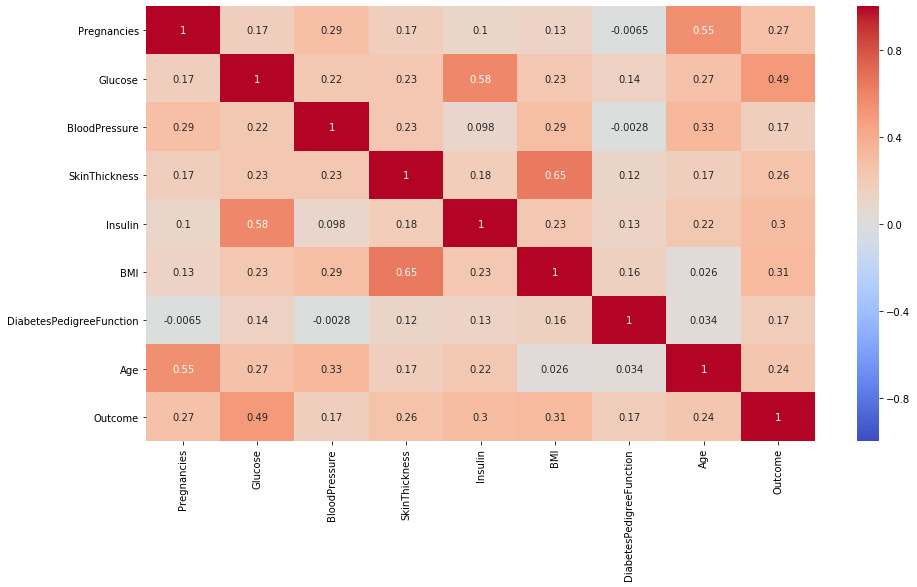
skin_thickness와 BMI 지수가 상관성이 높다.
Insulin과 Glucose가 상관관계가 있는지 알아보자.
Insulin & Glucose
sns.lmplot(data=df, x="Insulin_nan", y="Glucose", hue="Outcome")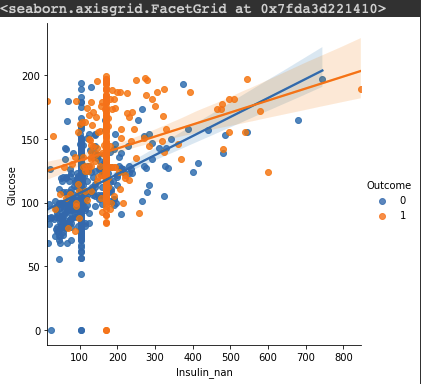
df["low_glu_insulin"] = (df["Glucose"] < 100) & (df["Insulin_nan"] <= 102.5)
df["low_glu_insulin"].head()
인슐린과 글루코스의 상관계수로 파생변수 log_glu_insulin을 만들어봤다.
pd.crosstab(df["Outcome"], df["low_glu_insulin"])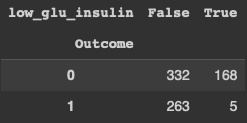
Outcome과 log_glu_insulin의 관계를 살펴보면,
log_glu_insulin이 True인 값의 발병 확률은 매우 낮다.
이상치 다루기
이상치를 확인하기 위해 상자수염그림을 그린다.
plt.figure(figsize=(15, 2))
sns.boxplot(df["Insulin_nan"])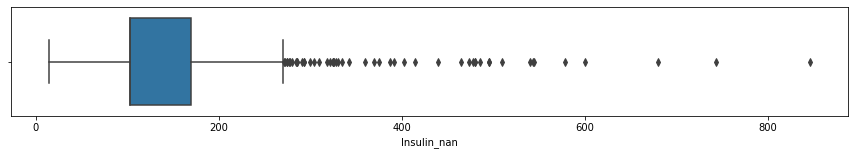
df["Insulin_nan"].describe()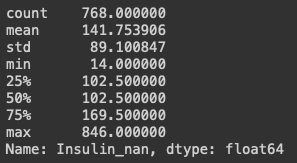
25%와 50%가 같은 이유는 null 값을 중앙값으로 채워줬기 때문입니다.
max 값이 크기 때문에 평균값이 높아 보입니다.
IQR값을 구합니다.
IQR3 = df["Insulin_nan"].quantile(0.75)
IQR1 = df["Insulin_nan"].quantile(0.25)
IQR = IQR3 - IQR1
IQR # 67.0OUT = IQR3 + (IQR * 1.5)
OUT # 270.0df[df["Insulin_nan"] > OUT].shape # (51,16)51개의 데이터가 이상치인데 이걸 제거하기에는 너무 많다.
따라서, 600이상이 되는 데이터를 제거한다는 결정을 내릴 수 있다.
데이터를 제거해 줄때는 train 데이터셋에서만 제거해야 한다.
train = df[df["Insulin_nan"] > 600].shape # (3,16)피처 스케일링
- https://scikit-learn.org/stable/modules/preprocessing.html#standardization-or-mean-removal-and-variance-scaling
- https://scikit-learn.org/stable/modules/generated/sklearn.preprocessing.StandardScaler.html
- https://scikit-learn.org/stable/modules/generated/sklearn.preprocessing.MinMaxScaler.html
- https://scikit-learn.org/stable/modules/generated/sklearn.preprocessing.RobustScaler.html
숫자의 범위가 다르면 feature별로 비중이 다르게 계산될 수 있으므로 스케일링 기법을 사용하기도 합니다.
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(df[["Glucose", "DiabetesPedigreeFunction"]])
scale = scaler.transform(df[["Glucose", "DiabetesPedigreeFunction"]])
scale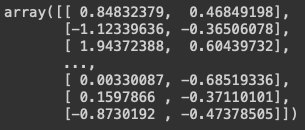
fit을 하고, transform하면 숫자가 변형됩니다.
스케일링한 값을 변수에 담아줍니다. 크게 데이터가 달라지지 않았습니다.
df[["Glucose", "DiabetesPedigreeFunction"]] = scale
df[["Glucose", "DiabetesPedigreeFunction"]].head()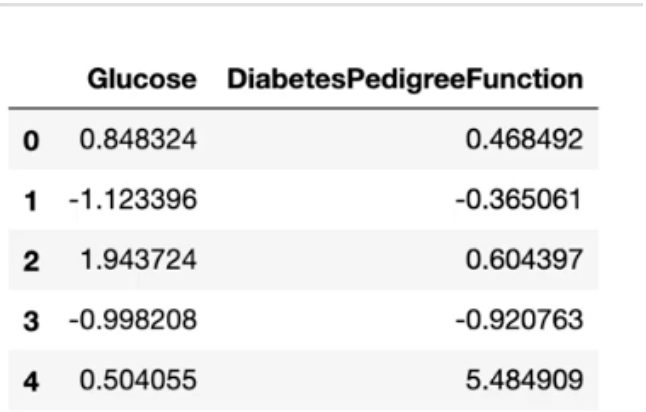
h = df[["Glucose", "DiabetesPedigreeFunction"]].hist(figsize=(15, 3))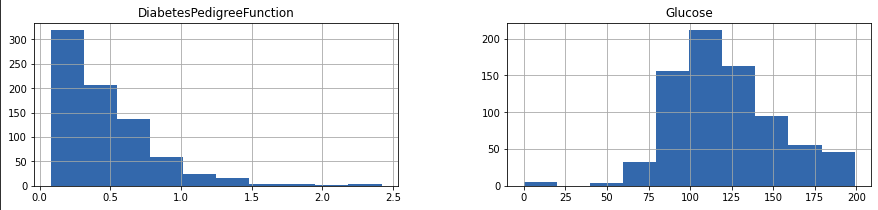
이 데이터셋에서는 스케일링이 크게 의미가 없지만 다른 프로젝트에서는 예측의 정확도를 높일 수 있습니다.
전처리는 EDA를 먼저 진행해 인사이트를 얻은 후 진행하는 것이 좋습니다.
