OPEN CV를 활용한 image blur 필터
Image blur
- 이미지의 공간에서 특정 부분의 노이즈를 지워주기 위한 목적으로 사용함, 예를 들면 얼굴에 있는 잡티 같이 점단위로 튀는 지점을 보정 해주는 목적
- 그 외 2D FFT로 역변환을 진행할 시 aliasing 현상을 제거 해주기 위한 목적으로 사용함
- blur라는 기술은 이미지 밝기 분포를 특정 지점으로 평준화 시켜준다는 개념으로 접근하는게 좋음
Opencv로 커널 연산
filter2D()
dst = cv2.filter2D(src, ddepth, kernel[, dst[, anchor[, delta[, borderType]]]])
dst입력 이미지와 동일한 크기와 채널을 가지는 이미지 행렬입력으로는 아래 3가지가 요구 된다.
1.src입력 이미지
2.ddepth출력 이미지의 채널
3.kernel컨볼루션을 진행할 커널, filter2D() 에서는 모든채널에 동일한 커널이 적용 되기 때문에 채널마다 다르게 적용하기 위해서는 채널마다 따로 filter2D 연산을 해줘야 함추가 옵션
1.anchor필터 포인트와 커널간의 오프셋 값 조절로 (-1,-1)이 기본값으로 되어있음. (-1,-1)은 커널의 중심지점
2.delta출력 이미지의 보정값으로 출력 결과에 상수를 더해줌
3.borderTypepixel extrapolation method.
Note: 일반적으로 추가 옵션은 잘 안씀OpenCV Documentation
filter2D()를 활용한 mean kernel 연산
- Mean kernel이란 연산지점이 되는 포인트와 인접구간의 평균치를 활용하여 값을 평준화 하는 방법
- 대표적인 3*3 mean kernel은 아래와 같다.
파이썬으로 구현
- module import
import cv2
import numpy as np
from matplotlib import pyplot as plt
Ham_image = cv2.imread('AdobeStock_347974747_438127_reduced.jpg',cv2.IMREAD_COLOR)
plt.imshow(Ham_image[:,:,::-1])
- 커널 생성 및 이미지 출력
kernel5 = np.ones((5,5)) / (5**2)
ham_avg_filter5 = cv2.filter2D(Ham_image,-1,kernel5)
plt.imshow(ham_avg_filter5[:,:,::-1]); 
- 이를 활용하여 커널 개수를 늘려보면 아래와 같이 표현이됨

- 커널의 행렬이 커질 수록 세부적인 디테일이 사라지는것이 확인이 됨
GaussianBlur를 활용한 이미지 평준화
-
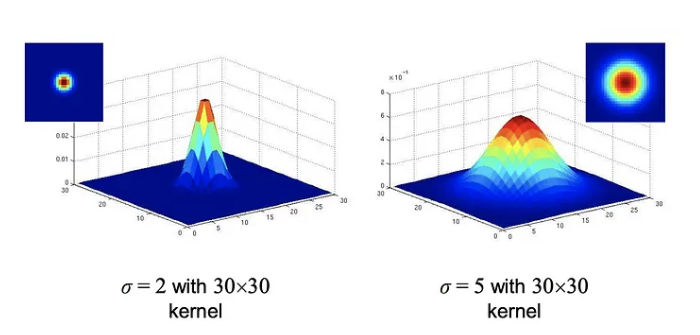
Gaussain커널은 각 지점 마다 가중치를 주어 이미지의 원점과 멀어질수록 값의 가중치를 낮추는 방법
-
mean filter를 활용하면서 발생하는 이미지의 정보를 소실을 방지하기 위해서 사용함
-
대표적인 가우시안 커널의 일반식은 다음과 같다.
- sigma는 정규분포를 의미
- 정규 분포의 값의 크기가 클수록 인접한 커널의 값이 커지게 됨
이미지 출처
Function Syntax
dst = cv2.GaussianBlur(src, ksize, sigmaX[, dst[, sigmaY[, borderType]]])
dst: 출력 이미지로 입력 이미지와 동일한 크기와 자료형을 가지게 됨3개의 입력이 필수로 필요함
src입력 이미지로, 모든 채널에 대해서 독립적으로 연산을 진행함, 그러나 이미지의 자료형은 CV_8U, CV_16U, CV_16S, CV_32F, CV_64F을 사용 해야됨ksize커널의 크기로 홀수 값을 지정해야 함sigmaXX방향으로의 표준편차
Optional arguments include:
sigmaY: Y방향으로의 표준편차로 0이 입력되면 SigmaX와 동일 한 값을 입력으로 사용함borderType이미지 외곽영역의 보간 법 지정
OpenCV Documentation
파이썬을 활용한 Gaussian kernel 구현
import cv2
import numpy as np
from matplotlib import pyplot as plt
Ham_image = cv2.imread('AdobeStock_347974747_438127_reduced.jpg',cv2.IMREAD_COLOR)
ham_avg_gauss5 = cv2.GaussianBlur(Ham_image,(5,5),5,5)
ham_avg_gauss10 = cv2.GaussianBlur(Ham_image,(9,9),5,5)
ham_avg_gauss25 = cv2.GaussianBlur(Ham_image,(25,25),5,5)
plt.figure(figsize = [100,20])
plt.subplot(4,1,1)
plt.imshow(Ham_image[:,:,::-1]);plt.axis('off');plt.title('original')
plt.subplot(4,1,2)
plt.imshow(ham_avg_gauss5[:,:,::-1]);plt.axis('off');plt.title('Gaussain Kernel 5*5')
plt.subplot(4,1,3)
plt.imshow(ham_avg_gauss10[:,:,::-1]);plt.axis('off');plt.title('Gaussain Kernel 9*9')
plt.subplot(4,1,4)
plt.imshow(ham_avg_gauss25[:,:,::-1]);plt.axis('off');plt.title('Gaussain Kernel 25*25')
- mean커널에 비해서 이미지의 포인트 지점들이 보정이 되는것을 확인이 가능함
Mean kernel과 Gaussian kernel의 비교
- 위의 이미지를 한번에 출력 하여 보면 다음과 같다

- 가장 차이가 심한 커널을 비교하여 히스토그램을 보면 아래와 같은 특성을 볼 수 있음
Ham_image_HSI = cv2.cvtColor(Ham_image,cv2.COLOR_BGR2HSV)
ham_avg_gauss25_HSI = cv2.cvtColor(ham_avg_gauss25,cv2.COLOR_BGR2HSV)
ham_avg_avg25_HSI = cv2.cvtColor(ham_avg_filter25,cv2.COLOR_BGR2HSV)
hist_original =cv2.calcHist(images = [Ham_image_HSI],
channels = [2],
mask = None,
histSize = [256],
ranges = [0,255])
hist_gauss =cv2.calcHist(images = [ham_avg_gauss25_HSI],
channels = [2],
mask = None,
histSize = [256],
ranges = [0,255])
hist_avg =cv2.calcHist(images = [ham_avg_avg25_HSI],
channels = [2],
mask = None,
histSize = [256],
ranges = [0,255])
plt.plot(hist_original,label='Original');plt.xlim([0,256]);plt.yscale('log')
plt.plot(hist_avg,label='Average Kernel 25*25');plt.xlim([0,256]);plt.yscale('log')
plt.plot(hist_gauss,label='Gaussian Kernel 25*25');plt.xlim([0,256]);plt.yscale('log')
plt.title('Kernal compairison of intensity scale')
plt.legend()
plt.show()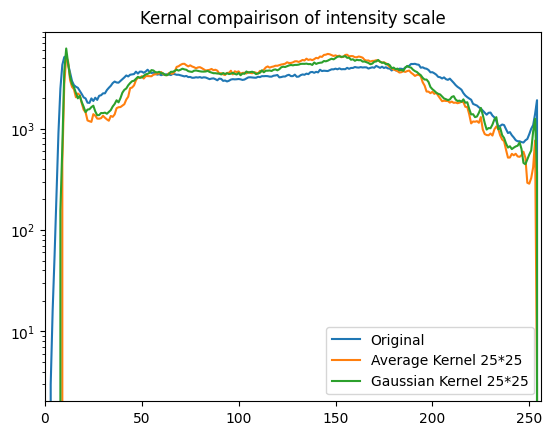
- 두 커널 전부 공통적으로 이미지의 중간지점(평균값)으로 값이 많이 분포가 되는것을 확인이 가능함
- 허나 gaussian kernel이 Mean kernel과 대비해서 엣지영역에서의 값 손실이 적음