[2022 MICCAI] Unsupervised Nuclei Segmentation using Spatial Organization Priors
Paper Review
디지털 병리에서 병리학자들이 IHC 염색 슬라이드로 다양한 바이오마커들 (Ki-67, HER2, CD3/CD8)을 분석하는데, 환자 biospy에서의 바이오마커의 식별은 치료 요법을 설계할 수 있도록 해준다. 그런데, 그들의 이미지 종류의 다양성과 특이성으로 인해 주석이 있는 데이터베이스를 사용하는 경우가 드물다. 결과적으로, 임상 셋팅에서 강건하고 효율적인 학습을 기반한 진단 시스템이 개발되고 적용되기가 어렵다.
이 연구는 다른 염색 프로토콜에 걸쳐 관찰되는 세포 조직의 전체적인 구성과 구조가 비슷하다고 보고 IHC 이미지에서의 비지도학습 nuclei segmentation을 수행하기 위해 H&E 염색 데이터베이스의 광범위한 가용성과 조직의 구성과 구조의 불변성을 모두 활용할 것을 제안한다. 공개도니 H&E 염색의 nuclei mask들과 비교를 거쳐 high-level nuclei 분포 priors들에 의존하는 적대적 생성 방법을 구현하고 평가한다.
1. Introduction
IHC 염색 조직 이미지에 대해 nuclei segmentation 모델을 학습시키는 것은 어려운 문제다. IHC 이미지는 immunostains를 사용해서 사람 눈에 안 보이는 nuclei의 표면, 세포막, 세포질에 있는 단백질을 판독하는 데에 활용된다. IHC는 비싸고 시간이 많이 걸리는 유전자 검사를 우회할 수 있기 때문에, 특히 cancer pathology에서 진단과 치료방법 선택 등에 널리 쓰인다.
IHC 이미지에서 핵을 자동으로 식별하고 분할하는 기능은 (i) 암 진단 시간을 단축하고, (ii) 일상적인 병리학에서 오진을 줄이며, (iii) 치료 반응 예측을 위한 세포 기반 학습 시스템의 성능을 개선할 수 있기 때문에 매우 중요합니다.
가장 유명한 nuclei segmentation 방법들은 최근 수작업으로 신중하게 얻어진 nuclei의 pixel-based annotation에 의존한다.
하지만 그 annotation을 하는 건..?
time-consuming, cumbersome, tedious and error-prone..
시간 오래 걸리고, 번거롭고, 지루하고, 오류가 생기기 쉽고.. yeah yeah
이게 광범위한 IHC 대한 분할 모델의 개발을 방해한다
이러한 문제를 완화하기 위한 semi-supervised 방법도 제안되었었는데, 여전히 whole slide 수준에서 사용하는 수작업으로 상호작용하는 것이 많은 시간 소모를 요구하게 된다. 반면에 color clustering 기반의 unsupervised segmentation 방법들은 성능이 안 좋아서 적용되기 힘들다.
이 연구는 간단한 아이디어로 해결할 수 있는 방법을 소개한다.
조직 내의 세포들의 공간적 구성과 모양 특징들은 병리 조직 슬라이드를 염색하기 위해 사용되는 염색 기법의 종류에 따라 크게 바뀌지 않는다는 사실을 exploit한다. 이미 대중에게 공개된 H&E 염색의 nuclei annotation을 활용해 잠재적으로 다양한 type의 immunostains에 대해서 segmentation 모델을 학습할 수 있는 강력하고 versatile (변하기 쉬운) adversarial-based 방법을 설계하고 평가한다. nuclei-based와 membranous-based immunostains과 가장 관련된 두 가지에 대해서 제안한 방법이 효과적임을 실험을 통해 보여준다.
2. Related Work
Nuclei segmentation, most of them are fully supervised!
nuclei segmentation은 조직병리슬라이드에서 다양한 nuclei를 정확하게 segmentation할 수 있는 방법들에 집중한 challenge들을 통해 많은 주목을 끌고 있다.
하지만, 그것들은 fully supervised 방법에 집중하고 대부분 H&E 염색 도메인이다.
HER2-stained segmentation (2018) 이나 StarDist (2018) 와 같이, H&E의 pixel-based annotation에 의존한 방법이 어떤 IHC 염색에 일반화되기도 하는데, 하지만 이러한 방법들은 새로운 염색에 specific한 color augmentation 전략을 필수적으로 사용한다.
실제론, 주석의 표현력과 주석의 양 사이에는 trade-off가 존재함..
많은 양의 annotated tiles는 훈련데티어의 더 높은 variability 덕분에 segmenation systems의 일반화성능을 더 높일 수 있다.
Unsupervised nuclei segmentation
거꾸로, 다양한 thresholding 기반 방법들이 unsupervised nuclei segmentation을 위해 조사되었는데, Otsu thresholding 아니면 contrained local thresholding에 기반한다.
Self-supervised nuclei segmentation ~ using attention (2020)에서는 attention module을 사용해서 입력 tile의 배율을 정확하게 분류하는 network를 훈련시키고 attention map이 nuclei 분할 맵을 변환 될 수 있는 H&E 염색에서 nuclei의 탐지 맵을 제공할 수도 있다는 걸 보였다.
28. Sahasrabudhe, M., Christodoulidis, S., Salgado, R., et al.: Self-supervised nuclei segmentation in histopathological images using attention. In: International Con- ference on Medical Image Computing and Computer-Assisted Intervention. pp. 393–402. Springer (2020)Cross-domain learning
어떤 도메인에서 다른 도메인으로 모델을 적응 adapation 시키는 패러다임을 뜻한다.
예로 들어, H&E 도메인에서 IHC 도메인으로..! 이 맥락에서 Adversarial learning with data selection for cross- domain histopathological breast cancer segmentation (2022) 에서는 tissue segmenation에서 H&E, IHC 도메인의 high-level feature의 분포를 matching하므로써 H&E-IHC cross-domain learning을 다뤘다.
다른 최근 방법들은 다양한 접근방법으로 segmenation network를 훈련시키기 위해 GAN을 활용했다.
GAN은 style transfer를 통해 image를 생성할 수 있고 어느 한 도메인의 annotation을 다른 도메인에 사용해서 U-Net이나 Mask-RCNN과 같은 supervised network를 훈련시키는 데에도 사용할 수 있다. [13, 14]
13. Lin, Z., Li, J., Yao, Q., et al.: Adversarial learning with data selection for cross- domain histopathological breast cancer segmentation. Multimedia Tools and Ap- plications pp. 1–20 (2022)
14. Liu, D., Zhang, D., Song, Y., et al.: Unsupervised instance segmentation in mi- croscopy images via panoptic domain adaptation and task re-weighting. In: Pro- ceedings of the IEEE/CVF conference on computer vision and pattern recognition. pp.더 나아가, AD-GAN (2021)은 style transfer를 위한 image-to-image translation을 하기 위한 auto-encoder과 같은 방법을 제안하는데, 이 방법은 segmenation과 transfer를 동시에 학습한다.
31. Yao, K., Huang, K., Sun, J., Jude, C.: AD-GAN: End-to-end unsupervised nuclei segmentation with aligned disentangling training. arXiv preprint arXiv:2107.11022 (2021)This Study
이러한 방법들과 달리, 여기 방법은 염색에 독립적인 조직학적 조직 특징을 encoding하고 identifying하여 segmenation level에서 활용가능한 정보를 사용한다. 저자들이 알기론 이런 scheme의 연구가 explored되고 fully supervised 성능에 가까운 걸 보여준 것은 처음이다.
3. Methodology
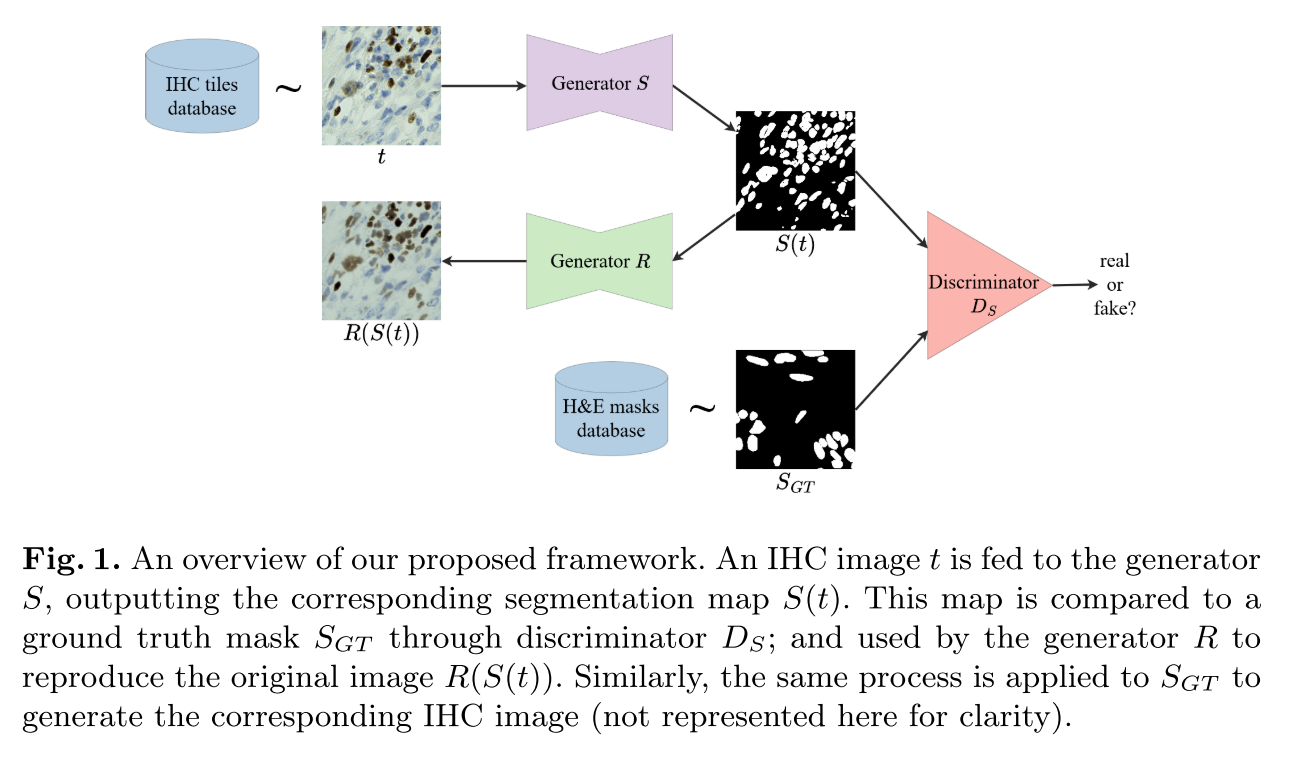
IHC image - nuclei mask 생성/복원과 생성된 predicted mask와 실제 H&E mask를 구분하는 task간의 adversarial한 관계 (Generator VS Discriminator) 로 학습하는 구조..
IHC 데이터로부터 predicted mask 를 만드는 Generator 과 H&E 마스크 데이터로부터 나온 와 를 real or fake로 구분하는 discriminator를 둬서 adversarial하게 학습하는 게 GAN의 기본 골자라면 거기에 를 다시 IHC 이미지를 복원하는 Generator 를 둬서 좀 더 IHC 염색에 대한 분포를 스스로 학습하도록 좀 더 보강한 느낌
는 noise로부터 시작할 것으로 보임
loss of dicriminator
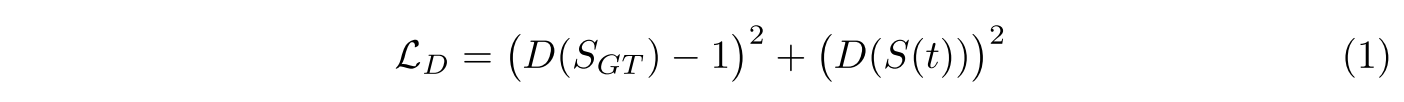
loss of generator
MSE loss 사용
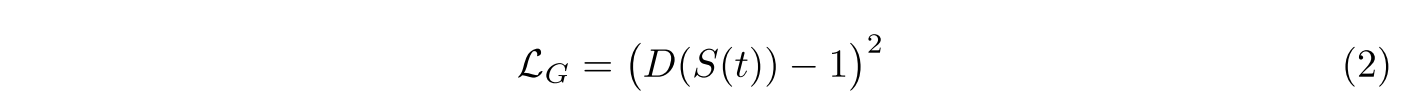
adversarial training with two losses above
위의 두 가진 loss를 가지고 실제 nuclei segmenatino map (shape prior) 과 닮은 nuclei object를 포함하는 segmetnation map을 생성하는 generator S를 훈련시켜야한다.
H&E mask 데이터베이스와 IHC 데이터베이스 및 생성 mask의 분포가 맞을때, 두 개의 loss가 최적화되어진다.
loss of generator
근데, 두 개 가지고 훈련 시키면 generator 가 false negative case를 만드는 경향을 보임. 그래서 예측된 로부터 IHC tile 를 복원하는 reconstructor 을 그 경향을 우회하기 위해 포함된다.
가 놓친 nuclei는 의 복원에서 error를 발생시키므로, 이는 곧 에게 false negative 숫자를 최소화하도록 한다.
sparsirty를 위해 norm을 사용함
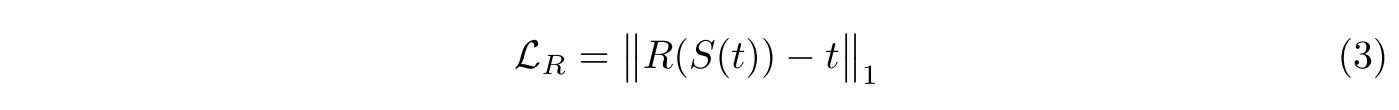
discrimitor
CycleGAN에 따라 R을 훈련시키기 위해 IHC 이미지와 복원된 IHC 이미지에 대한 discriminator를 더함. 위 discriminator loss를 사용한 것으로 보임
consistency loss
color augmentation한 IHC tile
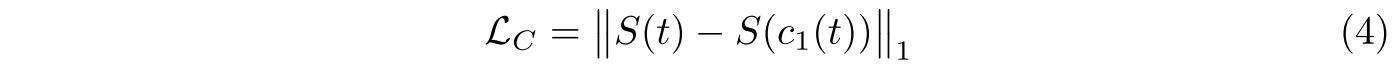
post processing segmenation maps
의 예측 logit에 sharpening fator r=60을 곱해서 를 sharpen한다.
total loss
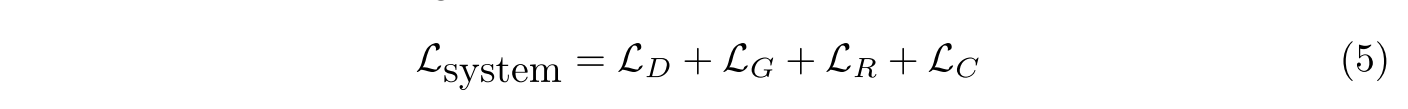
4. Experimental Configuration
Databases
DEEPLIFF dataset
- 1667 Ki67-stained fileds,
- 512 x 512 at 40x
- excluding the immunofluorescence data
- only for testing
BCDataset
- 1338 Ki-67 stained 640 pixel-width 40x
- 각 핵이 그 중심점의 single point로 annotation됨
- only for testing
WARWICK HER2 dataset
- 84 HER2-stained WSIs (50 training and 34 testing slides)..
patch extraction
- 512 x 512 and 256 x 256 after contour detection and filtering based on texture and lightness criteria
- K-Means clustering on the ResNet18 features of each patch, selected for each one the closest to centroids.
- isolation forest algorithm to remove the few artifacts after pre-processing steps
supervised mehthods
UNet
- fully supervised model
NuClick
- 핵의 중심점으로부터 nuclei segmentation을 하는 weakly supervised model
- 그걸 하기 위해 pathologist가 HER2 test 이미지에서 모든 핵 중심점으로 직접 annotation하고 DeepLIFF, BCDataset test set들의 각 핵 ground-truth mask의 중심을 계산하는 것으로 center를 얻었음
unsupervised methods
StarDist
- H&E로부터 훈련된 supervised 방법인데, extra annotation에 의존하지 않기 때문에, unsupervised 방법으로 두고 비교함
- QuPath의 plugin을 사용
Thresholding
- Gaussian filtered luminance image에서 Otsu thresolding
The proposed approach
- Unet style architectures for and
- PatchGAN-based discriminators for and
- 각 iteration에서 IHC tile 에 대해 S가 를 내뱉으면 20x, 40x의 H&E tiles의 nuclei instance mask를 가진 Pannuke dataset로부터 random하게 sample되는 와 함께 로 forward됨
- 비슷하게, 이 mask로부터 IHC image를 만들고 로 비교함
- HER2는 세포막 특성이 있기 때문에 deconvolved haematolxyin image만을 복원하도록 한다. nucleus를 segmentation하기 위해
Implementation Details
generators (S, R)
- 64 filters in the last conv layer and a dropout of 0.5
- Adam ( lr = 0.0002, =0.5, =0.999)
discriminators
- 64 filters and 3 layers in total
data aug
- rotation and flipping for nuclei invariance
- random resizing to 20x from 40x
UNet

- DeepLIFF dataset에 대한 최고 F1-score 기준
final model
250 epoch 이후 를 최소화하는 모델로 select
post-processing
mask에 median blur (ksize =5) 적용해서 noise를 줄여서 예측에 사용했고 HER2예 대해선 erosion (radius = 5)을 사용해 남은 artifact를 더 제거함
nuclei instance segmentation을 얻기 위해 마지막으로 opening, closing, watershed transform with labeling을 적용
공정한 비교를 위해 위와 같은 후처리가 Unet과 Thresholding output에 적용
5. Results
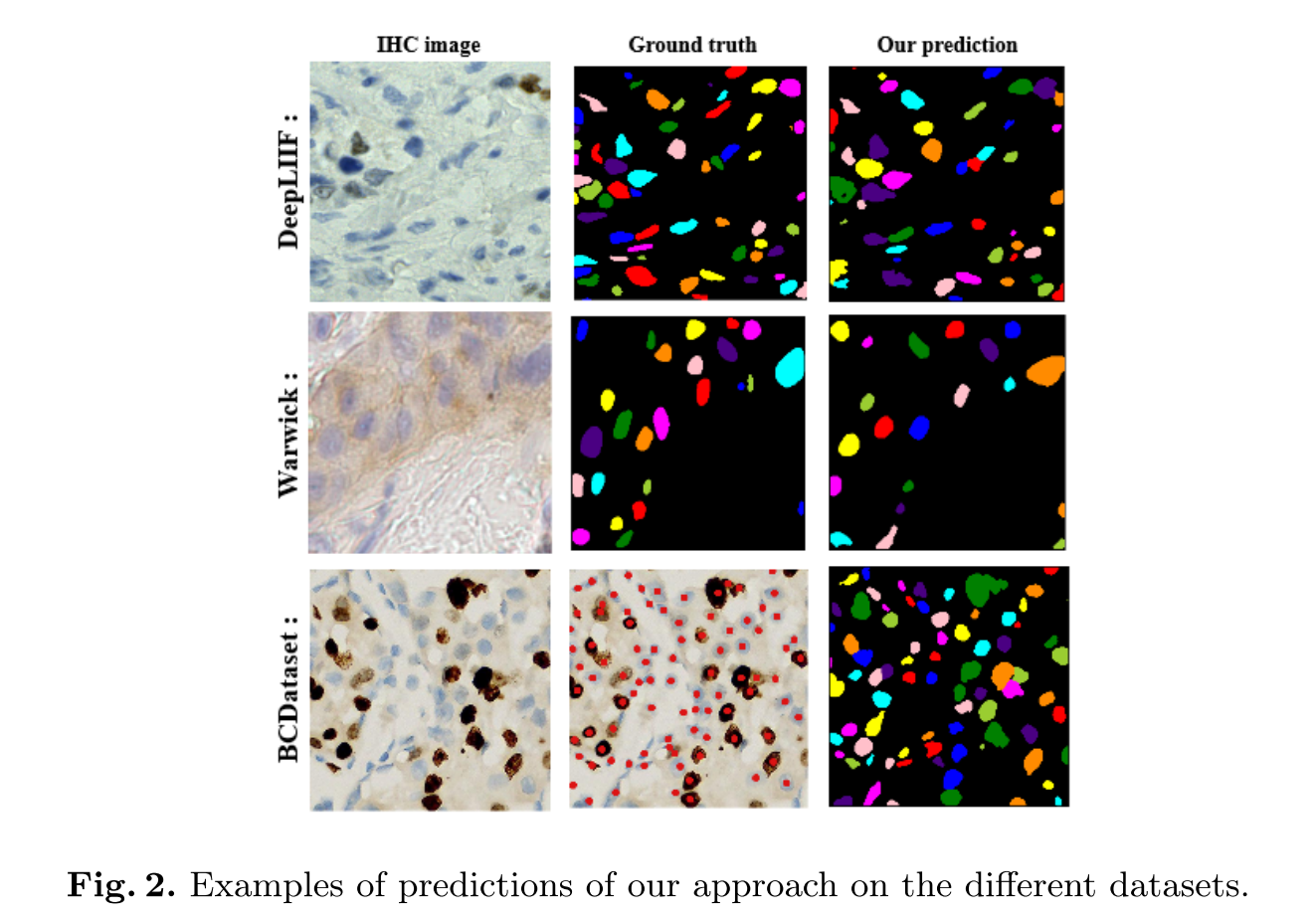
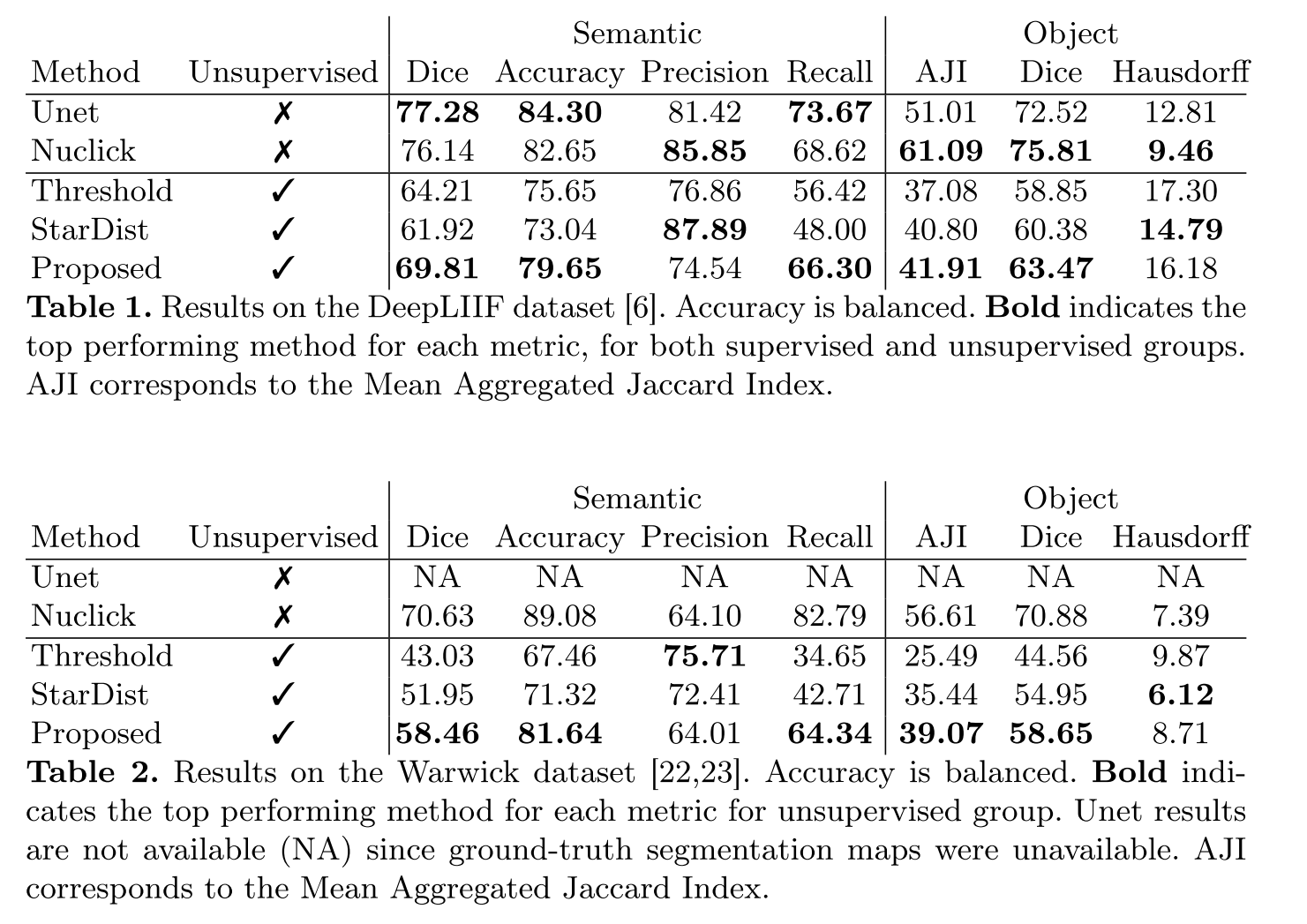
Table 1
unsupervised 중에 Dice와 balanced accuacy 최고 달성
StarDist가 best precision을 찍었지만 false negative는 환자 치료과정을 악화시킬 수 있기 때문에 precision에 recall을 교환하는 것이 임상에서 좀 더 낫다고 해석하고 있음
!이건 상황에 따라 다르므로 해석의 차이가 있음
어떤 factor를 볼 때, 위양성에 더 민감한 거라면 애매한 양성을 위음성으로 미루더라도 확실한 양성을 잘 잡는 모델을 설계해야하고 recall보다 precision이 높은 게 더 나을 수 있음
그렇더라도, 위 결과에선 StarDist의 recall (48.00) 이 너무 낮아 제 기능을 하는 모델로 임상에 쓰기엔 역부족하다는 게 보이는 것
UNet (fully supervised), Nuclick (weakly supervised)와 봤을때, balanced accuracy에서 5% 이하의 개선이 이루어진 걸 확인하였음
supervised 방법들이 낡아서 조금 민망한 비교이긴 하나, unsupervised라고 치더라도 괜찮은 성능이 나온다는 걸 보여줌
Table 2
unsupervised 중에 dice, balanced accuracy에서 압승, recall 승
proposed approach
더 높은 recall과 object metric으로 임상에 더 적합한 것을 입증했다.
훈련과 후처리에 조금의 수정을 함으로써 서로 다른 두 염색 조건에 성공적으로 자신들의 방법을 적용하여 사전훈련된 SOTA 알고리즘을 직접 적용하는 것보다 H&E information을 더 잘 활용할 수 있다.
6. Conclusion
spatial organizatio prios를 결합한 nuclei segmentation unsupervised framework를 제안했고 3개의 heterogeneous한 데이터셋에 대해서 실험을 진행해 unsupervised approach들을 앞서고 supervised approach와의 gap을 줄일 수 있었다.
지금은 단순히 nuclei segmentation을 하는데, nuclei type을 구분해내는 것도 임상에서 중요한 일이기 때문에 확장 연구가 굉장히 흥미로운 방향이 될 것이다.
7. Comments
방법
결국엔 IHC로부터 생성된 mask와 H&E mask를 비교한 discriminator와 generator간의 adversarial한 훈련 scheme인데.. 그게 spatial organization priors를 더 썼다고 말할 수 있는 건가..?
R을 추가해서 S가 nuclei를 놓치지 않도록 (false negative가 발생하지 않도록, recall이 떨어지지 않도록) 강제한 느낌이지.. spatial organization priors라는 말을 쓰기엔 내부적인 behavior를 밝힌 부분이 없다.
그런 말을 쓰려면 H&E image와 IHC image에 대한 feature를 가지고 염색에서 오는 차이를 invariant하게 만들어 버리고 nuclei/tissue의 모양, 구성에 대한 feature map을 matching하는 쪽이 좀 더 일리가 있어보인다. (domain adaptation 관점으로.. H&E에서 이미 훈련된 모델로 H&E랑 IHC 이미지 모두 입력으로 받아서?)
전체적인 훈련 scheme은 따른다면, 내부 구조의 변경을 통해 개선여지가 있을 것으로 보인다
최신 generative model (diffusion), 최신 classifier (ViT 계열) 을 쓰던가
내부적인 layer를 경량화시키고 최신화하던가..?
수치평가
unsupervised 비교군이 얕고 구식이다..
그리고 StarDist (2018) 의 IHC에서 그냥 사용했을 때 recall이 형편없다고 볼 수도 있는데, 입력을 변형해줌에 따라서 성능이 충분히 달라질 수도 있어서 완전히 신뢰하기 힘들다.
테스트 데이터셋으로 사용한 DeepLIFF (2022) 의 모델만 해도 Ki-67, nuclei biomarker에서 더 잘 될 것으로 보이고 PD-L1에서도 성능평가를 수행했었는데 HER2에서도 잘 되지 않을까 싶다
supervised approach에서 training set을 얼만큼식 사용했을때에는 제안하는 unsupervised approach가 오히려 낫더라라는 것도 있었으면 좋았겠다.. 어느 선까진 unsupervised approach가 나을 수 있다는 통찰을 얻을 수 있으니까
정성평가
너무 적다.. 잘 나온 거라도 좀 더 골라보지..
그럼에도 불구하고 false case에 대한 report를 안 하는 건 너무 아쉽다.
인접한 nuclei를 구분하는 게 여전히 어렵다던가 세포가 작으면 더 놓치더라 라던가
특히 어떤 case에 대해서 못한다던가 병리학적인 견해도 전혀 없어서 아쉽
technical하게는 훈련에 따라 생성/복원이 어떤 관계로 발전하는 지 보여주면 좋지 않을까
효용성
H&E mask를 활용하여 IHC 염색 이미지에서 별도의 annotation 없이 nuclei segmentation이나 다른 task를 할 수 있는 framework로 참고해볼 만하다.
하지만, 임상에 사용하기 위해선 정량적 평가를 해야하고 그것을 위해선 annotation 확보가 되어야하는데, DeepLIFF로 좀 더 나은 예측결과를 통해 annotation을 확보하고 annotation이 확보되면 그 또한 training에 쓰는 게 좀 더 확실한 성능을 확보할 수 있다
annotation이 확보가 되는데, unsupervised approach를 유지할 이유는 없다..
semi-supervised approach에 대한 비교도 필요하지 않을까..?
