야생의 K-Means Clustering

최근에 unsupervised learning에 대해 배웠습니다. Unsupervised learning의 핵심(목적)은 주어진 데이터 내에서 패턴을 찾아내는 것이고, 상황에 따라 이를 할 수 있는 여러 가지 방법들과 그 원리를 배웠습니다 (재밌고 신기하고 이런 방법을 처음 생각해낸 사람들은 뭘 먹고 그렇게 똑똑해졌을까 싶고 부럽더군요).
전 야생에서 unsupervised learning이 사용 된 예시를 찾아보고 싶었습니다. 구글에 "unsupervised machine learning clinical"을 쳐보니 이런 바로 연구를 찾을 수 있었습니다:
Histopathology(조직병리)란 신체 장기의 일부를 떼내서 현미경으로 보는 것을 의미하고, chronic kidney disease (CKD, 만성신장병)는 어떤 원인에서든 신장 기능이 떨어진 상태를 말합니다 (보통 당뇨가 원인입니다).
K-means clustering을 사용해서 신장 조직병리를 분석하고, 이를 바탕으로 만성신장병의 중증도를 예측한 논문인데요, 사람이 한 labelling없이도 조직병리 소견을 분석하여 임상적으로 의미있는 결론(중증도 예측)까지 이어질 수 있다는 것을 보여줬다는 의의가 있습니다. 처음 읽을 땐 한 4% 정도 이해했고 지금도 '그래 이건 그냥 이런게 있다고만 알자'하고 넘어간 부분이 엄청 많지만 일단 K-means clustering을 조직병리 분석에 어떻게 사용한건지 설명하고자 하는 것이 이 글의 목적입니다.
Bag of Visual Words란
우선 bag of visual words라는 방법에 대한 설명이 필요합니다. Natural language processing 분야에 "bag of words"라는 분석방법이 있는데요, 어떤 문서가 있으면 문서 내 단어들의 순서나 맥락따윈 신경쓰지 않고 그냥 단어 갯수만 세서 히스토그램을 만들고 히스토그램들끼리 비교하여 문서들이 서로 얼마나 비슷한지, 혹은 얼마나 다른지를 보는 분석입니다. Bag of visual words는 이 방법을 image 데이터에 적용한 것이고, 단어(word) 대신 image의 feature(혹은 여러 개의 feature로 이루어진 feature vector)를 사용하여 각 image의 feature histogram을 만들어 비교합니다. 이 때 feature란 그 이미지를 이루는 pattern들입니다. 서로 다른 질감들을 비교하기 위해 처음 사용했다고 하는데, 아래 사진을 보시면 어떤 느낌인지 이해가 갑니다:
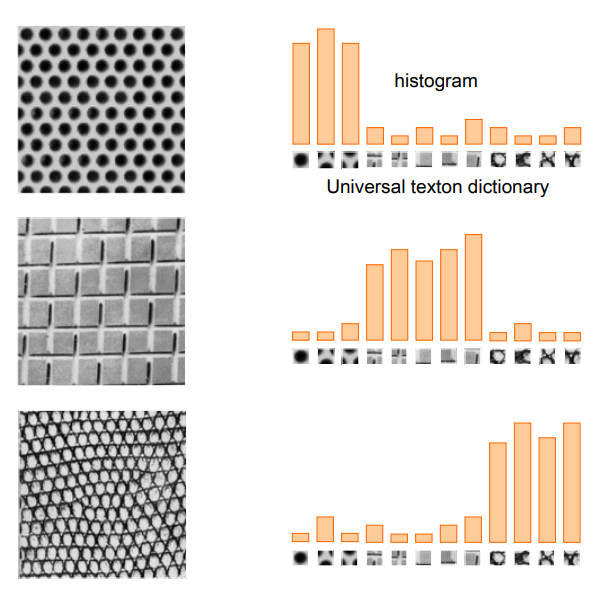
(Source: Kris Kitani, Carnegie Mellon University. 8.2 Bag of Visual Words (cmu.edu))
그럼 이미지 분석을 위한 feature선정은 어떻게 하는 걸까요? 여러 가지 방법(eg SIFT)이 있지만, 우리의 만성신장병 논문에선 신장 조직병리 사진들을 segmentation하는데 이용한 deep neural network의 한 layer를 feature set으로 사용했다고 했습니다 (= ResNet feature extraction).
이렇게 고른 feature들 중에서, 가장 대표적인 놈들을 몇 개 선정해서 걔네를 "code word"(= visual word)라 부르고, code word들의 전체 집합을 "codebook" 혹은 "visual dictionary"라 부릅니다:

(Source: Kris Kitani, Carnegie Mellon University. 8.2 Bag of Visual Words (cmu.edu))
수많은 feature들 중에서 code word들을 정할 때 바로 K-means clustering이 사용됩니다. Feature(혹은 feature vector)들 중 비슷한 애들끼리 군집화해서 평균(centroid)에 해당하는 녀석을 code word로 사용하는 것이죠.
그러면 새로운 image가 주어졌을 때, 그 image를 code word들의 histogram으로 나타낼 수 있습니다:
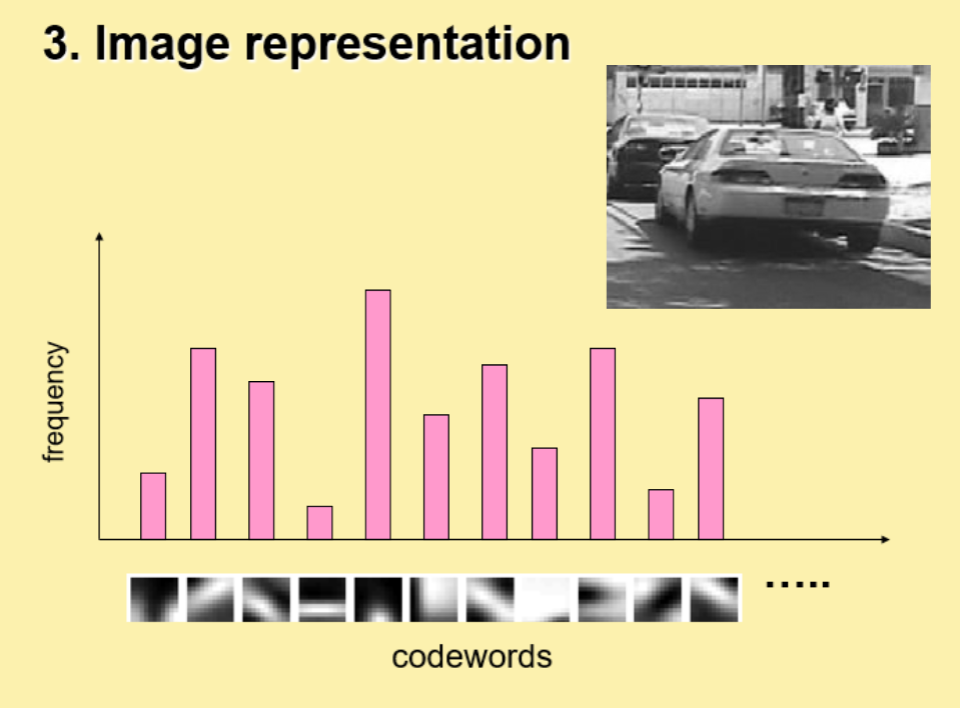
(Source: Li Fei-Fei, Rob Fergus, Antonio Torralba. part_1.ppt (live.com), Recognizing and Learning Object Categories)
신장병 논문에서의 K-means clustering 사용
우리의 신장병 논문에선 신장 조직병리 이미지들을 모두 256x256 pixels크기의 patch로 나누었고, 이 patch들을 9개의 클러스터로 나누는 K-means clustering을 하여 9개의 code word (=visual word)를 선정했습니다. 이 때 클러스터의 갯수는 silhouette이라는 알고리즘을 이용해 9로 정했다고 합니다 (강의에서 K-means clustering을 사용할 때 유의해야할 점 - 즉, 약점 - 중 하나가 클러스터의 갯수를 선정해야하는 것이라 배웠는데, 이 논문에서도 미래의 연구에선 k를 달리하여 분석을 해봐야 할 것임을 얘기하고 있습니다).논문의 Figure 6(A)가 K-means clustering을 통해 도출 된 9개의 visual word(=code word)들을 보여주고 있습니다. 6(B)는 현미경으로 본 신장조직 샘플이고, 6(C)는 6(B)의 각 patch(256x256 pixels)가 어떤 visual word의 클러스터 속하는지 보여주고 있습니다.
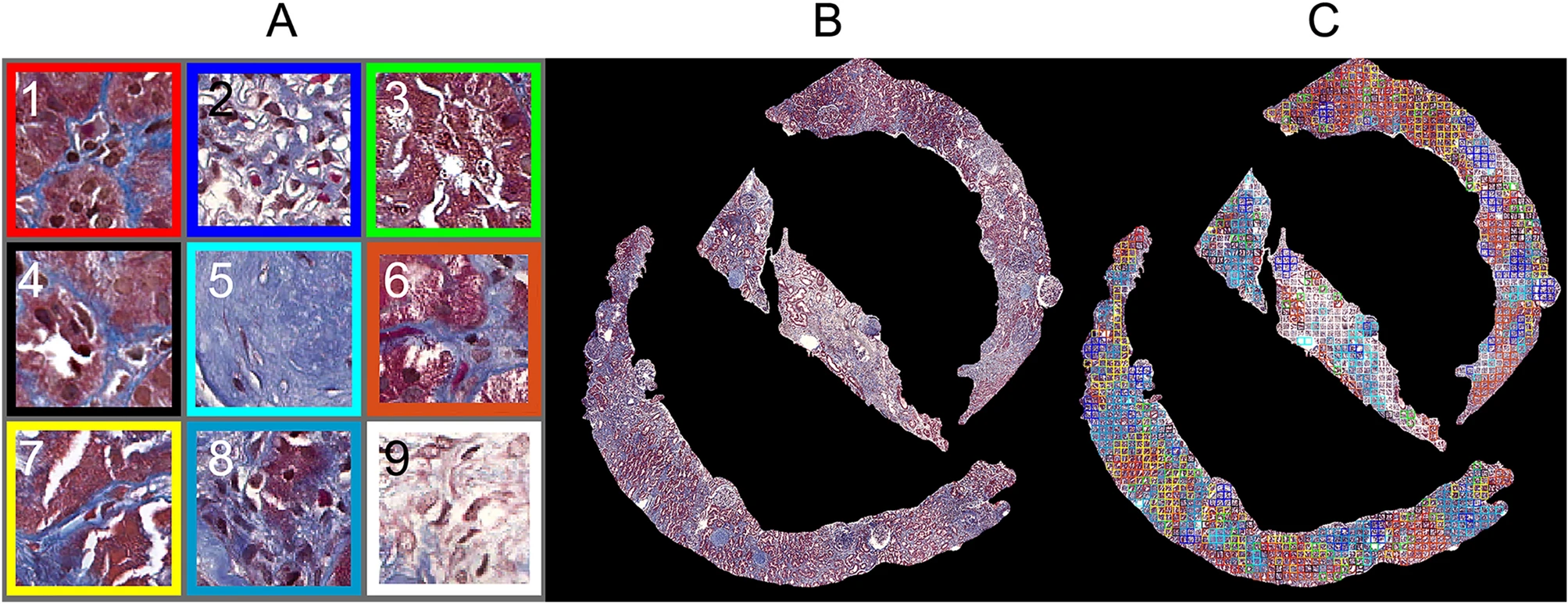
Figure 6 (A) A visual dictionary that consists of 9 representative visual words, (B) a represnetative cortex sample, and (C) its cluster map with colored patches. Each colored patch corresponds to its assigned visual word [1]
Figure 7은 Figure6(C)를 zoom in해서 보여주네요.

Figure 7. An example of cortex trichrome stained images with color-coded patches and zoomed images [1]
클러스터링의 결과물인 Figure 6(A)의 visual word들을 조금 더 자세히 살펴보도록 할텐데, 그 전에 신장 조직학에 대해 잠깐 설명하겠습니다. 신장은 크게 3 가지 조직(조직이란 세포들이 모여 형성 된, 한 가지 기능을 하는 세포들의 집합을 말합니다. 조직이 모이면 장기(eg, 신장)가 되고, 장기들이 모이면 한 개체(eg, 사람)이 됩니다)으로 이루어져 있습니다: 사구체 (glomeruli), 세관 (tubules), 실질 (interstitium). 사구체는 혈액으로부터 노폐물(소변)을 걸러내는 역할을 하고, 세관은 걸러진 소변을 방광으로 운반하며, 실질은 이 둘을 뺀 나머지라 생각하면 됩니다. 그러나 세관과 실질은 보통 붙어있기 때문에 tubulointerstitium이라는 이름으로 하나의 조직으로 취급하는 경우가 많습니다. 논문의 Figure4가 친절하게 현미경으로 본 사구체, 세관, 실질의 모습을 보여주고 있네요 (여기선 혈관(arterioles)도 따로 분류해서 보여주고 있군요 중요하진 않습니다)
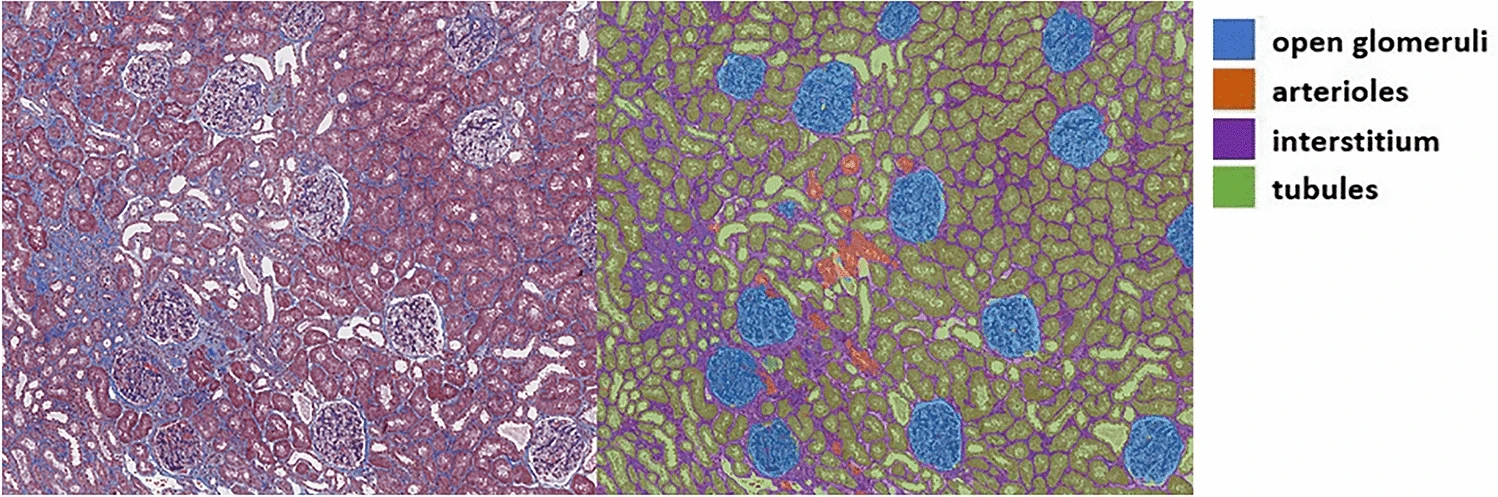
Figure 4. An example of a trichrome-stained image (left) and an automatically segmented image from our trained deep learning model (right) [1]
이제 Figure 6(A)의 visual word들을 보면, 잘 모르는 제가 봐도 2번은 정상 사구체 (약간의 염증이 있어보이긴 하지만 대충 정상), 5번은 병든 사구체인 것은 알아볼 수 있었습니다. 나머지 visual word들은 어떤 소견인지 저는 알아보지 못 했지만 다행히도 논문의 Table5에 친절히 설명되어있습니다:
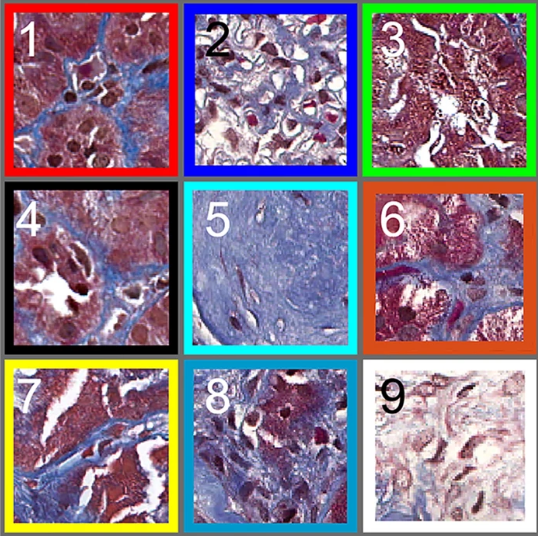
Figure 6(A) A visual dictionary that consists of 9 representative visual words [1]
1. 정상 세관/실질
2. 정상 사구체 (염증 약간)
3. 정상 세관/실질
4. 정상 세관/실질 + 실질 확장 약간
5. 사구체경화증 (병들고 기능 잘 못 하는 사구체란 뜻입니다)
6. 정상 세관/실질
7. 정상 세관/실질
8. 세관 위축 (세관이 병든 소견입니다)
9. 실질 확장 (이 또한 신장질환에서 관찰되는 소견인데, 5번 사구체경화증이나 8번 세관 위축소견만큼 중요한 소견은 아닙니다)
사람이 라벨링한 데이터를 보지 않고도 clustering만으로 신장조직을 이루는 사구체와 세관/실질을 구분했을 뿐만 아니라, 병든 사구체나 병든 세관의 모습도 구분해내었네요. 물론 이 상태에서는 알고리즘이 어떤 visual word가 신장병의 소견이고 어떤 것이 정상인진 알지 못하지만, 그 다음 단계에서 저자들이 가르쳐줍니다: 각 환자의 신장 조직병리 이미지에 대해 이 visual word들로 이루어진 히스토그램을 생성한 뒤 신장병이 얼마나 심한지를 예측하는 supervised learning을 진행했습니다 (환자들을 경도 만성신장병과 중등도/중증 만성신장병으로 분류하여 라벨링하고 random forest classifier를 트레이닝하는 방법 사용). 신장병 중증도 예측은 정확도 AUC 0.91 정도로 꽤 정확하게 예측할 수 있었습니다 (물론 경도 vs. 중등도/중증 두 그룹으로만 분류하면 됐기 때문에 task자체가 그렇게 어려운 것은 아닌 것 같지만요). 더 재밌는 것은 신장병의 중증도를 평가하는데 있어 random forest가 사용한 feature들의 feature importance를 계산하였을 때, visual word 2번 (정상 사구체 소견), 8번 (세관 위축 소견), 5번 (사구체경화증 소견)이 1, 2, 3순위로 가장 중요한 feature로 나타났습니다. 이것은 인간 병리학자가 현미경 소견으로부터 만성신장병의 중증도를 파악할 때 가장 중요하게 보는 소견들과 매우 비슷합니다 (사구체 중 몇 퍼센트가 정상모습인지, 사구체경화증이 일어난 사구체는 몇 얼마나 많은지, 세관 위축은 얼마나 진행됐는지 등을 보고 만성신장병의 중증도를 파악합니다).
여기서부턴 제 생각
이 연구에서 이용한 데이터셋은 만성신장병을 가진 사람들의 신장 조직병리소견들로 이루어져 있기 때문에, '정상 사구체' visual word도 염증을 일부 포함하고 있었지만, 만약 건강한 사람들의 신장 조직병리도 포함하여 learning을 진행한다면 완전 정상인 사구체들이 하나의 cluster를 이루고 별개의 visual word로 작용하여 신장병 중증도에 대한 예측 정확도를 더 높일 수 있을 것으로 생각됩니다. 또한, 사구체경화증(visual word #2)을 일으키는 원인들과 세관 위축(visual word #8)을 일으키는 원인들은 조금 다릅니다 (겹치기도 하지만요). 이 연구에서는 '만성신장병'(원인에 상관없이 신장기능이 저하 된 상태) 환자들의 조직병리를 대상으로 했지만, 각 환자가 어떤 원인에 의해 만성신장병을 가지게 되었는지에 대한 데이터도 라벨로 포함할 수 있다면, 중증도 예측뿐만 아니라 신장기능 저하의 원인 예측도 가능하지 않을까 하는 생각이 들었습니다.마무리
K-means clustering방법으로 supervised learning을 위해 필요한 라벨링을 대체해버린 좋은 예시인 것 같습니다. 그리고 사람이 제시한 라벨링을 통해 배우는 것보다 더 유기적(?)이면서도 체계적으로 각 이미지 내에 어떤 패턴들이 존재하는지 알고리즘을 통해 발견해낸거라서 더 first principles에 가까운 방법이라 느껴집니다. 또한 새로운 발견을 이끌 수 있는 잠재력도 있을 것 같습니다, 왜냐하면 unsupervised learning이 발견한 visual word 중 인간 병리학자들은 모르던 패턴이 만성신장병 중증도 예측에 중요한 것으로 나타났다면, 새로운 조직병리 패턴을 하나 발견하게 되는 것이니까요. 이런 면들 때문에 unsupervised learning은 supervised learning보다 왠지 좀 더 멋있는 느낌입니다.끝!
ML 방법론에 대해서도, 병리학에 대해서도 전문가가 아니라서 틀린 부분이 있을(많을) 수 있습니다. 지적해주세요! 설명이 충분치 않거나 궁금한 점도 말씀해주시면 추가 설명해보도록 하겠습니다.
:D
[1] Lee, J., Warner, E., Shaikhouni, S. et al. Unsupervised machine learning for identifying important visual features through bag-of-words using histopathology data from chronic kidney disease. Sci Rep 12, 4832 (2022). https://doi.org/10.1038/s41598-022-08974-8


와 야생에서 unsupervised learning이 사용 된 예시라니!! 이것이야말로 딥다이브네요!!!깊게 파고들어서 공부하는 모습 멋있습니다