복습문제
# 다항 회귀의 차수를 높일수록 과적합의 문제가 크게 발생한다
o
# 일반적으로 편향과 분산은 한쪽이 높아져도 다른 한쪽은 영향을 받지 않는다
x
# _ 규제를 적용하면 영향력이 크지 않은 회귀 계수 값을 0으로 변환한다
L1
# 분휴와 회귀의 차이는
# 분류의 예측값은 _이고
# 회귀의 예측값은 _라는 것입니다
카테고리 형태 / 연속된 숫자형
# 회귀에서 학습이 얼마나 잘 되었는가를 평가하는 지표는 무엇인가요?
[출제자 답변]
MSE, MAE, RMSE
# MAE(Mean Absolute Error)을 설명해주세요
- 보통 절대값을 취하여 더하는 방식
- 실제값과 예측값의 차이를 절대값으로 변환해 평균 값을 구한다
from sklearn.preprocessing import PolynomialFeatures
import numpy as np
import seaborn as sns
from sklearn.linear_model import LinearRegression
# y = 1 + 4x + x**2
X = np.arange(-10,10)
y = 1 + 4*X + X**2 + np.random.randn(20)*0.1
sns.scatterplot(x = X,y = y);
# 위 코드의 [ lr.coef_, lr.intercept_ ] 값을 구하세요
poly = PolynomialFeatures(degree=2)
poly_1 = poly.fit_transform(X.reshape(-1,1))
lr = LinearRegression()
lr.fit(X.reshape(-1, 1), y)
print(lr.coef_, lr.intercept_)
# [3.0050194] 34.003337566540615
# 회귀 예측의 핵심은 주어진 피처와 결정값 데이터 기반에서
# 학습을 통해 최적의 ___를(을) 찾아내는 것이다
회귀계수
# 규제를 적용한 모델 3가지
릿지, 라쏘, 엘라스틱넷
# MSE에 루트를 씌운 것 (RMSE)
# 실제 값과 예측값의 차이를 절대값으로 변환해 평균한것 (MAE)
# 실제값의 분산대비 예측값의 분산 비율을 지표로 하며,
# 1에 가까울 수록 예측 정확도가 높다 (R^2)
# 실제 값과 예측값의 차이를 제곱해 평균한것(MSE)
# 관련 함수들 적기
# ex) Cost Function : 비용함수
Loss Function : 손실함수
Objective Function : 목적함수
Error Function : 오차함수
# LinearRegression클래스는 예측값과 실제값의 RSS(잔차제곱의 합)을 최소화하여
# Ordinary Least Squares(일명, OLS)추정 방식으로 구현한 클래스이다.
# OLS 기반의 회귀 계수 계산은 피처의 독립성에 많은 영향을 받는데,
# 피처간의 (____)가 매우 높은 경우 오류에 매우 민감해진다.
# 이러한 현상을 다중 공선성(multi-collinearity)이라 하며,
# 이와같은 경우 (_______)한 피처만 남기고 제거하거나 (___)를 적용한다
상관관계, 중요한, ...
[출제자 답변]
상관관계, 중요한, PCA
# 회귀 모델에서 'MAE', 'MSE', 'RMSE'의 평가 지표는 수치가__ 잘 예측했다고 표현된다
낮을수록
# _는 회귀라는 이름이 붙어있지만, 사실은 분류에 사용되는 선형 모델이다.
로지스틱 회귀
# 릿지 회귀는 알파 값을 계속 증가시키면 히ㅗ귀계수 값이 작아진다
o
# 편향-분산 트레이드 오프란?
한쪽이 높으면 한쪽이 낮아지는 경향이 있다 → 편향이 높으면 분산은 낮아지고(과소적합) / 분산이 높으면 편향이 낮아진다(과적합)
따라서 둘의 합이 가장 최소가 되는 적당한 지점을 찾아야한다
# 선형회귀에 L2 규제를 추가해
# 상대적으로 큰 회귀 계수 값의 예측 영향도가 감소된 모델은 무엇인가?
ridge(릿지)
# 비용함수에 알파 값으로 패널피를 부여해 회귀 계수 값의 크기를 감소시켜 과적합을 개선하는 방식을 ()라고 한다.
규제위키북스의 '파이썬 머신러닝 완벅 가이드' 개정 2판으로 공부하고 있습니다. 실습 과정과 이해에 따라 내용의 누락 및 코드의 변형이 있을 수 있습니다.
회귀
라쏘 회귀
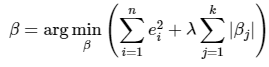
W의 절대값에 페널티를 부여하는 L1 규제를 선형 회귀에 적용한 회귀이다. L2규제가 회귀 계수의 크기를 감소시키는 데 반해, L1규제는 불필요한 회귀 계수를 급격하게 감소시켜 0으로 만들고 제거한다. 이러한 측면에서 L1 규제는 적절한 피처만 회귀에 포함시키는 피처 선택의 특성을 가지고 있다.
사이킷 런은 Lasso 클래스를 통해 구현할 수 있으며 주요 생성 파라미터는 alpha이다. 이는 라쏘 회귀의 alpha L1 규제 계수에 해당한다.
from sklearn.linear_model import Lasso, ElasticNet
from sklearn.model_selection import cross_val_score
# 인자로 회귀 모델의 이름, alpha값들의 리스트, 피처 데이터 세트와 타깃 데이터 세트를 받아서
# alpha값에 따른 회귀 모델의 폴드 평균 RMSE를 출력하고 회귀 계수 값들을 DataFrame으로 반환하는 사용자 함수
def get_linear_reg_eval(model_name, params=None, X_data_n=None, y_target_n=None, verbose=True, return_coeff=True) :
coeff_df = pd.DataFrame()
# 모델이름 출력
if verbose :
print('####', model_name, '####')
# 모델에 따라 설정
for param in params :
if model_name == 'Ridge' : model = Ridge(alpha=param)
elif model_name == 'Lasso' : model = Lasso(alpha=param)
elif model_name == 'ElasticNet' : model = ElasticNet(alpha=param, l1_ratio=0.7)
# 교차 검증
# cross_val_score는 evaluation metric만 반환하므로 모델을 다시 학습하여 회귀 계수 추출
neg_mes_scores = cross_val_score(model, X_data_n, y_target_n, scoring='neg_mean_squared_error', cv=5)
avg_rmse = np.mean(np.sqrt(-1*neg_mes_scores))
print(f'alpah {param}일 때 5 fold set의 평균 RMSE : {avg_rmse:.3f}')
# 회귀 계수
model.fit(X_data_n, y_target_n)
if return_coeff :
# alpha에 따른 피처별 회귀 계수를 Series로 변환하고 이를 DataFrame의 칼럼으로 추가
coeff = pd.Series(data=model.coef_, index=X_data_n.columns)
colname='alpha'+str(param)
coeff_df[colname] = coeff
return coeff_df
X_data = boston_df.iloc[:,:-1]
y_target = boston_df.iloc[:,-1]
lasso_alphas = [0.07, 0.1, 0.5, 1, 3]
lasso_coef = get_linear_reg_eval("Lasso", params=lasso_alphas, X_data_n=X_data, y_target_n=y_target)#### Lasso ####
alpah 0.07일 때 5 fold set의 평균 RMSE : 5.612
alpah 0.1일 때 5 fold set의 평균 RMSE : 5.615
alpah 0.5일 때 5 fold set의 평균 RMSE : 5.669
alpah 1일 때 5 fold set의 평균 RMSE : 5.776
alpah 3일 때 5 fold set의 평균 RMSE : 6.189알파가 0.07일 때 RMSE의 평균이 약 5.612로 가장 작은(좋은)것을 확인 할 수있다.
- 릿지 출력값
알파값이 0일 때, 5 folds의 평균 RMSE : 5.829
알파값이 0.1일 때, 5 folds의 평균 RMSE : 5.788
알파값이 1일 때, 5 folds의 평균 RMSE : 5.53
알파값이 10일 때, 5 folds의 평균 RMSE : 5.518
알파값이 100일 때, 5 folds의 평균 RMSE : 5.330어제 봐줬던 릿지보다는 약간 높은 결과인 것 같다.
lasso_coef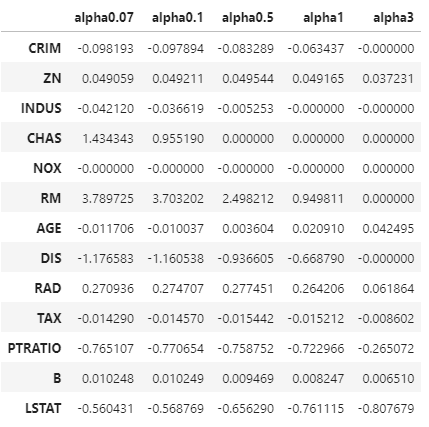
alpha(하이퍼 파라미터)의 크기가 증가함에따라 일부 피처의 회귀 계수는 아예 0으로 바뀌는 모습을 확인할 수 있었다. 회귀계수가 0인 피처는 회귀 식에서 제외되며 결과적으로 피처 선택의 효과를 얻을 수 있다.
Elastic Net
엘라스틱넷 회귀모형은 가중치(회귀계수) 절대값의 합과 제곱합을 동시에 제약조건(L1, L2 규제의 결합)을 가지는 모형이다.
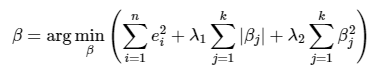
λ1, λ2 라는 두 개의 하이퍼 모수를 가진다.alpha값에 따라 회귀 계수의 값이 급격히 변동할 수 있는 것을 완화가기 위하여 L2규제를 라쏘회귀에 추가한 것인데, 단점은 두 규제가 결합되다보니 수행시간이 상대적으로 오래 걸린다는 점이다.
# l1_ratio는 0.7로 고정
elasitic_alphas = [0.07, 0.1, 0.5, 1, 3]
elasitic_coef = get_linear_reg_eval("ElasticNet", params=elasitic_alphas, X_data_n=X_data, y_target_n=y_target)#### ElasticNet ####
alpah 0.07일 때 5 fold set의 평균 RMSE : 5.542
alpah 0.1일 때 5 fold set의 평균 RMSE : 5.526
alpah 0.5일 때 5 fold set의 평균 RMSE : 5.467
alpah 1일 때 5 fold set의 평균 RMSE : 5.597
alpah 3일 때 5 fold set의 평균 RMSE : 6.068alpah가 0.5일때 RMSE가 가장 작다(좋다)
elasitic_coef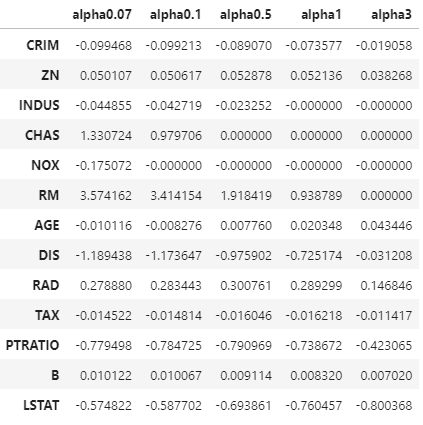
앞서 봤던 Lasso에 비해 alpha값이 커진다고 해서 0이 되는 값이 많지 않다.
선형 회귀 모델을 위한 데이터 변환
선형회귀 모델과 같은 선형 모델은 일반적으로 피처와 타깃값 간에 선형의 관계가 있다고 가정하고, 이러한 최적의 선형함수를 찾아내 결과값을 예측한다. 또한 피처값과 타깃값의 분포가 정규분포(=평균을 중심으로 종 모양으로 데이터 값이 분포된 형태)를 매우 선호한다.
사이킷런을 이용해 피처 데이터 세트에 적용하는 변환작업은 다음과 같은 방법이 있을 수 있다.
- StandardScaler 클래스를 이용해 평균이 0, 분산이 1인 표준 정규 분포를 가진 데이터 셋으로 변환하거나, MinMaxScaler 클래스를 이용해 최소값이 0이고 최대값이 1인 값으로 정규화를 수행
→ 예측 성능 향상을 크게 기대하기 어려운 경우가 많다 - 스케일링/정규화를 수행한 데이터 셋에 다시 다항 특성을 적용하여 변환하는 방법. 보통 1번 방법을 통해 예측 성능에 향상이 없을 경우 이와 같은 방법을 적용
→ 피처의 개수가 매우 많을 겨우 다항 변환으로 생성되는 피처의 개수가 기하급수로 늘어나 과적합의 이슈가 발생할 수 있다. - 원래 값에 log 함수를 적용하면 보다 정규 분포에 가까운 형태로 값이 분포된다. 이러한 변환을 로그변환이라고 부르는데, 매우 유용한 변환으로 실제 선형 회귀에서는 앞에서 소개한 방법보다는 로그 변환이 훨씬 많이 사용된다.
로지스틱 회귀
로지스틱 회귀는 선형 회귀 방식을 분류에 적용한 알고리즘이다. 일반적인 선형회귀와는 조금 다르다. 학습을 통해 선형 함수의 회귀 최적선을 찾는 것이 아니라, 시그모이드(Sigmoid)함수의 최적선을 찾고 이 시그모이드 함수의 반환값을 확률로 간주하며 그 확률에 따라 분류를 결정한다는 점이다.
단순 선형 회귀에서는 목표가 실수값 예측이다. 따라서 선형함수인 wx + b를 이용하여 예측할 수 있지만 로지스틱 회귀분석에서는 종속변수가 0또는 1이기 때문에 y = wx+b를 통해 예측하는 것은 의미가 없다. 그래서 Odds(오드, 오즈)값을 이용한다.
만약 독립변수가 하나라면 공식은 다음과 같다.
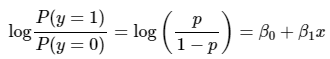
이 식을 정리하여 확률에 대해 나타내면 다음과 같다.
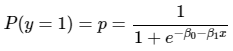
위 식에서 1/1+e(¯x)형태의 함수식을 시그모이드 함수라 한다.
구현하며 이해해보기
사이킷런에서는 LogisticRegression클래스를 이용하여 로지스틱 회귀를 해볼 수 있다. 앞에서 사용하던 위스콘신 유방암 데이터 셋을 기반으로 실습을 진행해보자
데이터 불러오기
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.datasets import load_breast_cancer
from sklearn.linear_model import LogisticRegression
# 데이터 불러오기
cancer = load_breast_cancer()
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split
# StaStandardScaler()로 평균이 0, 분산이 1이 나오도록 데이터 분포도 변환
scaler = StandardScaler()
data_scaled = scaler.fit_transform(cancer.data)
# train, test
X_train, X_test, y_train, y_test = train_test_split(data_scaled, cancer.target, test_size=0.3, random_state=0)선형회귀 계열인 로지스틱 회귀는 데이터의 정규 분포도에 따라 예측 성능이 영향을 받을 수 있으므로, 데이터에 먼저 정규 분포 형태의 표준 스케일링을 적용한 뒤 데이터 셋을 나누었다.
이제 로지스틱 회귀를 이용해 학습 및 예측을 수행하고, 정확도와 ROC-AUC값을 구해보자. solver의 값을 lbfgs로 설정하고 성능을 확인할 것인데, defalt값이 lbfgs이다.
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score, roc_auc_score
lr_clf = LogisticRegression()
lr_clf.fit(X_train, y_train)
lr_pred = lr_clf.predict(X_test)
lr_proba = lr_clf.predict_proba(X_test)[:,1].reshape(-1,1)
accuracy = accuracy_score(y_test, lr_pred)
auc = roc_auc_score(y_test, lr_proba)
print(f"accuracy: {accuracy:.4f}")
print(f"auc: {auc:.4f}")'accuracy: 0.9766
auc: 0.9947Solver와 max_iter외에 주요 하이퍼 파라미터로는 penaly와 C가 있다.
- penalty : L1규제와 L2규제를 설정하며, 디폴트는
l2이다. - C : 규제 강도를 조절하는 alpha값의 역수로 C값이 작을수록 alpha는 크고, 규제강도도 커진다.
L1과 L2 규제의 경우 solver설정에 따라 영향을 받는다. Liblinear, saga의 경우 L1, L2 규제가 모두 가능하지만 lbfgs, newton-cg, sag의 경우에는 L2규제만 가능하다.
# 실행시 에러가 나야하는 부분인데도 나지 않아서 보니
# ignore해놓은 상태였다.. 풀어주기
warnings.filterwarnings('default')
from sklearn.model_selection import GridSearchCV
params = {
"solver" : ['liblinear', 'lbfgs'],
"penalty": ["l2", "l1"],
"C": [0.01, 0.1, 1, 5, 10]
}
lr_clf = LogisticRegression()
grid_cv = GridSearchCV(lr_clf, param_grid = params, scoring="accuracy", cv=3)
grid_cv.fit(cancer.data, cancer.target)
print("최적 하이퍼 파라미터:", grid_cv.best_params_)
print("최적 평균 정확도:", round(grid_cv.best_score_, 4))수많은 경고와 함께 아래가 출력되었다.
최적 하이퍼 파라미터: {'C': 10, 'penalty': 'l1', 'solver': 'liblinear'}
최적 평균 정확도: 0.9561이는 solver가 lbfgs일 때 L1 규제를 지원하지 않음에도 해당 규제값을 입력했기 때문에 나오는 메시지이다.
회귀 트리
결정 트리와 크게 다르지 않다. 회귀를 위한 트리를 생성하고 이를 기반으로 회귀 예측을 하는 것인데, 리프 노드에서 예측 결정 값을 만드는 과정에 있어 차이가 있다.
결정 트리는 특정 클래스의 레이블을 결정하지만, 회귀 트리는 리프 노드에 속한 데이터 값의 평균값을 구해 회귀 예측 값을 계산한다.
또한 결정트리는 각 영역의 지니계수가 낮은 피처를 기준으로 분할하며, 회귀 트리는 RSS를 가장 잘 줄일수 있는 피처를 기준으로 분할한다.
from sklearn.ensemble import RandomForestRegressor
from sklearn.datasets import load_boston
from sklearn.model_selection import cross_val_score
# 보스턴 데이터
boston = load_boston()
boston_df = pd.DataFrame(boston.data, columns = boston.feature_names)
boston_df['PRICE'] = boston.target
y_target = boston_df['PRICE']
X_data = boston_df.drop(['PRICE'], axis=1,inplace=False)
# RandomForestRegressor
rf = RandomForestRegressor(random_state=0, n_estimators=1000)
neg_mse_scores = cross_val_score(rf, X_data, y_target, scoring="neg_mean_squared_error", cv = 5)
rmse_scores = np.sqrt(-1 * neg_mse_scores)
avg_rmse = np.mean(rmse_scores)
print(f'5 교차 검증의 개별 Negative MSE scores: {np.round(neg_mse_scores, 2)}')
print(f'5 교차 검증의 개별 RMSE scores: {np.round(rmse_scores, 2)}')
print(f'5 교차 검증의 평균 RMSE : {avg_rmse:.3f}')5 교차 검증의 개별 Negative MSE scores: [ -7.88 -13.14 -20.57 -46.23 -18.88]
5 교차 검증의 개별 RMSE scores: [2.81 3.63 4.54 6.8 4.34]
5 교차 검증의 평균 RMSE : 4.423랜덤포레스트회귀를 이용해 앞의 선형회귀에서 다룬 보스턴 주택 가격 예측을 수행, RMSE를 구해봤다. 랜덤 포레스트 외에도 XGB, LGBM 등 분류에서 사용한 알고리즘은 회귀 트리로 사용 가능하다.
선형회귀와 회귀 트리
warnings.filterwarnings('ignore')
from sklearn.linear_model import LinearRegression
from sklearn.tree import DecisionTreeRegressor
from sklearn.ensemble import RandomForestRegressor
from sklearn.ensemble import GradientBoostingRegressor
from xgboost import XGBRegressor
from lightgbm import LGBMRegressor
# boston sample: 100, feature: RM
boston_df_sample = boston_df[['RM','PRICE']].sample(n=100, random_state=0)
X_feature = np.array(boston_df_sample.RM).reshape(-1,1)
y_target = np.array(boston_df_sample.PRICE).reshape(-1,1)
# test data: 4.5 ~ 8.5 , 100개의 데이터
X_test = np.arange(4.5, 8.5, 0.04).reshape(-1,1)
# 모델 객체 생성
lr_reg = LinearRegression()
rf_reg2 = DecisionTreeRegressor(max_depth=2)
rf_reg7 = DecisionTreeRegressor(max_depth=7)
# 학습/예측
lr_reg.fit(X_feature, y_target)
rf_reg2.fit(X_feature, y_target)
rf_reg7.fit(X_feature, y_target)
lr_pred = lr_reg.predict(X_test)
rf_reg2_pred = rf_reg2.predict(X_test)
rf_reg7_pred = rf_reg7.predict(X_test)
# 시각화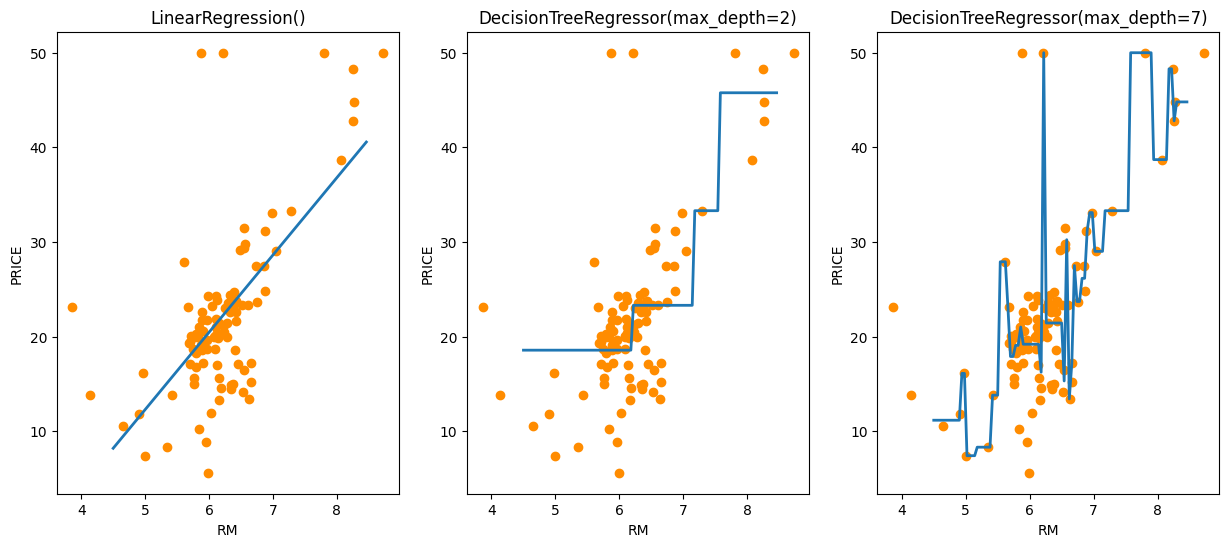
선형회귀는 직선으로 예측 회귀선을 표현하지만 회귀 트리의 경우 분할되는 데이터 지점에 따라 꺾이면서 계단 형태로 회귀선을 만든다. 차원이 높아진 경우 학습 데이터의 이상치 까지 학습하면서 복잡한 회귀선을 만든 모습을 확인할 수 있었고, 이경우 과적합이 되기 쉬운 모델이 되었음을 알 수 있다.
차원축소
매우 많은 피처로 구성된 다차원 데이터 세트의 차원을 축소해 새로운 차원의 데이터 셋을 생성하는 것이다. 일반적으로 차원이 증가할 수록 데이터 포인트 간의 거리가 기하급수적으로 멀어지게 되고, 데이터들 사이의 공간이 많이 비어있는 모습을 갖게 된다.
많은 다차원의 피처를 차원 축소해 피처수를 줄이면 더 직관적으로 데이터를 해석할 수 있다. 또한 학습 데이터의 크기가 줄어들어서 학습에 필요한 처리 능력도 줄일 수 있다.
PCA(Principal Component Analysis)
대표적인 차원 축소 기법 중 하나이다. 주성분 분석이라고도 함.
여러 변수간에 존재하는 상관관계를 이용해 이를 대표하는 주성분(Pricipal Component)를 추출해 차원을 축소한다. 축소시 기존 데이터 정보의 손실이 유실되는 것을 최소화히기 위해 가장 높은 분산을 가지는 데이터의 축을 찾아 이 축으로 차원을 축소하는데 이것이 PCA의 주성분이 된다. 일반적으로 차원 축소는 아래 두가지로 나눌 수 있다.
피처 선택(feature selection)
피처 선택, 즉 특성 선택은 말 그대로 특정 피처에 종속성이 강한 불필요한 피처는 아예 제거하고, 데이터의 특징을 잘 나타내는 주요 피처만 선택하는 것이다.피처 추출(feature extraction)
기존 피처를 저차원의 중요 피처로 압축하여 추출하는 것이다. 이렇게 새롭게 추출된 중요 특성은 기존의 치퍼가 압축된 것이므로.. 기존 피처와는 완전히 다른 값이 된다.
차원 축소는 단순히 데이터의 압축을 의미하는 것이 아니다. 더 중요한 것은 차원 축소를 통해 좀 더 데이터를 잘 설명할 수 있는 잠재적인 요소를 추출하는데에 있다. PCA, SVD, NMF는 이처럼 잠재적인 요소를 찾는 대표적인 차원 축소 알고리즘이다. 이 차원 축소 알고리즘은 매우 많은 픽셀로 이뤄진 이미지 데이터에서 잠재된 특성을 피처로 도출해 함축적 형태의 이미지 변환과 압축을 수행할 수 있다. 이렇게 변환된 이미지는 원본 이미지보다 훨씬 적은 차원이기 때문에 이미지 분류등의 분류 수행시 과적합 영향력이 작아져서 오히려 원본 데이터로 예측하는 것보다 예측 성능을 더 끌어올릴 수도 있다.
선형대수에서의 PCA
선형대수로 PCA를 해석하면 입력 데이터의 공분산 행렬을 고유값 분해하고, 구한 고유벡터에 입력 데이터를 선형 변환하는 것이다.
→ 고유벡터는 PCA의 주성분 벡터로서 입력 데이터의 분산이 큰 방향을 나타낸다
→ 고유값은 고유벡터의 크기를 나타내며 동시에 입력데이터의 분산을 나타낸다.
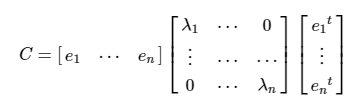
- 공분산 행렬 C는 (교유벡터 직교 행렬 고유값 정방행렬 고유벡터 직교행렬의 전치행렬)로 분해된다.
- ei , λi는 각각 i번째 고유벡터, 해당 고유벡터의 크기를 의미한다.
- e₁는 가장 분산이 큰 방향을 가진 고유벡터이며 e₂는 e₁에 수직이면서 다음으로 가장 분산이 큰 방향을 가진 고유벡터이다.
PCA 수행과정
- 입력 데이터 세트의 공분산 행렬을 생성한다
- 공분산 행렬의 고유벡터와 고유값을 계산한다
- 고유값이 가장 큰 순으로 K개(PCA변환 차수)만큼 고유벡터를 추출한다.
- 고유값이 가장 큰 순으로 추출된 고유벡터를 이용해 새롭게 입력 데이터를 변환한다.
구현하며 이해하기 - IRIS데이터
from sklearn.datasets import load_iris
import pandas as pd
import numpy as np
# 데이터 가져오기
iris = load_iris()
X_features = iris.data
y_labels = iris.targetiris(붓꽃) 데이터는 [ 0:sepal lenght, 1:sepal width, 2:petal length, 3:petal width ] 의 4가지 속성으로 이루어져있다. 원본 데이터가 어떻게 분포되어있는지 2차원으로 시각화해보자
iris_df['target'] = iris.target
markers = ["^", "s", "o"]
for i, marker in enumerate(markers) :
x_axis_data = iris_df[iris_df['target']==i][0]
y_axis_data = iris_df[iris_df['target']==i][1]
plt.scatter(x_axis_data, y_axis_data, marker=marker, label=iris.target_names[i])
plt.legend()
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.show()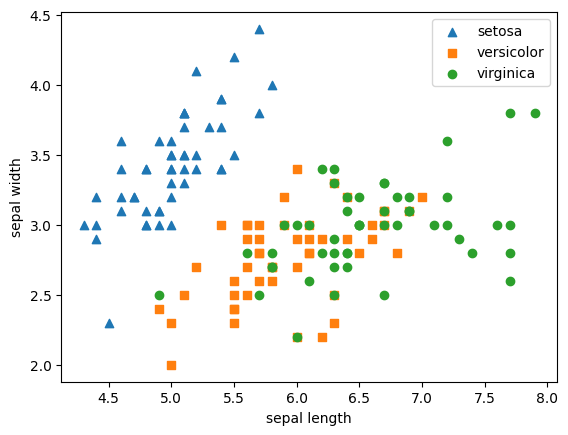
setosa의 경우 sepal width가 3이상, sepal length가 6이하인 곳에 대부분 분포되어 있고 versicolor, virginica는 sepal width, sepal length만으로는 분류하기 어려워 보인다.
PCA는 여러 피처 값을 연산하기 때문에 스케일에 영향을 받으므로 pca로 압축하기 전에 피처 스케일링 작업이 필요하다. 여기서는 표준정규분포(StandardScaler)로 변환하였다.
# Target 값을 제외하고 모든 속성값을 StandardScaler를 이용해 표준 정규 분포를 가지는 값들로 변환하자
from sklearn.preprocessing import StandardScaler
iris_scaled = StandardScaler().fit_transform(X_features)
iris_scaledarray([[-9.00681170e-01, 1.01900435e+00, -1.34022653e+00,
-1.31544430e+00],
[-1.14301691e+00, -1.31979479e-01, -1.34022653e+00,
-1.31544430e+00],
[-1.38535265e+00, 3.28414053e-01, -1.39706395e+00,
-1.31544430e+00],
[-1.50652052e+00, 9.82172869e-02, -1.28338910e+00,
-1.31544430e+00],
[-1.02184904e+00, 1.24920112e+00, -1.34022653e+00,
-1.31544430e+00],
..이하생략PCA를 이용해 기존 차원을 2차원으로 변환하자
from sklearn.decomposition import PCA
# 2차원 PCA데이터로 변환
pca = PCA(n_components=2)
pca.fit_transform(iris_scaled)
iris_pca = pca.transform(iris_scaled)
print(iris_pca)[[-2.26470281 0.4800266 ]
[-2.08096115 -0.67413356]
[-2.36422905 -0.34190802]
[-2.29938422 -0.59739451]
[-2.38984217 0.64683538]
[-2.07563095 1.48917752]
[-2.44402884 0.0476442 ]
[-2.23284716 0.22314807]
[-2.33464048 -1.11532768]
...이하생략print(iris_scaled.shape,'\n',iris_pca.shape)(150, 4)
(150, 2)원본 붓꽃 데이터 세트와 PCA로 변환한 데이터 세트의 정확도를 확인해보자
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
rf_clf = RandomForestClassifier(random_state=156)
orignal_data = cross_val_score(rf_clf, iris_scaled, y_labels, scoring='accuracy', cv=3)
pca_data = cross_val_score(rf_clf, iris_pca, y_labels, scoring='accuracy', cv=3)
print(f'원본 데이터 교차검증 개별 정확도 : {orignal_data}\n원본 데이터 평균 정확도 : {np.mean(orignal_data)}')
print()
print(f'PCA 변환 데이터 교차검증 개별 정확도 : {pca_data}\nPCA 변환 데이터 평균 정확도 : {np.mean(pca_data)}')원본 데이터 교차검증 개별 정확도 : [0.98 0.94 0.96]
원본 데이터 평균 정확도 : 0.96
PCA 변환 데이터 교차검증 개별 정확도 : [0.88 0.88 0.88]
PCA 변환 데이터 평균 정확도 : 0.88원본 데이터 세트 대비 예측 정확도는 PCA 변환 차원 개수에 따라 예측성능이 떨어질 수 밖에 없다. 실습결과 원본 데이터의 평균 정확도에 비해 PCA변환 데이터의 평균 정확도가 약 8% 하락한 것을 확인할 수 있었다. 속성개수가 절반 감소한 것을 고려한다면 변환후에도 원본 데이터의 특성을 상당부분 유지하고 있음을 알 수 있는 부분
실습 - UCI 신용카드 고객 데이터
!pip install xlrd
# credit card clients
import pandas as pd
# 1번 인덱스를 header로 삼고, 데이터가 있는 덱셀 시트명은 'Data'이다
df = pd.read_excel('./default of credit card clients.xls', header=1, sheet_name='Data').iloc[0:, 1:]
print(df.shape)
df[:3]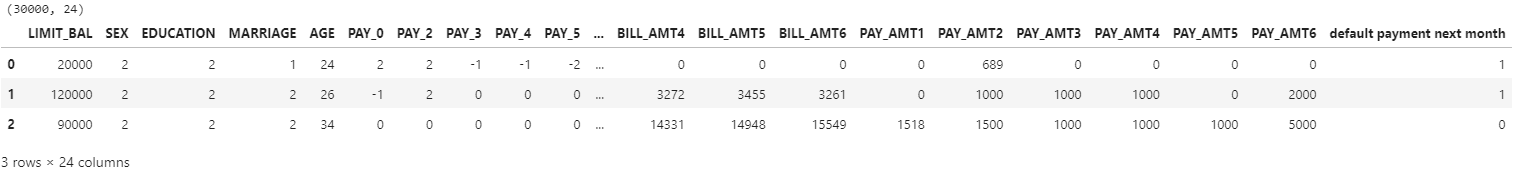
target으로 잡아야하는 컬럼은 default payment next month으로 다음달 연체 여부를 의미한다(0은 정상납부, 1은 연체)
PAY_0 다음에 PAY_2가 나와 있어 PAY_0를 PAY_1으로 변경하고 target의 컬럼명도 default로 변경한 후 상관관계를 보도록 하자
# 컬럼명 변경
df.rename(columns={'PAY_0':'PAY_1','default payment next month':'default'}, inplace=True)
y_target = df['default']
X_features = df.drop('default', axis=1)
# 상관관계 보기
corr = X_features.corr()
corr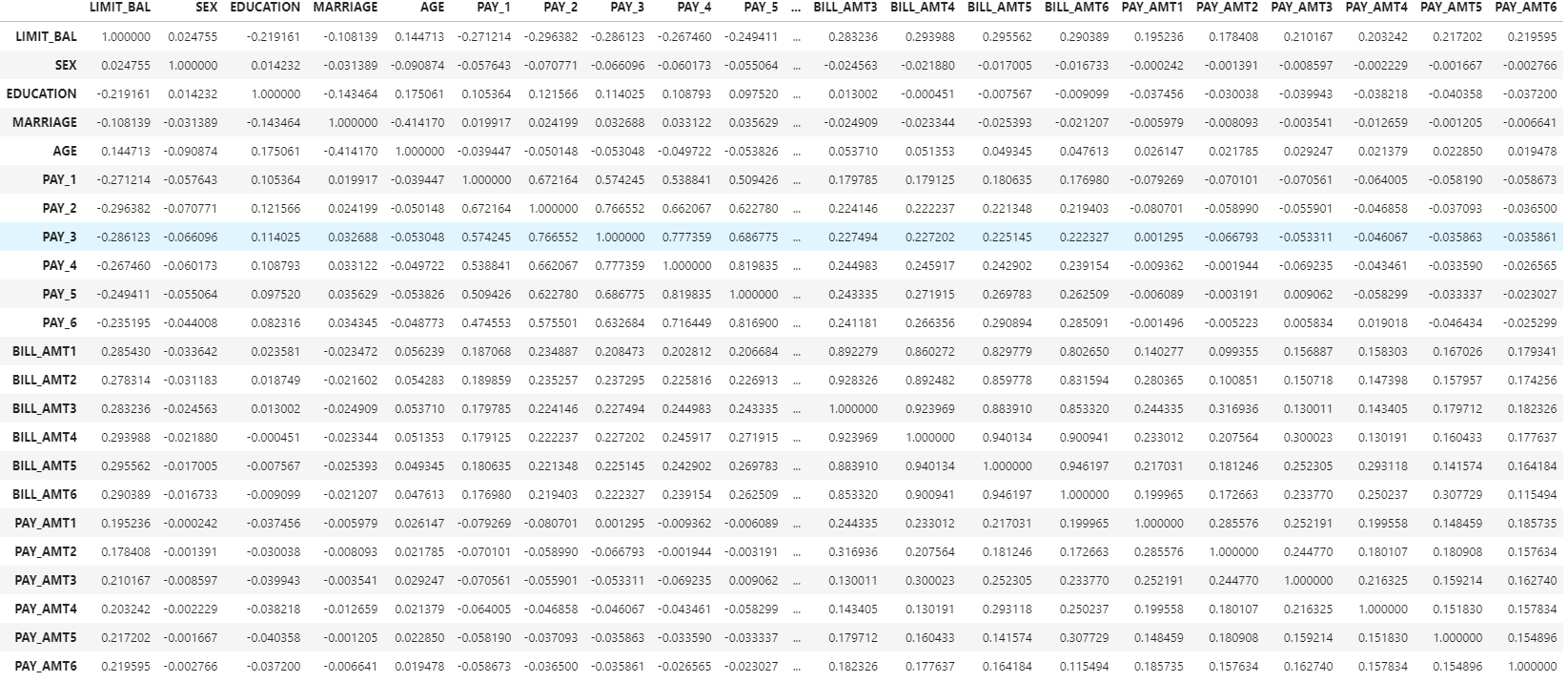
어...... 한눈에 안보인다. 예상은 했지만 더 안보인다. 이래서 책에서는 heatmap으로 시각화하여 보라고 했나보다
import seaborn as sns
import matplotlib.pyplot as plt
plt.figure(figsize=(14, 14))
sns.heatmap(corr, annot=True, fmt='.1g')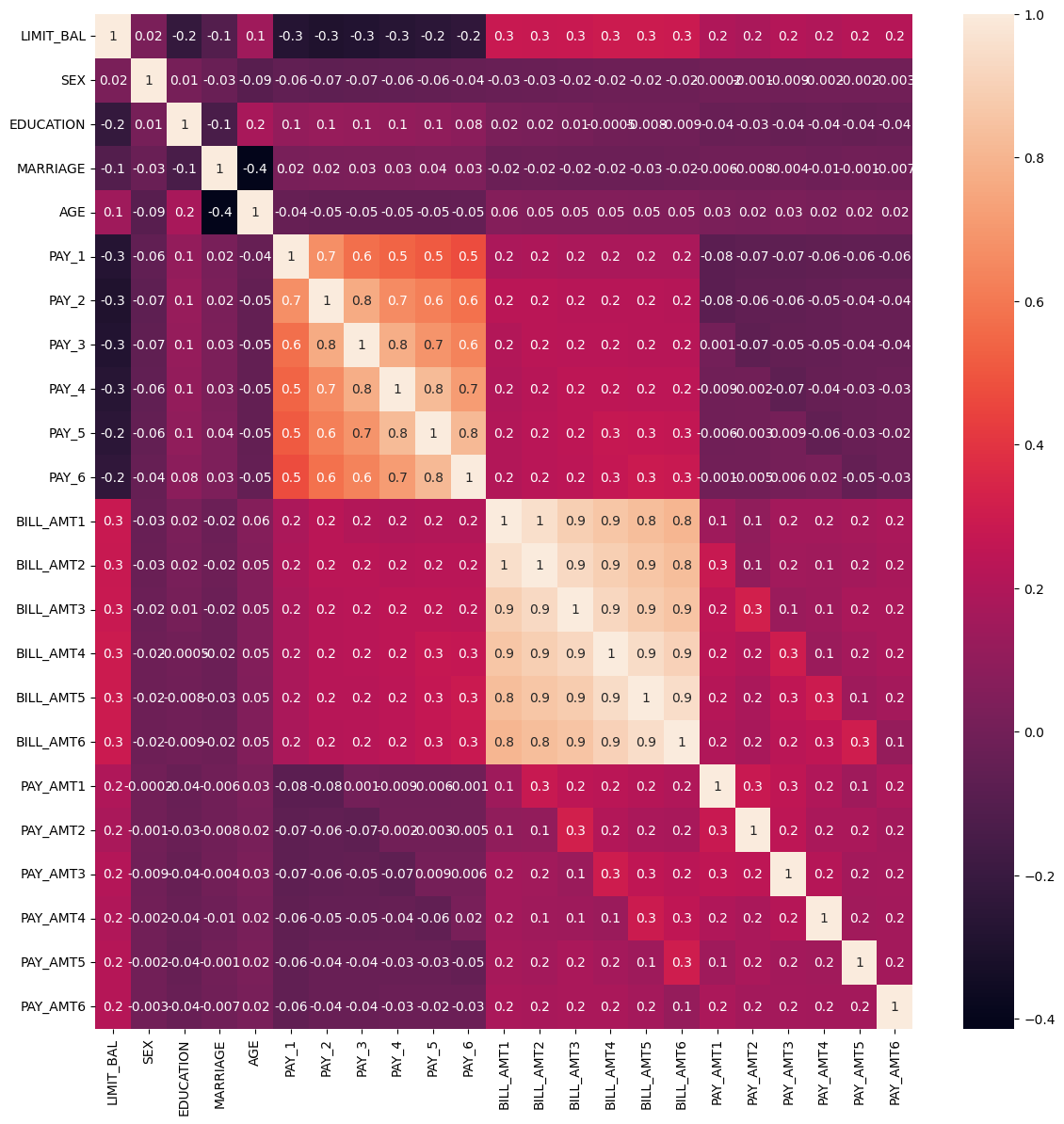
BILL_AMT1 ~ BILL_AMT6 간의 상관계수가 높은 것으로 보인다. 이렇게 높은 상관돌르 가진 속성들은 소수의 PXA만으로도 자연스럽게 이 속성들의 변동성을 수용할 수 있으니 솎아내자
# BILL_AMT1~6만 솎아내기
df.info()
# 이름도 변경할 겸 데이터 안전을 위해 df를 카피하여주자
card_df = df.copy()
# BILL_AMT1 ~ 6까지의 6개 속성만 뽑아주기
card_df = card_df.iloc[:, 11:17]
card_df
이제 순서대로 변환을 진행하면 된다.
1. scaling 변환
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
card_bill_scaler = scaler.fit_transform(X_features)
card_bill_scalerarray([[-1.13672015, 0.81016074, 0.18582826, ..., -0.30806256,
-0.31413612, -0.29338206],
[-0.3659805 , 0.81016074, 0.18582826, ..., -0.24422965,
-0.31413612, -0.18087821],
[-0.59720239, 0.81016074, 0.18582826, ..., -0.24422965,
-0.24868274, -0.01212243],
...,
[-1.05964618, -1.23432296, 0.18582826, ..., -0.03996431,
-0.18322937, -0.11900109],
[-0.67427636, -1.23432296, 1.45111372, ..., -0.18512036,
3.15253642, -0.19190359],
[-0.90549825, -1.23432296, 0.18582826, ..., -0.24422965,
-0.24868274, -0.23713013]])- pca 변환
from sklearn.decomposition import PCA
# 2차원 PCA데이터로 변환
pca = PCA(n_components=2)
card_bill_pca = pca.fit_transform(card_bill_scaler)
print(card_bill_pca)[[-1.88796247 -0.9061075 ]
[-0.76469577 -2.10928777]
[-0.84740789 -1.0721793 ]
...
[ 0.35745734 -3.31275505]
[ 0.65055187 0.72290108]
[-0.14556442 -0.80975087]]- pca 변환 이전에 대하여 logisticregression으로 분류
from sklearn.linear_model import LogisticRegression
from sklearn.model_selection import cross_val_score
lr_clf = LogisticRegression(random_state=156)
cross_val_score(lr_clf, card_bill_scaler, y_target, scoring='accuracy', cv=3)
# cv에 따른 차이도 궁금하여 돌려봄
# (cv=5) array([0.803 , 0.806 , 0.8125 , 0.81616667, 0.812 ])
# (cv=3) array([0.806 , 0.8138, 0.8086])- pca 변환한 logisticRegression으로 분류
cross_val_score(lr_clf, card_bill_pca, y_target, scoring='accuracy', cv=3)
# cv에 따른 차이도 궁금하여 돌려봄
# (cv=5) array([0.792 , 0.79316667, 0.79466667, 0.79966667, 0.797 ])
# (cv=3) array([0.7936, 0.7969, 0.7959])결과적으로
print(card_bill_scaler.shape,'\n',card_bill_pca.shape)(30000, 23)
(30000, 2)전체 23개 속성의 약 25%수준인 6개의 PCA컴포넌트 만으로도 원본 데이터를 기반으로 한 분류예측 결과에 비해 약간의 예측 성능 저하를 보였다. 작은 수치는 아니지만 전체 속성의 25%만으로도 이 정도의 예측 성능을 유지할 수 있다는 것은 PCA의 뛰어난 압축 능력을 잘 보여주는 것이라 생각해볼 수 있다.