위키북스의 '파이썬 머신러닝 완벅 가이드' 개정 2판으로 공부하고 있습니다. 실습 과정과 이해에 따라 내용의 누락 및 코드의 변형이 있을 수 있습니다.
군집평가(Cluster Evaluation)
분류(Classification)와 비슷해 보일수는 있지만 그 성격이 많이 다르다.
붓꽃 데이터 세트의 경우 결과값이 저장된 타깃 레이블이 있었지만 대부분의 군집화 데이터 세트는 이런식으로 비교할 만한 타깃 레이블을 가지고 있지 않다.
- 데이터 내에 숨어있는 별도의 그룹을 찾아서 의미를 부여한다. 동일한 분류 값에 속하더라도 그 안에서 더 세분화된 군집화를 추구하기도 한다. 서로 다른 분류 값의 데이터도 더 넓은 군집화 레벨화 등의 영역이 있다.
→ 이러한 군집화의 성능을 평가하는 대표적인 방법으로 실루엣 분석이 있다. (다만 비지도 학습의 특성상 어떤 지표라도 정확하게 성능을 평가하기는 어렵다)
실루엣 분석(Silhouette analysis)
각 군집 간의 거리가 얼마나 효율적으로 분리되어 있는지를 나타낸다. 다른 군집과의 거리는 떨어져있고, 동일 군집 데이터끼리는 얼마나 서로 가까운지를 나타내는 것이다. 실루엣 분석은 실루엣 계수를 기반으로 한다.
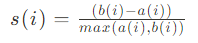
실루엣 계수
- Silhouette coefficient
- a(i) : i번째 데이터에서 자신이 속한 군집내의 다른 데이터까지의 거리들의 평균
- b(i) : i번째 데이터에서 가장 가까운 타 군집내의 다른 데이터까지의 거리들의 평균
- 개별 데이터가 가지는 군집화 지표로, 해당 데이터가 같은 군집 내의 데이터와 얼마나 가깝게 군집화 되어있고,
다른 군집에 있는 데이터와는 얼마나 멀리 분리되어 있는지를 나타내는 지표이다.
- -1 ~ 1 사이의 값을 가지며 1에 가까울 수록 근처 군집과 멀리 떨어져 있다는 의미이다.
- 0에 가까울 수록 근처 군집과 가까워진다는 것이고, 음수값은 i번째 데이터가 아예다른 군집에 할당되었다는 의미이다.붓꽃 데이터 세트를 이용한 군집 평가
silhouette_samples()를 사용하면 개별 데이터의 실루엣 계수를 구할 수 있고, silhouette_score()는 전체 실루엣 계수의 평균으로 silhouette_samples()의 평균과 같다. 두 함수 모두 인자로 개별 데이터(피처)와 군집 정보를 입력 받는다.
import numpy as np
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.datasets import load_iris
from sklearn.cluster import KMeans
# iris 데이터
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
iris_df = pd.DataFrame(data=iris.data, columns=feature_names)
# KMeans
kmeans = KMeans(n_clusters=3, init="k-means++", max_iter=300, random_state=0)
kmeans.fit(iris_df)
# 데이터 프레임에 cluster 추가
iris_df["cluster"] = kmeans.labels_
from sklearn.metrics import silhouette_samples, silhouette_score
# 개별 데이터 실루엣 계수
score_samples = silhouette_samples(iris.data, iris_df.cluster)
print(f'silhouette_samples( ) return 값의 shape: {score_samples.shape}')
# 데이터 프레임에 실루엣 계수 추가
iris_df['silhouette_coeff'] = score_samples
# 모든 데이터의 평균 실루엣 계수
average_score = silhouette_score(iris.data, iris_df.cluster)
print(f'붓꽃 데이터셋 Silhouette Analysis Score: {average_score:.3f}')silhouette_samples( ) return 값의 shape: (150,)
붓꽃 데이터셋 Silhouette Analysis Score: 0.553평균 실루엣 계수 값이 약 0.553으로 나왔다.
iris_df.groupby("cluster").mean()[["silhouette_coeff"]]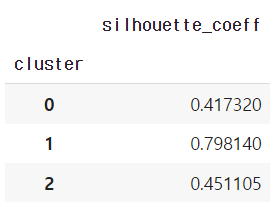
iris_df에서 군집 칼럼별로 그룹지어서 칼럼의 평균값을 구해보니 위와 같이 나왔다. 1번군집의 평균은 약 0.8ㄹ로 높은 편이지만 0, 2번 군집의 평균값은 0.41, 0.45로 상대적으로 낮은 것을 알 수 있었음.
군집 개수 최적화
전체 데이터의 평균 실루엣 계수 값이 높다고 해서 반드시 최적의 군집 개수로 군집화가 잘 되었다고 볼 수는 없다. 특정 군집 내의 실루엣 계수 값만 너무 높고, 다른 군집은 내부 데이터 끼리의 거리가 멀어 계수가 낮아도 전체 평균은 높을 수 있기 때문이다.
군집끼리 적당히 거리를 유지하면서도 군집 내의 데이터가 서로 뭉쳐있어야 적절한 군집 개수가 설정되었다고 할 수 있다.
실루엣 계수 시각화 함수-1
사이킷런의 문서에 있는 시각화 소스코드 를 사용하여 보자
def visualize_silhouette_sklearn(range_n_clusters, X):
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import numpy as np
for n_clusters in range_n_clusters:
# Create a subplot with 1 row and 2 columns
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(18, 7)
# The 1st subplot is the silhouette plot
# The silhouette coefficient can range from -1, 1 but in this example all
# lie within [-0.1, 1]
ax1.set_xlim([-0.1, 1])
# The (n_clusters+1)*10 is for inserting blank space between silhouette
# plots of individual clusters, to demarcate them clearly.
ax1.set_ylim([0, len(X) + (n_clusters + 1) * 10])
# Initialize the clusterer with n_clusters value and a random generator
# seed of 10 for reproducibility.
clusterer = KMeans(n_clusters=n_clusters, random_state=10)
cluster_labels = clusterer.fit_predict(X)
# The silhouette_score gives the average value for all the samples.
# This gives a perspective into the density and separation of the formed
# clusters
silhouette_avg = silhouette_score(X, cluster_labels)
# Compute the silhouette scores for each sample
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
# Aggregate the silhouette scores for samples belonging to
# cluster i, and sort them
ith_cluster_silhouette_values = \
sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_clusters)
ax1.fill_betweenx(np.arange(y_lower, y_upper),
0, ith_cluster_silhouette_values,
facecolor=color, edgecolor=color, alpha=0.7)
# Label the silhouette plots with their cluster numbers at the middle
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
# Compute the new y_lower for next plot
y_lower = y_upper + 10 # 10 for the 0 samples
ax1.set_title('Number of Cluster : '+ str(n_clusters)+'\n' \
'Silhouette Score :' + str(round(silhouette_avg,3)))
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
# The vertical line for average silhouette score of all the values
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([]) # Clear the yaxis labels / ticks
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
# 2nd Plot showing the actual clusters formed
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(X[:, 0], X[:, 1], marker='.', s=30, lw=0, alpha=0.7,
c=colors, edgecolor='k')
# Labeling the clusters
centers = clusterer.cluster_centers_
# Draw white circles at cluster centers
ax2.scatter(centers[:, 0], centers[:, 1], marker='o',
c="white", alpha=1, s=200, edgecolor='k')
for i, c in enumerate(centers):
ax2.scatter(c[0], c[1], marker='$%d$' % i, alpha=1,
s=50, edgecolor='k')
ax2.set_title("The visualization of the clustered data.")
ax2.set_xlabel("Feature space for the 1st feature")
ax2.set_ylabel("Feature space for the 2nd feature")
plt.suptitle(("Silhouette analysis for KMeans clustering on sample data "
"with n_clusters = %d" % n_clusters),
fontsize=14, fontweight='bold')
plt.show()# 가상데이터: 피처 2개, 클러스터 4개
from sklearn.datasets import make_blobs
X, y = make_blobs(n_samples=500, n_features=2, centers=4, cluster_std=1,
center_box=(-10.0, 10.0), shuffle=True, random_state=1)
# K-Means K: 2
visualize_silhouette_sklearn([2], X)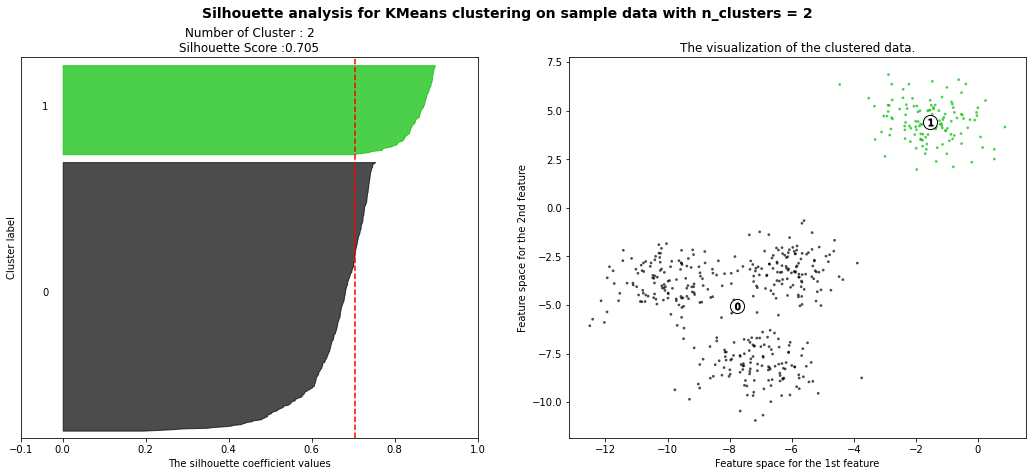
주어진 데이터에 대해서 군집의 개수를 2개로 설정했을 때 이다. 이때 평균 실루엣 계수는 약 0.705로 꽤 높게 나타났다.
왼쪽 그림은 개별 군집에 속하는 데이터의 실루엣 계수를 2차원으로 나타낸 것이다. X축은 실루엣 계수값이고 Y축은 개별 군집과 이에 속하는 데이터인데, 개별 군집은 Y축에 숫자 값으로 0, 1로 표시되어 있습니다. 이에 해당하는 데이터는 일일이 숫자 값으로 표시되지는 않았지만 Y축의 높이로 추측해볼 수 있다. 점선으로 표시된 선은 전체 평균 실루엣 계수값을 나타낸다.
오른쪽의 그림을 보면 시각화를 더 쉽게 이해할 수 있었는데 1번 군집은 비교적 데이터들이 잘 모여있고 다른 군집과 멀리 떨어져 있는 모습이다. 하지만 0번 군집의 경우는 내부 데이터끼리 흩어져 있는 걸 확인해볼 수 있었다.
만약 군집이 4개인 경우라면 어떨까
visualize_silhouette_sklearn([4], X)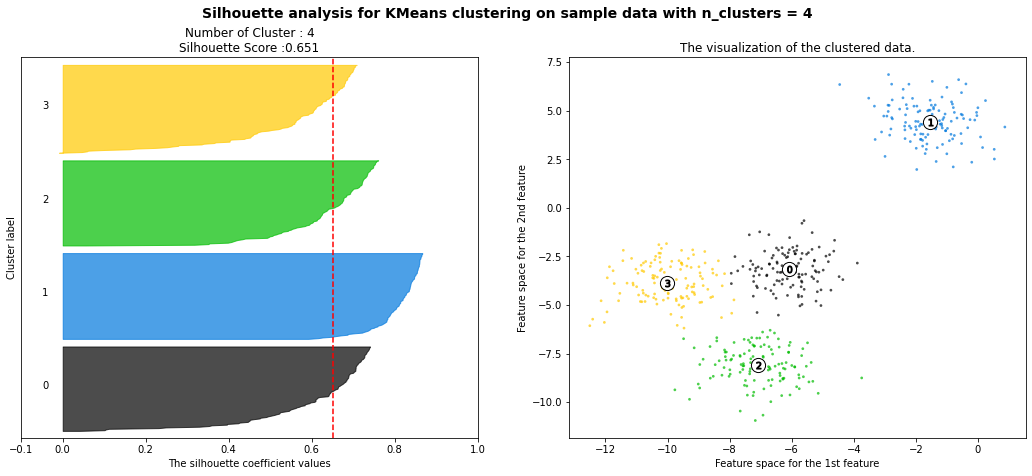
평균 실루엣 계수 값은 0.651이 나왔다. 개별 군집의 평균 실루엣 계수값이 비교적 균일하게 (각 그래프가 Y축을 차지하고 있는 범위가 꽤 균일) 위치하고 있는 것을 확인할 수 있었고, 모든 데이터가 평균보다는 높은 계수값을 가지고 있는 모습이였다.
실루엣 계수 시각화 함수-2
책 부록으로 제공되는 소스코드에 있는, 원본 소스 코드를 좀 더 간략하게 커스터 마이징한 소스코드이다. 위 사이킷런 코드에서 왼쪽 그림의 군집별 평균 실루엣 계수값을 구하는 부분만 별도의 함수로 만들어 놓은 것이다.
### 여러개의 클러스터링 갯수를 List로 입력 받아 각각의 실루엣 계수를 면적으로 시각화한 함수 작성
def visualize_silhouette(cluster_lists, X_features):
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import math
# 입력값으로 클러스터링 갯수들을 리스트로 받아서, 각 갯수별로 클러스터링을 적용하고 실루엣 개수를 구함
n_cols = len(cluster_lists)
# plt.subplots()으로 리스트에 기재된 클러스터링 수만큼의 sub figures를 가지는 axs 생성
fig, axs = plt.subplots(figsize=(4*n_cols, 4), nrows=1, ncols=n_cols)
# 리스트에 기재된 클러스터링 갯수들을 차례로 iteration 수행하면서 실루엣 개수 시각화
for ind, n_cluster in enumerate(cluster_lists):
# KMeans 클러스터링 수행하고, 실루엣 스코어와 개별 데이터의 실루엣 값 계산.
clusterer = KMeans(n_clusters = n_cluster, max_iter=500, random_state=0)
cluster_labels = clusterer.fit_predict(X_features)
sil_avg = silhouette_score(X_features, cluster_labels)
sil_values = silhouette_samples(X_features, cluster_labels)
y_lower = 10
axs[ind].set_title('Number of Cluster : '+ str(n_cluster)+'\n' \
'Silhouette Score :' + str(round(sil_avg,3)) )
axs[ind].set_xlabel("The silhouette coefficient values")
axs[ind].set_ylabel("Cluster label")
axs[ind].set_xlim([-0.1, 1])
axs[ind].set_ylim([0, len(X_features) + (n_cluster + 1) * 10])
axs[ind].set_yticks([]) # Clear the yaxis labels / ticks
axs[ind].set_xticks([0, 0.2, 0.4, 0.6, 0.8, 1])
# 클러스터링 갯수별로 fill_betweenx( )형태의 막대 그래프 표현.
for i in range(n_cluster):
ith_cluster_sil_values = sil_values[cluster_labels==i]
ith_cluster_sil_values.sort()
size_cluster_i = ith_cluster_sil_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_cluster)
axs[ind].fill_betweenx(np.arange(y_lower, y_upper), 0, ith_cluster_sil_values, \
facecolor=color, edgecolor=color, alpha=0.7)
axs[ind].text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
axs[ind].axvline(x=sil_avg, color="red", linestyle="--")
# iris 군집 개수 최적화
visualize_silhouette([ 2, 3, 4, 5 ], iris.data)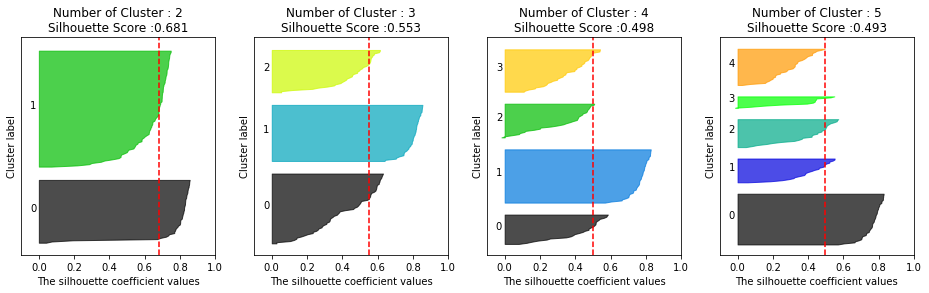
군집의 갯수를 2개로 했을 때, 전체 실루엣 계수 평균이 가장 높고 군집별 실루엣 계수 평균도 비교적 균일하게 보인다.
평균이동
평균이동(mean shift)는 KMeans와 유사하게 군집의 중심으로 지속적으로 움직이며 군집화를 수행한다. 차이점은 데이터가 모여있는, 밀도가 가장 높은 곳으로 중심을 이동시킨다는 점이다.
- K-means : 중심에 소속된 데이터의 평균 거리 중심으로 이동한다.
- 평균 이동 : 데이터가 모여있는 밀도가 가장 높은 곳으로 중심으로 이동한다.
수행과정
- 평균 이동은 KDE(Kernerl Density Estimation)를 이용해서 확률 밀도 함수를 구한다.
- 데이터가 집중적으로 모여있어 확률 밀도 함수가 피크인 점을 군집 중심점으로 선정
- 이런 방식을 전체 데이터에 반복 적용하면서 데이터의 군집 중심점을 찾는다.
KDE(Kernerl Density Estimation)
커널 함수를 통해 어떤 변수의 확률 밀도 함수를 추정하는 대표적 방식. 관측된 데이터 각각에 커널 함수를 적용한 값을 모두 더한 뒤 데이터 건수로 나눠 확률 밀도 함수를 추정한다.
개별 관측 데이터에 커널 함수를 적용한 뒤, 이 적용 값을 모두 더한 후 데이터의 건수로 나눠 확률 밀도 함수를 추정한다.
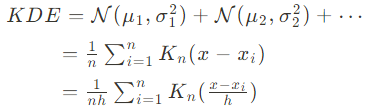
K : 커널함수
x : 확률 변수값
xi : 관측값
h : 대역폭(bandwidth)대역폭은 KDE 형태를 결정하는 요소로 어떻게 설정하느냐에 따라 확률 밀도 추정 성능을 좌우한다. 대역폭이 작을수록 좁고 뾰족한 KDE를 가지며 이는 변동성이 큰 방식으로 확률 밀도 함수를 추정해 과적합(over-fitting)하기 쉽다. 반대로 값이 클수록 넓고 완만한 KDE로 지나치게 단순화하여 과소적합되기가 쉽다.
→ 모델의 성능이 h에 따라서 좌우되기 때문에 평균 이동(mean shift)의 군집화에 있어서 h를 계산하는 것은 매우 중요한 일이다.
일반적으로 평균 이동 군집화는 대역폭이 클수록 평활화된 KDE로 인해 적은 수의 군집 중심점을 가지며 대역폭이 적을수록 많은 수의 군집 중심점을 가진다. 또한 K-Means와는 달리 군집의 개수를 지정하지 않고 대역폭에 따라 군집화를 수행한다.
장단점
데이터를 특정 형태로 가정하거나 특정 분포 기반 모델로 가정하지 않으면 유연한 군집화가 가능하며, 이상치의 영향력이 크지 않으며 군집의 개수를 정할 필요가 없다.
하지만 알고리즘의 수행시간이 오래 걸리고 대역폭(bandwidth)의 크기에 따른 군집화 영향이 매우 크다.
예제
make_blobs()의 cluster_std를 0.7로 정한 3개의 군집 데이터에 대해 bandwidth를 0.8로 설정한 평균 이동 군집화 알고리즘을 적용한 예제이다.
import numpy as np
from sklearn.datasets import make_blobs
from sklearn.cluster import MeanShift
# 클러스터 3개인 가상데이터
X, y = make_blobs(n_samples=200, n_features=2, centers=3, cluster_std=0.7, random_state=0)
# 평균이동
meanshift = MeanShift(bandwidth = 0.8)
cluster_labels = meanshift.fit_predict(X)
print(f"Mean Shift Cluster 유형: {np.unique(cluster_labels)}")Mean Shift Cluster 유형: [0 1 2 3 4 5]군집이 3개인 데이터로 평균 이동을 했지만, 6개의 군집으로 지나치게 세분화 된 모습
meanshift = MeanShift(bandwidth = 2.5)
cluster_labels = meanshift.fit_predict(X)
print(f"Mean Shift Cluster 유형: {np.unique(cluster_labels)}")Mean Shift Cluster 유형: [0 1]대역폭을 더 증가시키니 2개의 군집이 되었다. 이런 식으로 bandwidth의 값은 군집화 개수에 큰영향을 미치므로 MeanShift에서는 이 bandwidth를 최적화 값으로 설정하는 것이 매우 중요하다.
사이킷런은 최적화된 값을 찾기 위해 estimate_bandwidth()함수를 제공한다. 이는 파라미터로 피처 데이터 세트를 입력해주면 최적화된 값을 반환해준다!
from sklearn.cluster import estimate_bandwidth
bandwidth = estimate_bandwidth(X)
print('bandwidth 값:', round(bandwidth,3))bandwidth 값: 1.816뭔가 결과가 출력되었다. 이 값으로 다시 평균이동을 진행해보면
meanshift = MeanShift(bandwidth = 1.816)
cluster_labels = meanshift.fit_predict(X)
print(f"Mean Shift Cluster 유형: {np.unique(cluster_labels)}")Mean Shift Cluster 유형: [0 1 2]최적 대역폭으로 평균이동시 3개의 군집으로 군집화 된것을 확인할 수 있었으며 이를 시각화 해 살펴본다면
# 데이터 프레임 생성
cluster_df = pd.DataFrame(data=X, columns=['ftr1', 'ftr2'])
cluster_df['target'] = y
cluster_df["meanshift_label"] = cluster_labels
# 클러스터별 중심 위치 좌표
centers = meanshift.cluster_centers_
# cluster 값 종류
unique_labels = np.unique(cluster_labels)
markers=['o', 's', '^', 'P','D','H','x']
for cluster in unique_labels:
# 각 군집 시각화
cluster_v = cluster_df[cluster_df['meanshift_label'] == cluster]
plt.scatter(x=cluster_v['ftr1'], y=cluster_v['ftr2'], edgecolor='k', marker=markers[cluster] )
# 군집별 중심 위치 시각화
center_xy = centers[cluster]
plt.scatter(x = center_xy[0], y = center_xy[1], s=300, color='white',
alpha=0.9, edgecolor='k', marker = markers[cluster])
plt.scatter(x = center_xy[0], y = center_xy[1], s=70, color='k',
edgecolor='k', marker = f"${cluster}$")
plt.show()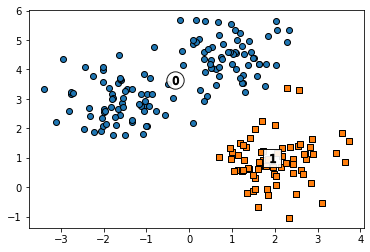
cluster_df.groupby(['target','meanshift_label']).size()target meanshift_label
0 0 65
1 2
1 1 67
2 0 66
dtype: int64평균이동은 K-Means처럼 중심으로 가지고 있으므로 위처럼 clustercenters 속성 사용이 가능하다. 꽤나 군집이 잘 되었음을 확인 할 수 있다.
GMM
- Gaussian Mixture Model
- 데이터가 여러개의 정규 분포를 가진 데이터 집합들이 섞여서 생성된 것이라는 가정하에 군집화를 수행한다. 데이터가 퍼진 모양이 원형이 아니라면 K-Means의 정확도는 매우 낮아지게 되는데, 이런 점을 극복하기 위해서 GMM 군집화는 데이터가 여러개의 가우시안 분포를 가진 데이터의 집합들이 섞여서 생성된 것이라는 가정하에 군집화를 수행하는 방법이다.
- GMM은 여러 개의 정규 분포 곡선을 추출하고, 개별 데이터가 그 중 어떤 정규 분포에 속하는 지 결정한다. 이와 같은 방식은 GMM에서 모수추정이라 불리는데 이는 대표적으로 2가지를 추정한다. (개별 정규 분포의 평균과 분산 / 각 데이터가 어떤 정규 분포에 해당되는지의 확률)
GMM을 이용한 붓꽃 데이터 세트 군집화
GMM은 확률 기반 군집화이고 K-Means는 거리 기반 군집화이다. 이를 비교해자
from sklearn.datasets import load_iris
# 데이터 로드
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
iris_df = pd.DataFrame(iris.data, columns = feature_names)
iris_df["target"] = iris.target
iris_df[:3]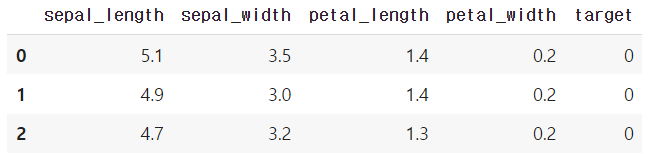
GaussianMixture 객체의 가장 중요한 초기화파라미터는 n_components 이다. 이는 gaussian mixture 모델의 총 개수로 K-Means의 n_clusters와 같이 군집의 개수를 정하는데 중요한 역할을 수행한다.
from sklearn.mixture import GaussianMixture
# GMM: n_components = 모델의 총 수
gmm = GaussianMixture(n_components=3, random_state=0)
gmm.fit(iris.data)
gmm_cluster_labels = gmm.predict(iris.data)
# target, gmm_cluster 비교
iris_df["gmm_cluster"] = gmm_cluster_labels
iris_df.groupby(["target","gmm_cluster"]).size()target gmm_cluster
0 0 50
1 1 5
2 45
2 1 50
dtype: int64gmm의 경우 ↑
kmeans의 경우 ↓
kmeans = KMeans(n_clusters=3, init='k-means++', max_iter=300, random_state=0).fit(iris.data)
kmeans_cluster_labels = kmeans.predict(iris.data)
iris_df['kmeans_cluster'] = kmeans_cluster_labels
iris_df.groupby(["target","kmeans_cluster"]).size()target kmeans_cluster
0 1 50
1 0 48
2 2
2 0 14
2 36
dtype: int64위와 같은 어떤 알고리즘에 더 뛰어나다는 의미가 아니라 붓꽃 데이터 세트가 GMM 군집화에 더 효과적이라는 의미로 볼 수 있다.
DBSCAN
DBSCAN(Density Based Spatial Clustering of Applications with Noise)는 밀도 기반 군집화의 대표적인 알고리즘이다. 특정 공간 내에 데이터 밀도 차이를 기반 알고리즘으로 하고 있어 복잡한 기하학적 분포를 가진 데이터에도 군집화를 잘 수행한다.
두 가지 주요 파라미터
- 입실론 주변 영역(epsilon) : 개별 데이터를 중심으로 입실론 반경을 가지는 원형의 영역
- 최소 데이터 개수(min points) : 개별 데이터의 입실론 주변 영역에 포함되는 타 데이터의 개수
데이터 포인트
입실론 주변 영역 내에 포함되는 최소 데이터 개수를 충족시키는가 아닌가에 따라 데이터 포인트를 아래와 같이 정의할 수 있다.
- 핵심 포인트(Core Point) : 주변 영역 내에 최소 데이터 갯 이상의 타 데이터를 가지고 있을 경우
- 이웃 포인트(Neighbor Point) : 주변 영역 내에 위치한 타 데이터
- 경계 포인트(border point): 핵심 포인트는 아니지만 핵심 포인트를 이웃 포인트로 가지는 데이터
- 잡음 포인트(noise point): 핵심 포인트가 아니면서 동시에 경계 포인트도 아닌 데이터
입실론 주변 영역의 최소 데이터 개수를 포함하는 밀도 기준을 충족시키는 데이터인 '핵심 포인트'를 연결하면서 군집화를 구선하는 방식이다.
실습하며 이해하기
DBSCAN 클래스를 이용해 붓꽃 데이터 세트를 군집화. 일반적으로 eps 값으로는 1 이하의 값을 설정한다.
from sklearn.datasets import load_iris
# 데이터 로드하기
iris = load_iris()
feature_names = ['sepal_length','sepal_width','petal_length','petal_width']
iris_df = pd.DataFrame(iris.data, columns = feature_names)
iris_df["target"] = iris.target
from sklearn.cluster import DBSCAN
# dbscan
dbscan = DBSCAN(eps = 0.6, min_samples = 8, metric = "euclidean")
dbscan_labels = dbscan.fit_predict(iris.data)
# cluster label 추가
iris_df["dbscan_cluster"] = dbscan_labels
iris_df.groupby(["target", "dbscan_cluster"]).size()target dbscan_cluster
0 -1 1
0 49
1 -1 4
1 46
2 -1 8
1 42
dtype: int64출력을 보면 특이하게 -1이 군집 레이블로 있는 것을 알수 있는데, 이 -1은 노이즈에 속하는 군집을 의미한다. 따라서 위 붓꽆 데이터 세트은 0과 1 두개의 군집으로 군집화 되었다는 것으로 볼 수 있다.
DBSCAN은 군집의 개수를 알고리즘에 따라 자동으로 지정하므로 DBSCAN에서 군집의 개수를 지정하는 것은 무의미하다고 할 수 있다. 시각화 하여 보자
# GMM에서 사용한 시각화 함수
def visualize_cluster_plot(clusterobj, dataframe, label_name, iscenter=True):
# 군집별 중심 위치: K-Means, Mean Shift 등
if iscenter:
centers = clusterobj.cluster_centers_
# Cluster 값 종류
unique_labels = np.unique(dataframe[label_name].values)
markers=['o', 's', '^', 'x', '*']
isNoise=False
for label in unique_labels:
# 군집별 데이터 프레임
label_cluster = dataframe[dataframe[label_name]==label]
if label == -1:
cluster_legend = 'Noise'
isNoise=True
else:
cluster_legend = 'Cluster '+str(label)
# 각 군집 시각화
plt.scatter(x=label_cluster['ftr1'], y=label_cluster['ftr2'], s=70,
edgecolor='k', marker=markers[label], label=cluster_legend)
# 군집별 중심 위치 시각화
if iscenter:
center_x_y = centers[label]
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=250, color='white',
alpha=0.9, edgecolor='k', marker=markers[label])
plt.scatter(x=center_x_y[0], y=center_x_y[1], s=70, color='k',\
edgecolor='k', marker='$%d$' % label)
if isNoise:
legend_loc='upper center'
else:
legend_loc='upper right'
plt.legend(loc=legend_loc)
plt.show()
from sklearn.decomposition import PCA
# 2차원 평면에서 표현하기 위해 n_components=2로 피처 데이터 셋 변환
pca = PCA(n_components=2, random_state=0)
pca_transformed = pca.fit_transform(iris.data)
# visualize_cluster_2d 함수는 ftr1, ftr2칼럼을 좌표에 표현하므로 PCA 변환값을 해당 칼럼으로 생성
iris_df["ftr1"] = pca_transformed[:,0]
iris_df["ftr2"] = pca_transformed[:,1]
visualize_cluster_plot(dbscan, iris_df, "dbscan_cluster", iscenter=False)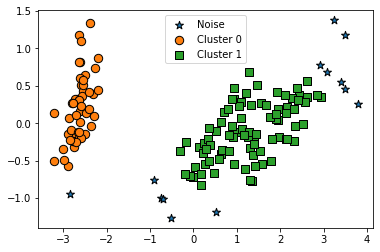
오.. 별로 표현된 값은 모두 노이즈다. PCA로 2차원 표현하면 이상치인 노이즈 데이터가 명확히 드러한다. DBSCAN을 적용할 때는 특정 군집 개수로 군집을 강제하지 않는것이 좋다.
일반적으로 eps의 값을 크게 하면 반경이 커져 포함하는 데이터가 많아지므로 노이즈 갯수가 감소하고, min_samples를 크게 하면 주어진 반경 내에서 더 많은 데이터를 포함해야므로 노이즈 갯수가 증가한다.
# dbscan
dbscan = DBSCAN(eps = 1, min_samples = 8, metric = "euclidean")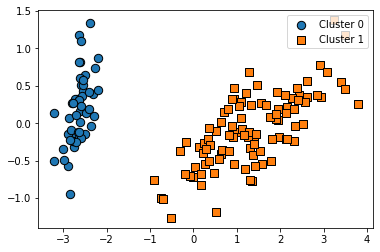
dbscan = DBSCAN(eps = 2, min_samples = 8, metric = "euclidean")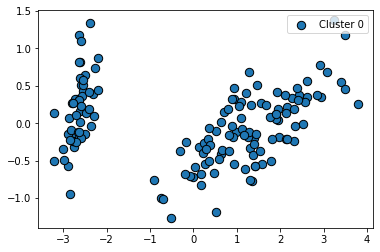
eps의 값을 변화시켰더니 위와 같은 그림이 나왔다. 확실히 값을 증가시켰더니 -1(노이즈 데이터) 수가 줄어든 것을 확인 할 수 있었다.
dbscan = DBSCAN(eps = .5, min_samples = 8, metric = "euclidean")
dbscan = DBSCAN(eps = .1, min_samples = 8, metric = "euclidean")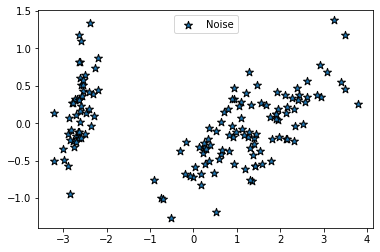
eps를 줄이자 노이즈 데이터가 기존보다 많이 증가하는 모습을 확인 할 수 있었다. 이런 변화는 min_samples 의 값을 크게했을때도 볼 수 있다.