랜덤 포레스트란?
랜덤 포레스트(Random Forest)는 배깅을 적용한 의사결정나무의 앙상블 알고리즘이다. 랜덤 포레스트는 나무가 아니라 모인 숲의 수준으로 하나의 트리 모델이 아닌 다수의 부트스트랩 표본으로 트리 모델을 만든 후 그 결과를 취합해 분류의 경우에는 다수결로, 회귀의 경우에는 평균을 출력합니다.
이는 배깅과 동일한 방법이나 트리 모델의 분할 기준(노드)을 정하는 방법에서 차이가 있다. 배깅은 노드마다 모든 독립변수 내에서 최적의 분할을 찾는 방법을 사용하지만, 랜덤 포레스트는 독립변수들을 무작위로 추출하고, 추출된 독립변수 내에서 최적의 분할을 만들어 나가는 방법을 사용한다.
하나의 트리 모델에서 발생할 수 있는 과적합 문제가 랜덤 포레스트에서는 줄어들고, 예측 성능 또한 높게 나타난다.
패키지 설치 및 불러오기
> install.packages('randomFoerst)
> library(randomForest)Data Load
> train <- read.csv('ch6-2_train.csv', header=TRUE, stringsAsFactors=TRUE)
> test <- read.csv('ch6-2_test.csv', header=TRUE, stringsAsFactors=TRUE)
> str(train)
'data.frame': 240 obs. of 4 variables:
$ wing_length: int 238 236 256 240 246 233 235 241 232 234 ...
$ tail_length: int 63 67 65 63 65 65 66 66 64 64 ...
$ comb_height: int 34 30 34 35 30 30 30 35 31 30 ...
$ breeds : Factor w/ 3 levels "a","b","c": 1 1 1 1 1 1 1 1 1 1 ...랜덤 포레스트
> rf <- randomForest(breeds ~ ., data=train, type='class')
> names(rf)
[1] "call" "type" "predicted"
[4] "err.rate" "confusion" "votes"
[7] "oob.times" "classes" "importance"
[10] "importanceSD" "localImportance" "proximity"
[13] "ntree" "mtry" "forest"
[16] "y" "test" "inbag"
[19] "terms"변수 중요도
> rf$importance
MeanDecreaseGini
wing_length 52.77529
tail_length 68.02902
comb_height 30.44215varImpPlot() 함수를 이용하면 importance객체를 그래프로 그릴 수 있다.
> varImpPlot(rf)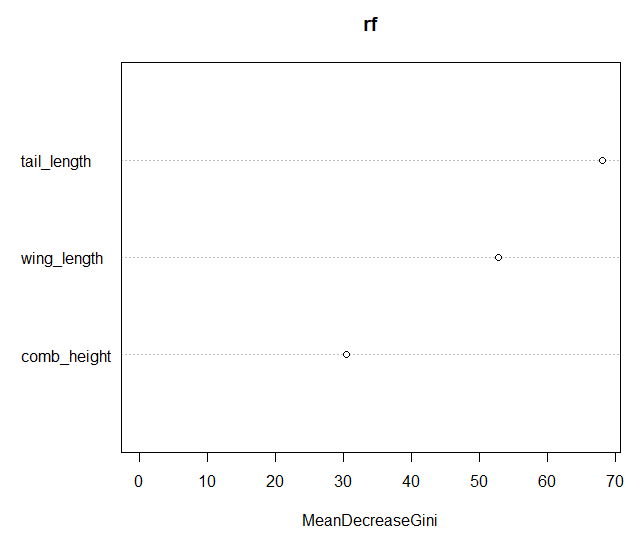
모델 성능 평가
> test$pred <- predict(rf, newdata=test, type='class')
> library(caret)
> confusionMatrix(test$pred, test$breeds)
Confusion Matrix and Statistics
Reference
Prediction a b c
a 20 1 0
b 0 17 1
c 0 2 19
Overall Statistics
Accuracy : 0.9333
95% CI : (0.838, 0.9815)
No Information Rate : 0.3333
P-Value [Acc > NIR] : < 2.2e-16
Kappa : 0.9
Mcnemar's Test P-Value : NA
Statistics by Class:
Class: a Class: b Class: c
Sensitivity 1.0000 0.8500 0.9500
Specificity 0.9750 0.9750 0.9500
Pos Pred Value 0.9524 0.9444 0.9048
Neg Pred Value 1.0000 0.9286 0.9744
Prevalence 0.3333 0.3333 0.3333
Detection Rate 0.3333 0.2833 0.3167
Detection Prevalence 0.3500 0.3000 0.3500
Balanced Accuracy 0.9875 0.9125 0.9500