오늘 review해 볼 논문은 2015년도에 나온 U-Net입니다!
제목 : U-Net : Convolutional Networks for Biomedical Image Segmentation
저자 : Olaf Renneberger et al.
학회 : Medical Image Computing and Computer-Assisted Intervention(MICCAI 2015)
게재 년도 : 2015년
인용수 : 94,526회
0.필수 개념
먼저 필수 개념에 대해서 알아보고 시작하겠습니다~
논문 제목에도 나와있듯이 U-Net은 Biomedical Image Segmentation을 위한 네트워크인데요
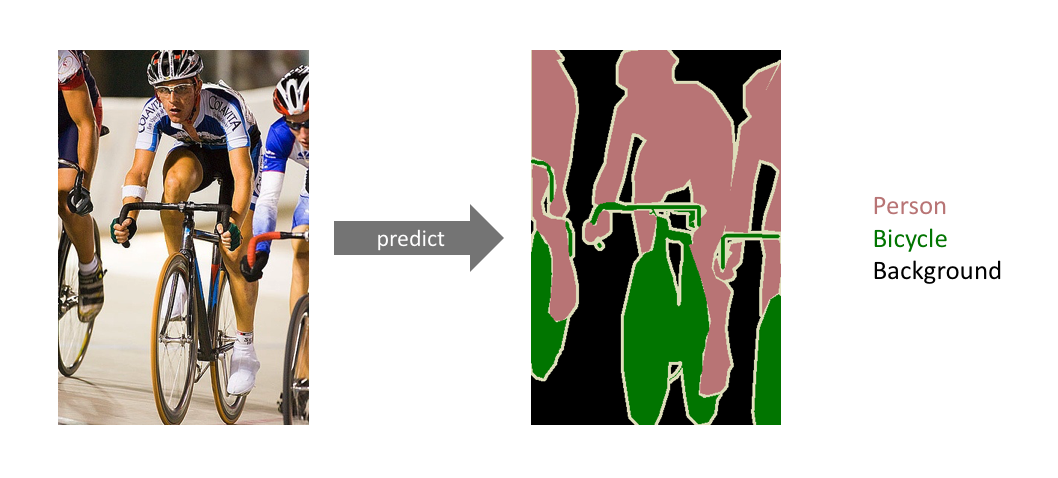
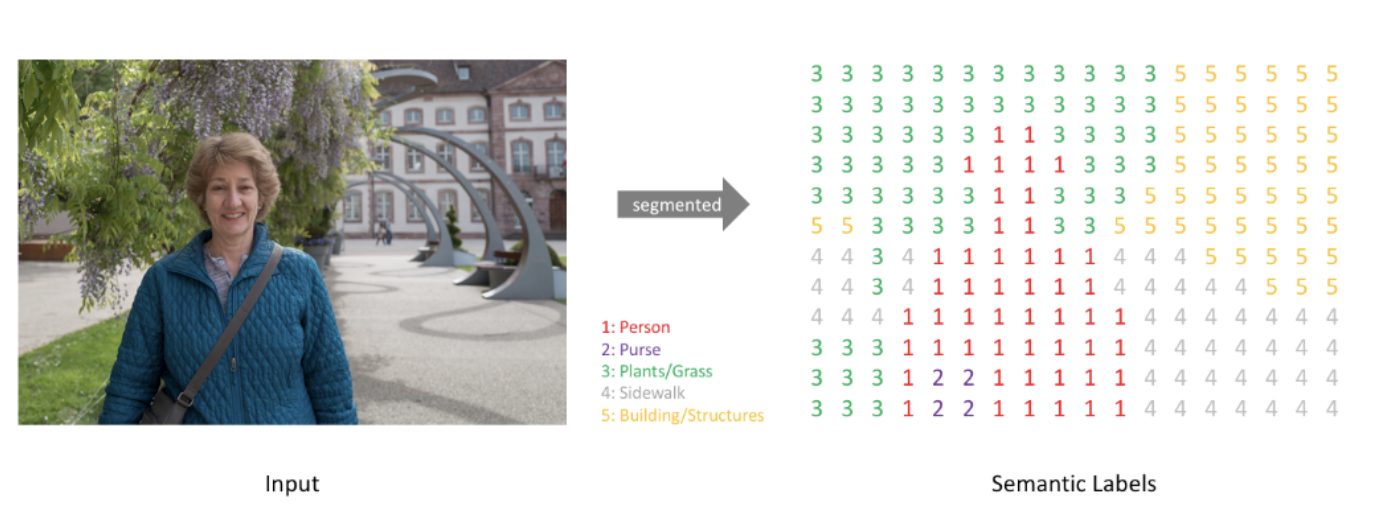
여기서 Image Segmentation이 뭔지 알아보고 가겠습니다
Image Segmentation이란?
정의 : 이미지 내에 있는 각 물체(object)들을 의미 있는(semantic) 단위로 분할(segmentation)하는 작업을 의미합니다
->각 pixel마다 클래스(class)를 할당하는 작업입니다
- 일반적인 분류(classification)에서는 단일 이미지를 하나의 클래스로 분류합니다
- 분할(segmentation)에서는 각 픽셀마다 하나의 클래스로 분류합니다
Semantic segmentation을 진행하면, 이미지가 주어졌을 때 (높이 X 너비 X 1) 크기를 가지는 한 장의 분할 맵(segmentation map)이 생성됩니다
다음으로는 convolutional network에 대해서도 알아보겠습니다~
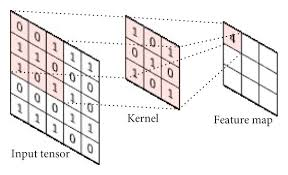
CNN
CNN에서는 filter 또는 kernel이라고 부르는 것을 사용합니다
- 각 filter는 입력에서 특정한 특징(feature)를 잡아내어 특징맵(feature map)을 생성합니다
- 하나의 filter는 sliding 하면서 convolution 연산을 통해 feature map을 계산합니다
- 일반적으로 CNN 분류 모델에서 깊은 layer로 갈수록 채널의 수가 증가하고, 너비와 높이는 감소합니다
- convolution layer의 서로 다른 fitler들은 각각 적절한 특징(feture) 값을 추출하도록 학습됩니다
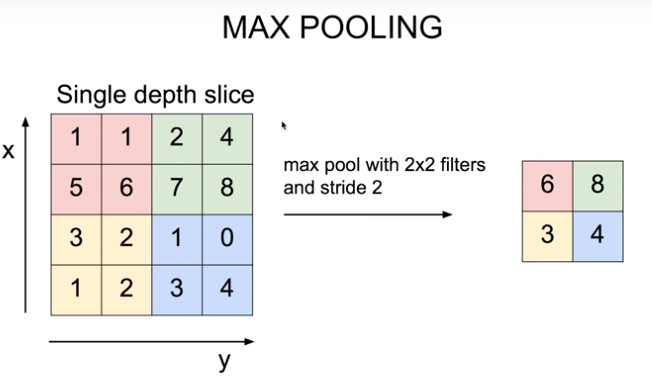
Max Pooling
CNN 분류 모델에서 중요한 정보는 유지한 상태로 해상도(너비와 높이)를 감소시키는 방법 중 하나입니다
- 가장 큰값을 가지는 원소(element)만 남깁니다
Down-Sampling & Up-Sampling
Down-Sampling : 너비와 높이 감소를 도와줍니다(해상도 감소)
Up-Sampling : 너비와 높이 증가를 도와줍니다(해상도 증가)
Fully Conovlutional Network(FCN)
Semantic Segmentation 모델을 위해 기존에 이미지 분류에서 우수한 성능을 보인 CNN 기반 모델(AlexNet, VGG16, GoogLeNet)을 목적에 맞춰 변형시킨 것입니다
- FCN은 단순히 Convolutional layer로만 이루어져 있는 네트워크입니다
지금까지 간단한 필수 개념에 대해서 알아보았습니다
이제 본격적으로 논문 review를 시작해보겠습니다!
1. U-Net
U-Net은 Biomedical 분야에서 이미지 분할(Image Segmentation)을 목적으로 제안된 End-to-End 방식의 Fully-Convolutional Network 기반 모델입니다
네트워크의 모양이 서로 대칭되는 U자 모양으로 보여 U-Net이라는 이름이 붙여지게 되었습니다
U-Net은 context를 캡처하는 축소 경로(contracting path)와 정확한 localization을 가능하게 하는 대칭 확장경로(symmetric expanding path)로 구성됩니다
2. Network Architecture
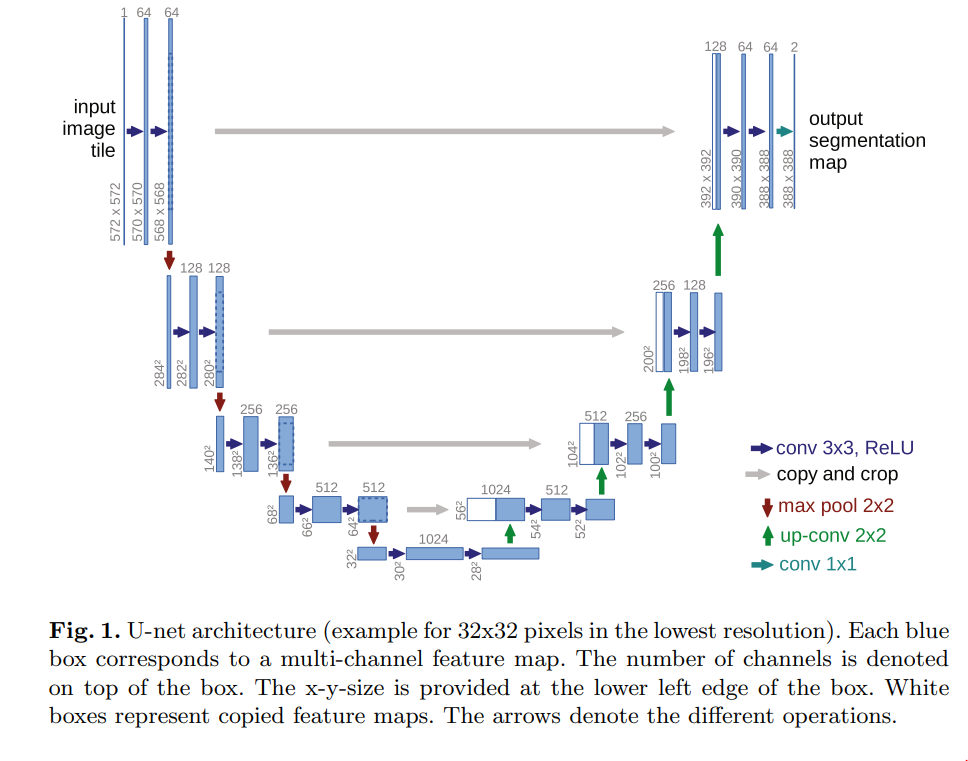
U-Net의 전체 Architecture는 위와 같습니다
전체적인 네트워크는 Fully Connected layer가 없는 Fully Convolutional Network(FCN) Architecture로 구성됩니다
이번에는 Contracting Path와 Expanding Path에 대해서 알아보겠습니다!
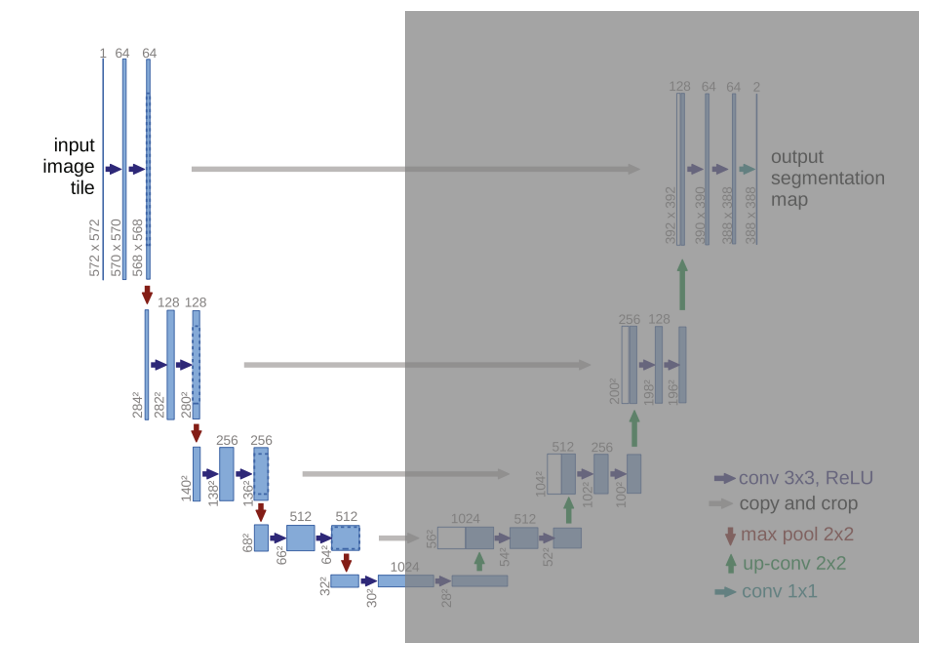
1. Contracting Path
- U-Net Architecture의 초반 부분입니다
- 2 X 2 Max Pooling을 사용해 해상도를 줄입니다
-> 해상도(너비와 높이)가 2배 감소하게 됩니다 - Conv 연산으로 채널 크기는 2배 증가하게 됩니다
- 일반적인 CNN 모델처럼 다음 형식을 반복합니다
-> Conv 연산들 -> ReLU -> Max Pooling
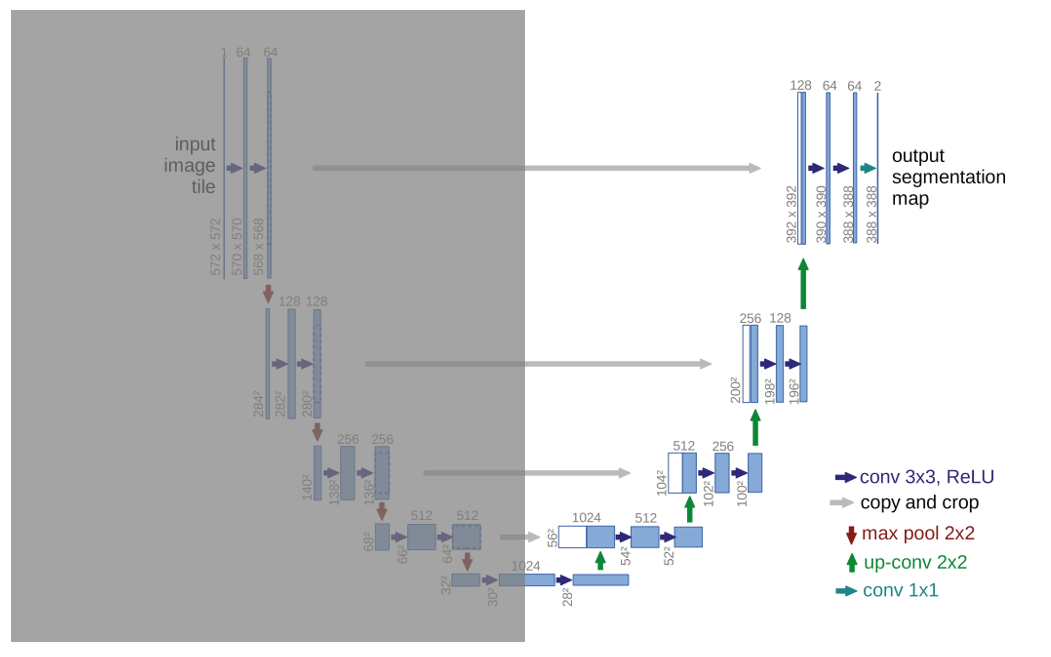
2. Expanding Path
- U-Net Architecture의 후반 부분입니다
- 2 X 2 Convolution(Up-Convolution)을 사용해 해상도를 늘립니다
-> 해상도(너비와 높이)가 2배 증가하게 됩니다 - Conv 연산으로 채널 크기는 2배 감소하게 됩니다
- 수축 경로(Contracting Path)에서 처리된 feature map을 그대로 확장(expanding path)에 가져와서 채널 level에 붙여서 사용합니다
-> 앞 부분의 layer에서 추출되었던 feature에 대한 정보들을 뒷부분의 layer에서 그대로 사용할 수 있어, 보다 학습 난이도가 낮아지고 좋은 결과를 얻을 수 있습니다 - 마지막 layer에서 1 X 1 convolution 연산을 수행합니다
Overlap-tile 전략
U-Net에서는 크기가 큰 이미지를 사용하는 경우, 이미지 전체를 사용하는 대신 overlap-tile 전략을 사용합니다
FCN의 특성상 입력 이미지의 해상도에는 제한이 없습니다.
하지만 U-Net 구조상 출력 이미지의 해상도가 입력 이미지보다 작습니다
따라서 tile A와 tile B의 일부 영역이 겹치는 Overlap-tile 방식을 적용합니다
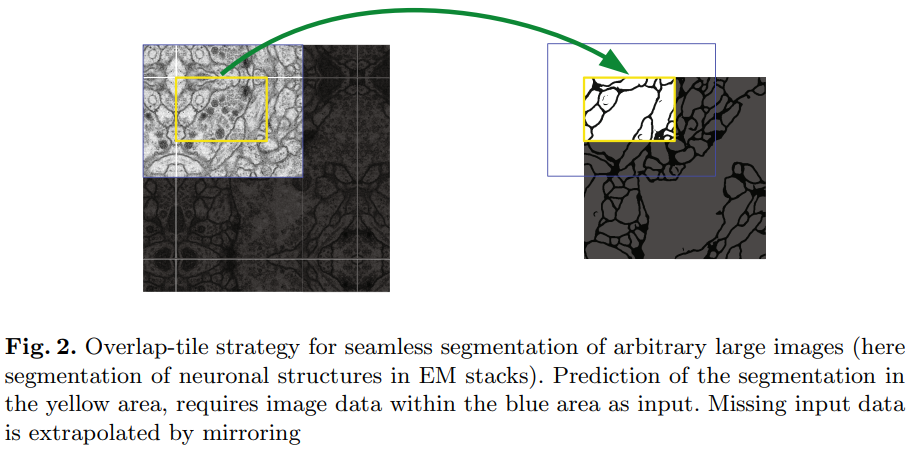
위 그림에서 알 수 있듯이, 실제로 노란색 영역의 segmentation이 필요하면 더 큰 범위(파란색 영역)의 patch를 삽입합니다
이때 이미지의 경계 부분은 extrapolation을 사용하는데, 본 논문에서는 mirroring 방법이 사용되었습니다
mirroring 방법을 사용하면 원본 이미지의 경계 부분이 거울에 반사된 것처럼 확장되게 됩니다.
예를 들어서) 노란 부분에 대해서 왼쪽 위 부분을 segmentation 해야한다면, 왼쪽 위는 이미지가 존재하지 않는 부분이기 때문에 간단하게 이미지가 존재하는 부분을 사용해서 mirroring을 통해 이미지 path를 만들어 준 뒤에 이것들을 다 포함해서 네트워크의 입력으로 넣게 됩니다
3. Training
Object Function
U-Net은 segmentation을 위한 네트워크로, 픽셀 단위(pixel-wise)로 소프트맥스(softmax)를 사용합니다
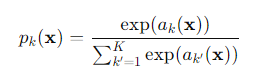

본 논문에서 사용되는 softmax 함수는 일반적으로 사용하는 softmax와 동일합니다
각각의 pixel마다 확률값을 예측할 수 있도록 하기 위해서, 모두 pixel마다 확률값을 구하는 형태로 softmax를 사용하게 됩니다
학습을 위해 cross-entropy를 사용합니다
true label만 고려하므로, 일반 cross entropy 공식과 동일합니다
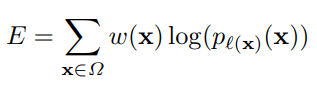
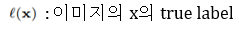
본 수식에서는 log를 사용하여 확률값이 증가할 수 있도록 했습니다
또한 일반적인 cross-entropy 함수에 w(x)라는 추가적인 가중치 함수를 사용하였습니다
이를 통해 각각의 pixel마다 가중치를 부여해서, 더 학습이 잘 수행될거나 덜 수행될 수 있도록 만들 수 있습니다
Touching Cells Separation
U-Net에서는 세포(cell)을 명확히 구분하기 위해 작은 분리 경계(small separation border)를 학습합니다
이때 세포를 명확히 구분하기 위해서는 가중치 함수를 사용합니다
w(x)는 인접한 셀(touching cell) 사이에 있는 배경 label에 대해 높은 가중치를 부여합니다

이때, exp는 지수 함수를 의미합니다
'-'가 붙은 이유는, 거리 값이 작을수록 가중치가 커지게 하기 위해서 입니다
즉, 세포와의 거리가 가까우면 가중치(weight)가 증가하도록 하는식이라고 볼 수 있습니다
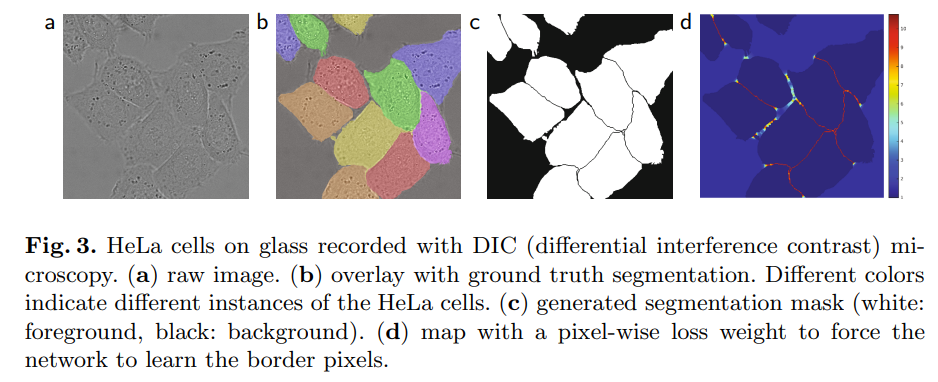
위 그림을 보면.. 특히 c번을 보면 서로 붙어있는 여러개의 세포에 대해서 보다 명확하게 배경으로 분리된 것을 알 수 있습니다
Data Augmentation
의료 데이터에서는 학습 데이터의 수가 적은 경우가 많으므로, 데이터 augmentation이 필요합니다
- 본 논문에서는 일반적인 data augmentation 기술을 사용합니다
- 추가적으로 Elastic Deformation 방법을 사용합니다
-> Elastic Deformation : 각각 grid에 대해서, 보다 비선형적으로 변형을 가해서 만들어진 데이터 또한 학습 데이터로 사용하는 방식

4. Experiments
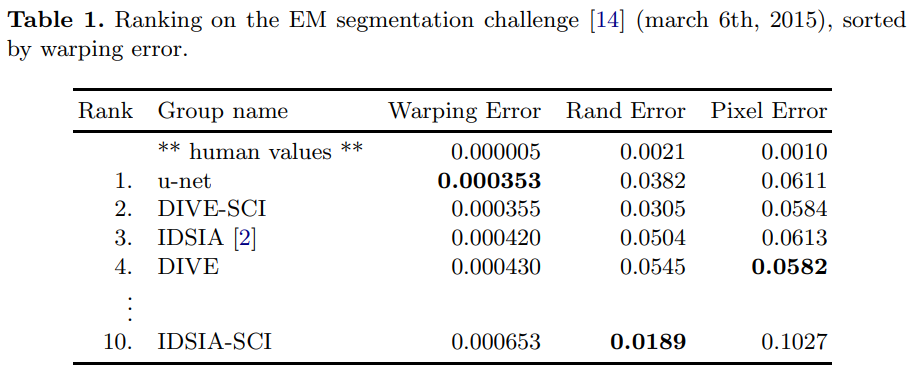
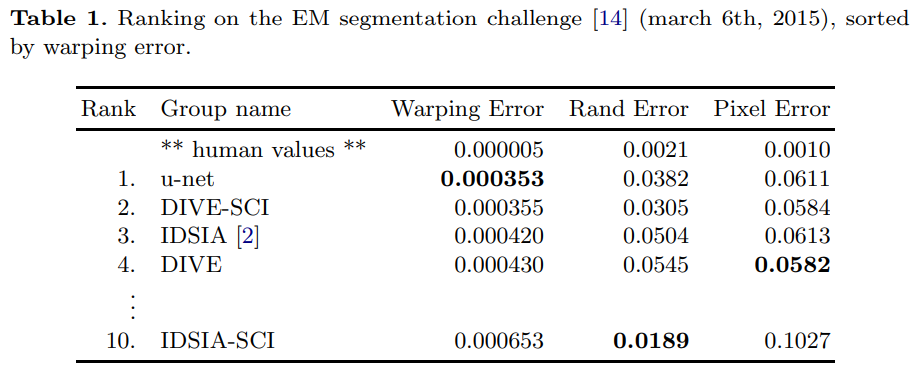
EM(Electron Microscopy) segmentation 대회의 데이터세트로 평가를 진행한 결과입니다
Warping error를 기준으로 정렬한 결과, 사람을 제외하고 U-Net이 가장 우수한 정확도를 보인 것을 알수 있습니다
- Warping error: 시계열 데이터 또는 비선형 매칭에서 두 패턴을 정렬할 때 생기는 왜곡으로 인한 오차.
- Rand error: 군집화 또는 이미지 분할에서, 실제 군집과 예측된 군집 간의 불일치를 측정하는 오차.
- Pixel error: 이미지 처리에서 개별 픽셀 단위로 실제 값과 예측 값 간의 차이를 계산한 오차.
또한 U-Net에서는 추가적인 두 개의 데이터세트(ISBI cell tracking challenge 2014 and 2015)에 대해서 평가를 진행했습니다
- PHC-U373 : 35개의 부분적으로 주석이 있는 학습 이미지 데이터 세트
- DIC-HeLa : 20개의 부분적으로 주석이 있는 학습이미지 데이터 세트
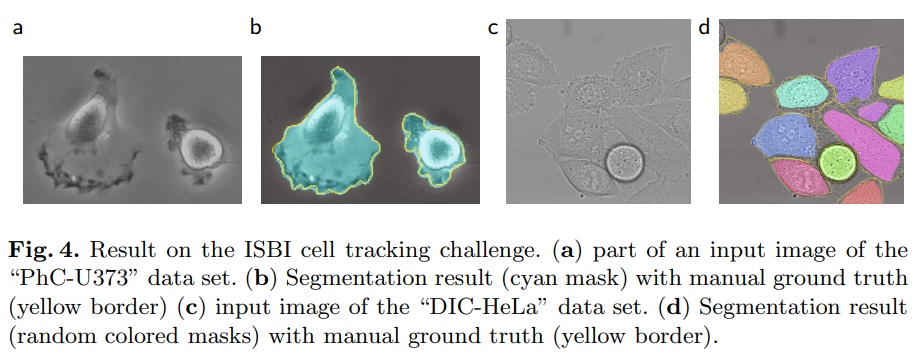
위 결과를 보면, 노란색 테두리(ground truth)와 상당히 유사한 segmenation 결과를 보인 것을 알 수 있습니다
b 그림을 봤을때 초록색은 실제 U-Net이 예측한 내용이며, 노란색 경계선은 정답 label로 정답 label과 거의 유사한 형태로 예측한 것을 알 수 있습니다
추가적인 두 개의 데이터세트(ISBI cell tracking challenge 2014 and 2015)에 대해서 평가를 진행합니다
PHC-U373과 DIC-HeLa 두 데이터 세트에 대해 이전 연구보다 우수한 IOU(Intersection Over Union)을 보였습니다
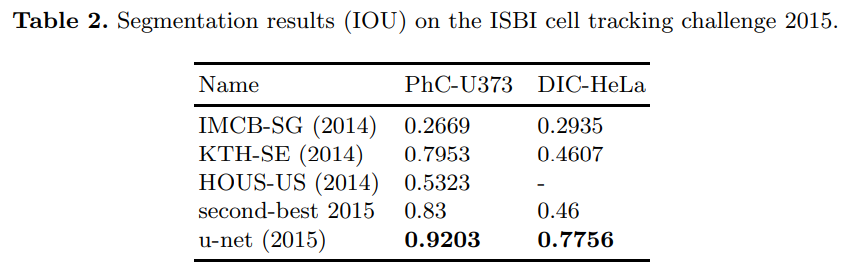
위 결과를 보면 U-Net은 0.9203으로 굉장히 좋은 분류 결과를 보였습니다
또한 이전 연구 결과와 비교해 봤을 때, 두드러진 IOU 점수 향상을 볼 수 있습니다
- IOU : 예측된 영역과 실제 영역의 겹친 부분을 전체 영역으로 나눈 비율로, 물체 검출에서 정확도를 평가하는 지표

5. 마무리
지금까지 U-Net에 대해서 살펴보았습니다
U-Net Architecture는 매우 다양한 생체의료 segmentation application에서 매우 우수한 성능을 발휘합니다
elastic deformation으로 데이터를 augmentation하여 주석 달린 이미지가 거의 필요하지 않으며, 매우 합리적인 교육 시간을 가진다는 특징을 알 수 있었습니다
이러한 U-Net Architecture는 다양한 많은 작업에 적용될 수 있으며, 현재도 많이 사용되고 있습니다