2015년 발표된 'U-Net: Convolutional Networks for Biomedical Image Segmentation' 논문을 읽고 정리한 글입니다. 원문 보러가기
1. Introduction
본 논문은 'fully convolutional network' (이하 FCN) 라고 불리는 모델을
변형하고 확장했습니다.
U-Net 구조에서 하나의 중요한 변경점은 upsampling 부분에서
feature의 채널 수가 많다는 점입니다.
이는 네트워크가 맥락(context)정보를 더 높은 resolution layer 들에게
전달할 수 있게 합니다.
또한 FCN을 변형한 모델답게 fc 층을 가지지 않습니다.
이런 전략은 overlap-tile 전략을 사용하여 임의의 큰 이미지에 대해
부드러운 segmentation을 가능하게 합니다.
이미지의 테두리 영역에 있는 픽셀들을 예측하기 위해
손실(missing)된 내용(context)을 입력 이미지를 미러링하여 예측합니다.
이러한 방법은 resolution이 GPU 메모리에 의해 제한되기 때문에
네트워크를 큰 이미지에 적용하는데 중요합니다.
2. Network Architecture
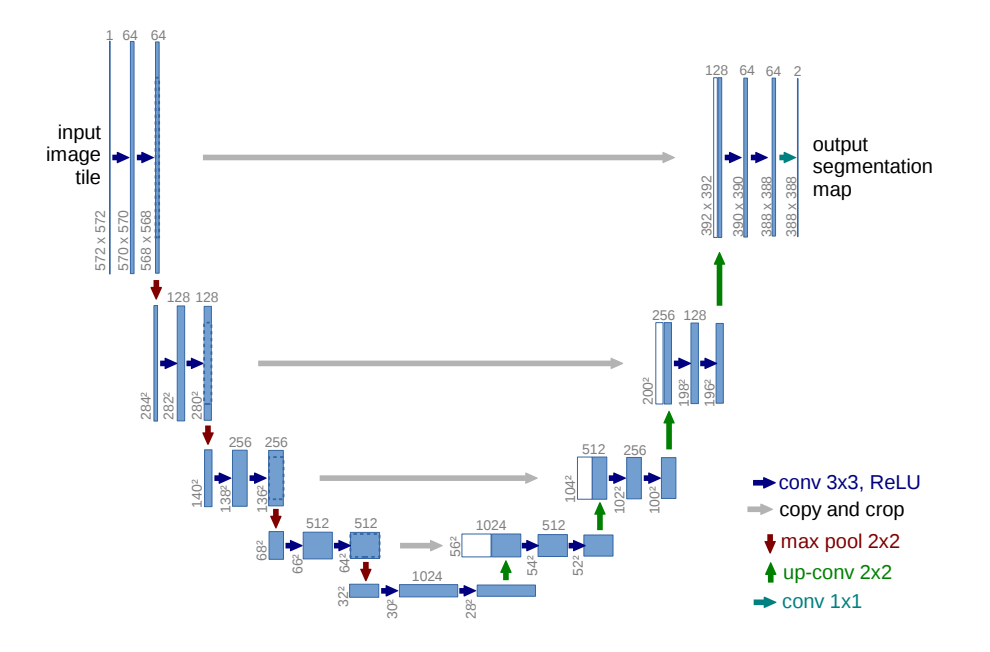 [U-Net의 구조]
[U-Net의 구조]
U-Net은 압축(contracting) 부분과 확장(expansive) 부분으로 이루어져 있습니다.
각각 맥락(context)를 포착하고, 정확한 localization을 가능하게 합니다.
압축(contracting) 부분은 일반적인 합성곱 네트워크의 구조를 따르고 있습니다.
ReLU 활성함수를 적용하여 3x3 convolution을 두번 적용합니다.
이후 2x2 max pooling(stride=2)을 적용하여 다운샘플링을 진행합니다.
이때 다운샘플링이 진행될때 마다 채널이 두배 증가합니다.
이와 반대로 확장(expansive) 부분의 업샘플링은 채널을 두배 감소시키며
2x2 up-convolution을 적용시킵니다.
그리고 스킵 연결을 진행하면서 ReLU 활성함수를 적용한
3x3 convolution을 두번 적용합니다.
논문에서는 스킵 연결이 합성곱을 진행할때 마다 테두리 픽셀에 대한 정보를 잃어버리기 때문에
필수적이라 합니다.
마지막 층에서는 1x1 convolution을 가집니다.
이때 모델의 출력인 segmentation map의 크기는 합성곱 진행시
패딩을 적용시키지 않았기 때문에 원본 입력보다 작습니다.
3. Training
모델이 사용하는 손실함수는 최종 특성맵에 대한 pixel-wise soft-max와
cross entropy를 조합하였습니다.
이때 soft-max는 다음과 같이 정의될 수 있습니다.
는 픽셀 에 대한 최종 특징맵 의 activation, K는 클래스의 개수,
는 maximum-function 입니다.
cross entropy는 다음과 같이 정의됩니다.
은 각 픽셀에 대한 true label
는 weight map 입니다.
3.1 Weight Map
논문의 저자는 서로 맞닿아있는 세포들의 미세한 간격을
모델이 학습하도록 강제하기 위해 weight map을 사용하였습니다.
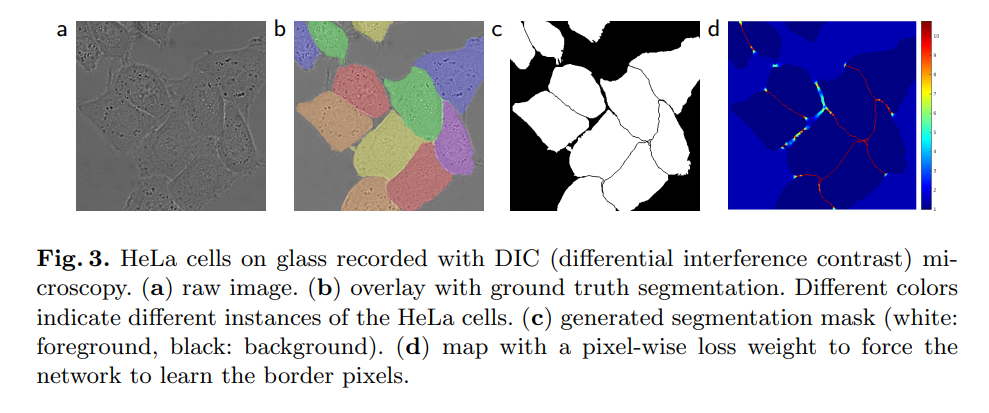 [weight map]
[weight map]
weight map은 다음과 같이 정의됩니다.
은 가장 가까운 세포와의 거리 이고
는 두번째로 가까운 세포와의 거리 입니다.
또한 논문의 저자들은 그리고 픽셀로 정의했습니다.
4. Conclusion
U-Net 모델은 적은 양의 훈련 데이터셋에도 불구하고 elastic deformation을 통하여
높은 성능을 달성하면서도 빠른 속도를 보였습니다.
