[Segmentation] CellViT: Vision Transformers for Precise Cell Segmentation and Classification 리뷰
Transformer
논문 제목
CellViT: Vision Transformers for Precise Cell Segmentation and Classification
URL: https://arxiv.org/abs/2306.15350
인용수 : 185회 (25.8.18 기준)
Abtract
H&E(헤마톡실린&에오신)으로 염색된 조직 이미지에서 세포핵을 검출하고 분할하는 것은 중요한 임상 과제이고, 광범위한 응용 분야에 필수적인 태스크임. 하지만 염색 및 크기의 다양성, 경계 겹침, 핵 군집화로 인해 어려운 태스크이다. 이를 위해 CNN이 광범위하게 사용되어 왔지만 본 논문에서는 트랜스포머 기반 네트워크의 잠재력을 탐구했다고함.
본 논문에서 CellViT라고 불리는 Vision Transformer 기반의 딥러닝 아키텍처를 사용하여 디지털화된 조직 샘플에서 세포핵을 자동으로 instance segmentation 하는 새로운 방법을 소개하였음. CellViT는 가장 어려운 세포핵 개체 분할 데이터셋 중 하나인 PanNuke 데이터셋으로 훈련 및 평가되었다. 이 데이터셋은 19개 조직 유형에 걸쳐 5개의 임상적으로 중요한 클래스로 분류된 약 200,000개의 주석 달린 세포핵으로 구성되어 있다.
최근 발표된 Segment Anything Model과 1억 4백만 개의 조직학적 이미지 패치로 사전 훈련된 ViT 인코더를 활용하여, 대규모의 in-domain 및 out-of-domain에서 사전 훈련된 비전 트랜스포머의 우수성을 입증함. 이를 통해 PanNuke 데이터셋에서 평균 Panoptic Quality 0.50과 F1-detection 0.83이라는 SOTA 성능을 달성했음.
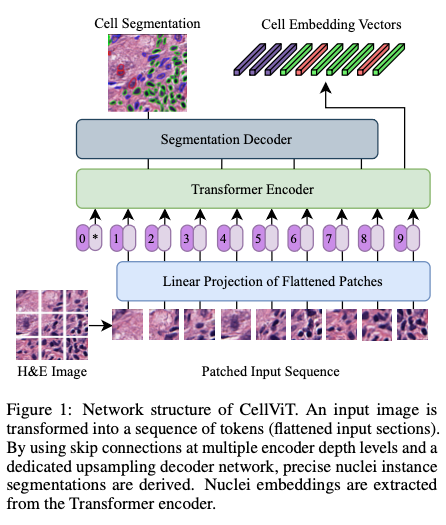
Introduction
- 조직 내 세포와 그 분포를 분석하는 진단을 위한 표준 절차이다. 하지만 세포 수준에서의 대규모 분석은 시간이 많이 소요되며 관찰자 내 및 관찰자 간 편차가 크다는 문제점이 있음.
- 병리학용 고속 스캐너의 발달로 디지털화된 조직 샘플(WSI)을 생성하는 것이 가능해졌으며 컴퓨터 비전(CV) 알고리즘의 적용을 가능하게 했음.
- WSI를 분석하는 기존 알고리즘들은 종종 이미지 영역의 특징 추출기로 사용되는 CNN에 기반을 둠. 이 알고리즘들은 임상 등급의 성능을 달성함에도 불구하고 해석 가능성에 한계가 있어 새로운 인간-해석 가능 바이오마커를 정의하는 데 어려움을 줌.
- 본 논문에서는 디지털화된 조직 샘플에서 세포핵의 자동 개체 분할을 위해 비전 트랜스포머에 기반한 CellViT를 제안. 제안된 네트워크 아키텍처는 핵 분할의 선두 모델 중 하나인 HoVer-Net과 유사한 U-Net 형태의 인코더-디코더 구조를 기반으로 함. ViT 구조를 통해 이미지 내 모든 세포 간의 관계를 이해할 수 있으며 long-range dependencies를 활용하여 분할 성능을 실질적으로 향상시킴.
Methods
CellViT 아키텍처는 3D volumetric images를 위한 UNETR 모델에서 영감을 받았지만 2D 이미지 처리를 위해 모델 구조를 변형했다고함. Segmentation map을 계산하기 위해 단일 디코더 브랜치를 사용하는 전통적인 네트워크와 다르게 제안 네트워크는 HoVer-Net의 접근법에서 착안하여 세 개의 개별적인 multi-task output branches를 사용함.
첫 번째 브랜치는 모든 핵의 이진 분할 맵(nuclei prediction, NP)을 예측하여 그 경계와 모양을 포착함. 두 번째 브랜치는 수평 및 수직 거리 맵(horizontal-vertical prediction, HV)을 생성하여 정확한 위치 파악과 경계 설정을 위한 중요한 공간 정보를 제공. 세 번째 브랜치는 핵 유형 맵(nuclei type map, NT)을 예측하여 다른 핵 유형의 분류를 가능하게 함.
- NP-branch : 이진으로 nuclei map을 예측함.
- HV-branch : 핵 픽셀들로부터 각 핵의 질량 중심까지의 수평 및 수직 거리를 -1과 1 사이로 정규화하여 예측.
- NT-branch : 핵 유형을 instance segmentation maps으로 예측.
세 출력들을 통합하기 위해 추가적인 후처리 단계를 거침. 서로 다른 브랜치로부터 얻은 정보를 병합하고, 겹치는 핵들을 분리하여 정확한 개별 분할을 진행하며 핵 유형 맵을 기반으로 핵의 클래스를 결정하는 과정을 포함.
Network Structure
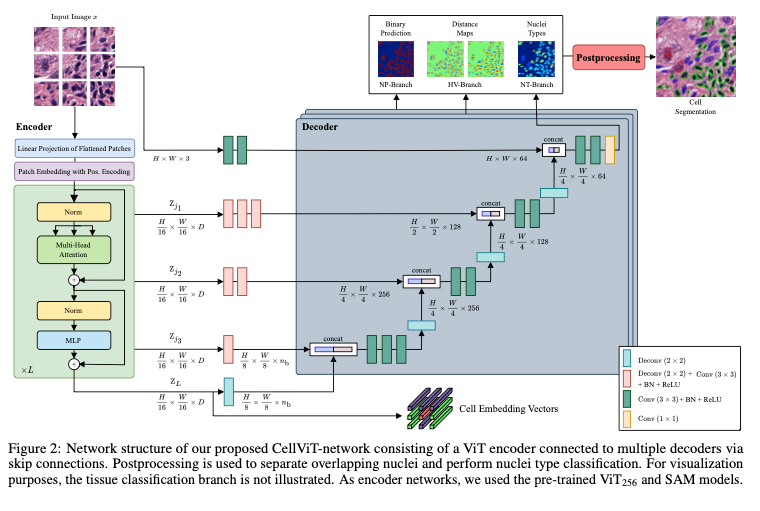
네트워크에서는 비전 트랜스포머를 이미지 인코더로 통합하고, skip connections를 통해 업샘플링 디코더 네트워크에 연결함. 이 아키텍처는 비전 트랜스포머의 강점을 이미지 인코더로서 개체 분할에 활용하면서도 세밀한 정보를 잃지 않도록 해줌. CellViT는 총 5개의 스킵 커넥션을 사용.
위 그림처럼 세 가지 브랜치(NP, HV, NT)는 동일한 스킵 커넥션과 동일한 이미지 인코더를 공유함. 유일한 차이점은 각 브랜치에 특화된 디코더의 분리된 업샘플링 경로임.
Target and Losses
더 빠른 훈련과 네트워크의 더 나은 수렴을 위해 각 네트워크 브랜치에 대해 서로 다른 손실 함수의 조합을 사용. 전체 손실 함수는 아래와 같음
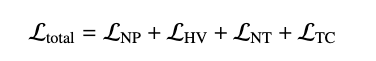
여기서 는 NP-브랜치의 손실, 는 HV-브랜치의 손실, 는 NT-브랜치의 손실, 그리고 는 TC-브랜치의 손실을 나타냄.
개별 브랜치 손실 함수는 다음과 같은 가중치가 적용된 손실 함수들로 구성.
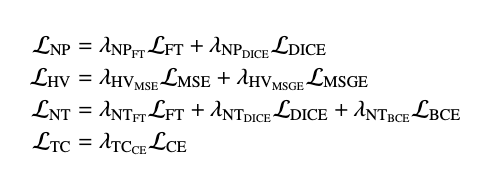
개별 segmentation loss는 아래 수식이고,
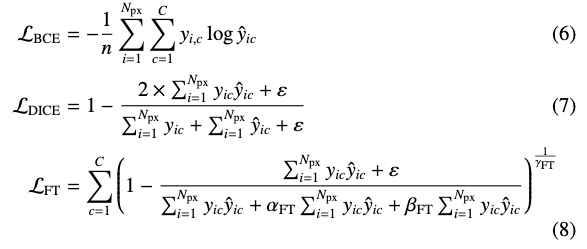
조직 분류 손실 함수로는 cross-entropy를 사용.
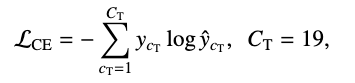
Postprocessing
네트워크가 분리된 핵들의 semantic instance segmentation을 직접적으로 제공하지 않기 때문에 정확한 결과를 얻기 위해서는 후처리가 필요하다고함.
서로 다른 브랜치로부터 얻은 정보를 병합하고, 겹치는 핵들을 분리하여 정확한 개별 분할을 보장하고, 핵 유형 맵을 기반으로 핵의 클래스를 결정하는 과정이 포함됨.
Nuclei Separation and Classification
서로 인접하거나 겹치는 핵들을 분리하기 위해 HoVer-Net의 검증된 후처리 파이프라인을 활용했다고함. 이는 핵 경계 사이의 전이와 핵과 배경 사이의 경계를 포착하기 위해 수평 및 수직 거리 맵의 gradients를 계산하는 것을 포함. 해당 지점에서는 그래디언트 값에 상당한 변화가 발생. 그 후 Sobel operator를 적용하여 거리 맵 내에서 이웃 픽셀 간에 상당한 차이가 있는 영역을 식별. 최종 경계를 생성하기 위해 marker-controlled watershed algorithm을 사용. 핵 클래스를 계산하기 위해 분리된 핵의 출력은 핵 유형 예측과 병합됨. 이를 위해 NT 예측 맵을 사용하여 핵 영역 내에서 majority voting을 통해 다수 클래스가 모든 핵 픽셀에 할당됨.
Experimental Setup
Datasets
PanNuke
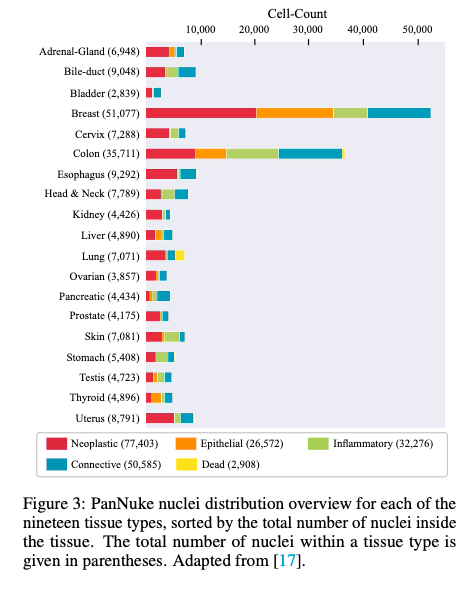
이 데이터셋은 19개의 다른 조직 유형과 5개의 고유한 세포 카테고리에 걸쳐 7,904개의 256×256 픽셀 이미지에 주석 처리된 189,744개의 핵을 포함하고 있음. 세포 이미지들은 x40 배율에서 0.25μm/px의 해상도로 캡처되었다.
MoNuSeg
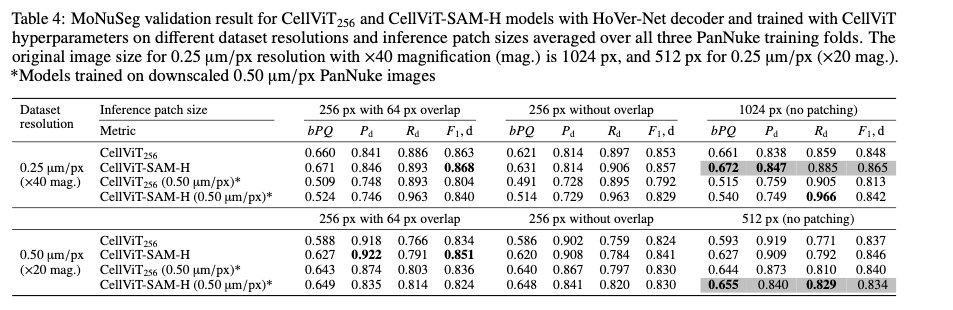
해당 데이터셋은 훨씬 작고 핵을 다른 클래스로 나누지 않음. 해당 연구에서는 모델을 평가하기 위해 MoNuSeg의 테스트 데이터셋만 사용함. 테스트 데이터셋은 x40 배율 0.25μm/px 해상도로 촬영된 14개의 1000×1000 픽셀 이미지로 구성됨. CellViT 입력을 위해 1024×1024 픽셀 크기로 조정하였음.
CoNSeP
0.25μm/px의 해상도와 1000×1000 픽셀의 이미지 크기를 가진 41개의 H&E 염색 결장직장 선암 WSI로 구성되어 있으며, 우리는 이를 MoNuSeg 데이터와 유사하게 1024×1024 픽셀로 재조정하였음.
Experiments
실험은 PanNuke 데이터셋을 3-Fold 교차 검증을 사용하여 실험을 수행하고 세 분할 모두에 대한 평균 결과를 나타내었음.
Evaluation Metrics
Nuclear Instance Segmentation Evaluation
일반적으로 semantic segmentation의 평가 지표로는 Dice coefficient나 Jaccard index가 사용됨. 하지만 두 지표는 핵의 검출 품질을 고려하지 않기 때문에 핵 개체 분할을 평가하기에는 불충분함. 따라서 다음과 같은 세 가지 요구사항을 평가할 수 있는 지표가 필요함.
- 배경으로부터 핵을 분리할 것.
- 개별 핵 인스턴스를 검출하고 겹치는 핵을 분리할 것
- 각 인스턴스를 분할할 것
이를 만족하는 지표인 PQ(Panoptic Quality)를 사용했다고함.
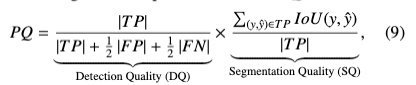
여기서 는 Intersection-over-Union을 나타냄. 위 식에서 y는 정답(GT) 세그먼트를, 는 예측된 세그먼트를 나타내며, 쌍 은 하나의 정답 세그먼트와 하나의 예측된 세그먼트로 이루어진 고유한 매칭 집합임.
각 클래스에 대해, 의 고유한 매칭은 예측된 세그먼트와 정답 세그먼트를 세 가지 집합으로 나눔.
- TP : 매칭된 세그먼트 쌍, 즉 정확하게 검출된 인스턴스 (정답, 예측 일치)
- FP : 매칭되지 않은 예측된 세그먼트, 대응하는 정답 인스턴스가 없는 예측된 인스턴스 (정답이 아닌걸 예측)
- FN : 매칭되지 않은 정답 세그먼트 대응하는 예측된 인스턴스가 없는 정답 인스턴스 (정답인데 예측된게 없음)
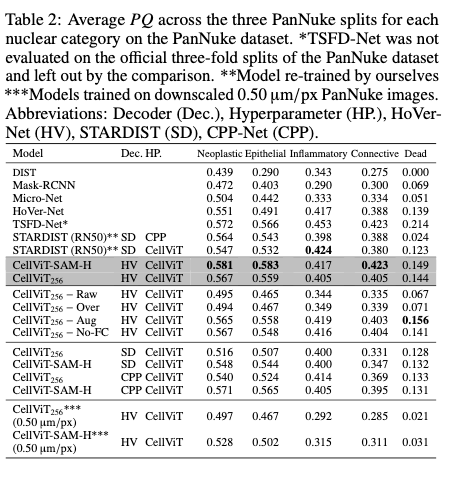
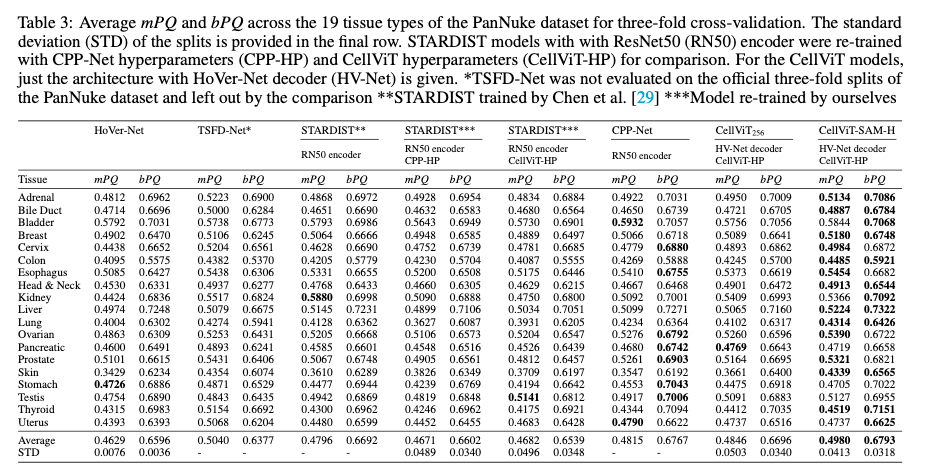
PQ 점수는 직관적으로 두 부분으로 분해될 수 있는데 분류 및 검출 시나리오에서 흔히 사용되는 F_1 Score와 유사한 검출 품질이며 매칭된 세그먼트들의 평균 IoU인 분할 품질임. 공정한 비교를 보장하기 위해 본 논문에서는 모든 핵이 하나의 클래스(핵 vs 배경)에 속한다고 가정하는 bPQ와 핵 클래스를 고려하는 더 어려운 mPQ를 모두 사용합니다. mPQ를 계산할 때는 각 핵 클래스에 대해 독립적으로 PQ를 계산한 후 모든 클래스에 대해 그 결과를 평균냄.
Nuclear Classification Evaluation
모델의 검출 품질을 평가하기 위해 일반적으로 사용되는 검출 지표를 사용. (F1 Score, Precision, Recall)
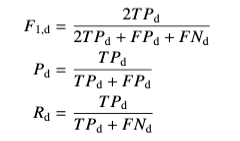
아랫첨자 d는 인스턴스(핵) 검출에 대한 지표이고,
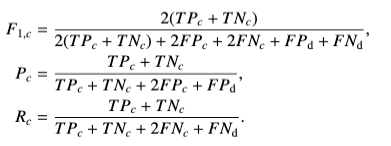
아랫첨자 c는 클래스 분류에 대한 지표.
Results
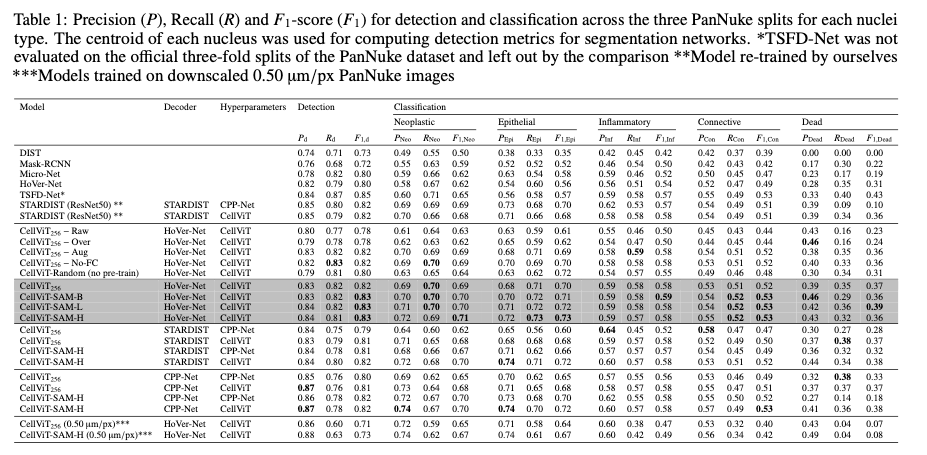
Detection Quality on PanNuke
Segmentation Quality on PanNuke
MoNuSeg Test Performance
Discussion and Conclusion
본 연구는 사전 훈련된 ViT_256 및 SAM을 활용하여 PanNuke 데이터셋에서 기존 모델을 크게 능가하는 SOTA 수준의 세포핵 검출 및 분할 성능을 달성한 새로운 딥러닝 모델 CellViT를 제안. CellViT는 큰 추론 패치 크기를 사용해 기존 모델보다 1.85배 빠른 추론 속도를 보여주며 분할과 동시에 각 핵의 고유한 특징 벡터(토큰)를 추가 계산 없이 효율적으로 추출할 수 있음. 이렇게 추출된 특징 벡터는 향후 그래프 기반 네트워크와 같은 해석 가능한 후속 연구에 활용될 잠재력을 가지며 특정 세포 패턴과 직접 연관된 알고리즘 설계의 가능성을 보여줬음. 다만 이 모델은 주로 고해상도(0.25μm/px) 이미지에서 안정적인 성능을 보이며 저해상도에서는 성능 저하가 나타나고 추가 데이터셋을 통한 외부 검증이 필요하다는 한계가 있음.
