베이지안 최적화? -> 그리드 서치처럼 하이퍼 파라미터 최적화를 수행하는 방법 중 하나
그리드 서치가 있는데 굳이 이걸 왜 배울까?
그리드 서치의 문제점
하이퍼 파라미터 튜닝 수행 방법
1. 그리드 서치
2. 랜덤 서치 (그리드 서치를 랜덤하게 해주는 것)
3. 베이지안 최적화
4. 수동 튜닝
그리드 서치
- 하이퍼 파라미터 갯수가 많을 때 문제 발생
- Gradient Boosting 기반 알고리즘은 튜닝할 하이퍼 파라미터 개수가 많고 범위가 넓어서 경우의수가 너무 많음
- 경우의수가 많을 때 데이터가 크면 파라미터 튜닝에 굉장히 오랜 시간이 투입되어야 함
GridSearchCV: 수행 시간이 너무 오래 걸림, 데이터 세트 작을 때 유리
RandomizedSearch: 최적 하이퍼 파라미터 검출에 태생적 제약, 데이터 세트 클 때 유리
단, 파라미터 갯수가 많지 않을 때는 그리드 서치 적용이 오히려 좋을 수 있음
베이지안 최적화 개요
베이지안 최적화가 필요한 순간
- 가능한 최소의 시도로 최적의 답을 찾아야 할 경우 (ex: 금고 털기)
- 개별 시도가 너무 많은 시간/자원이 필요할 때
베이지안 최적화
- 미지의 함수가 반환하는 값의 최소 또는 최댓값을 만드는 최적해를 짧은 반복을 통해 찾아내는 최적화 방식
- 새로운 데이터를 입력 받았을 때 최적 함수를 예측하는 사후 모델을 개선해 나가면서 최적 함수를 도출
- 대체 모델(Surrogate Model)과 획득 함수로 구성
- 대체 모델은 획득 함수로부터 최적 입력 값을 추천 받은 뒤 이를 기반으로 최적 함수 모델을 개선
- 획득 함수는 개선된 대체 모델을 기반으로 다시 최적 입력 값을 계산
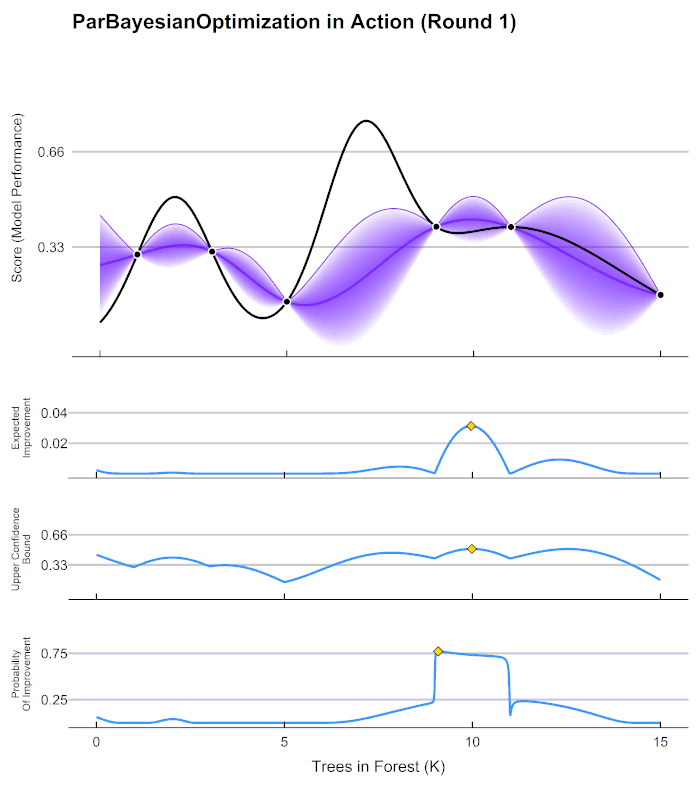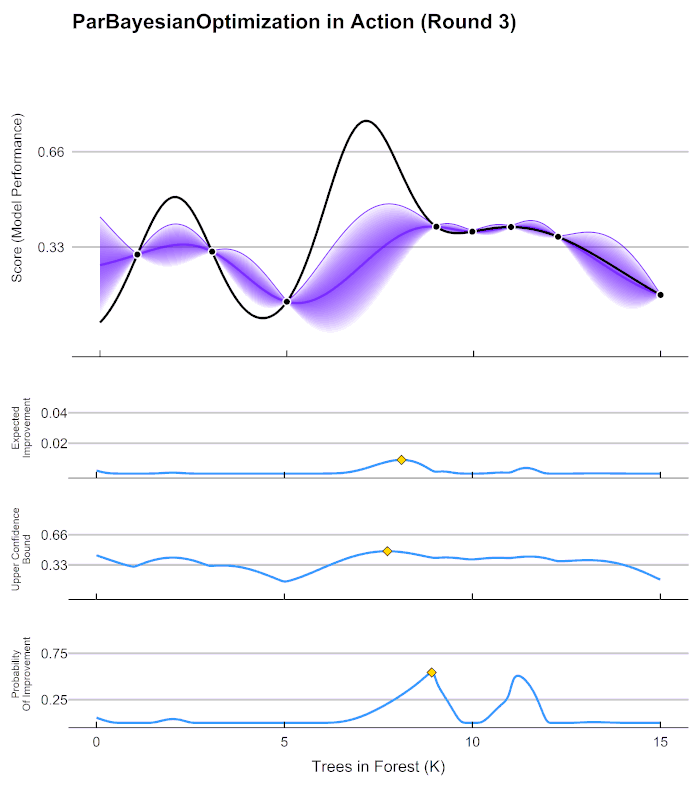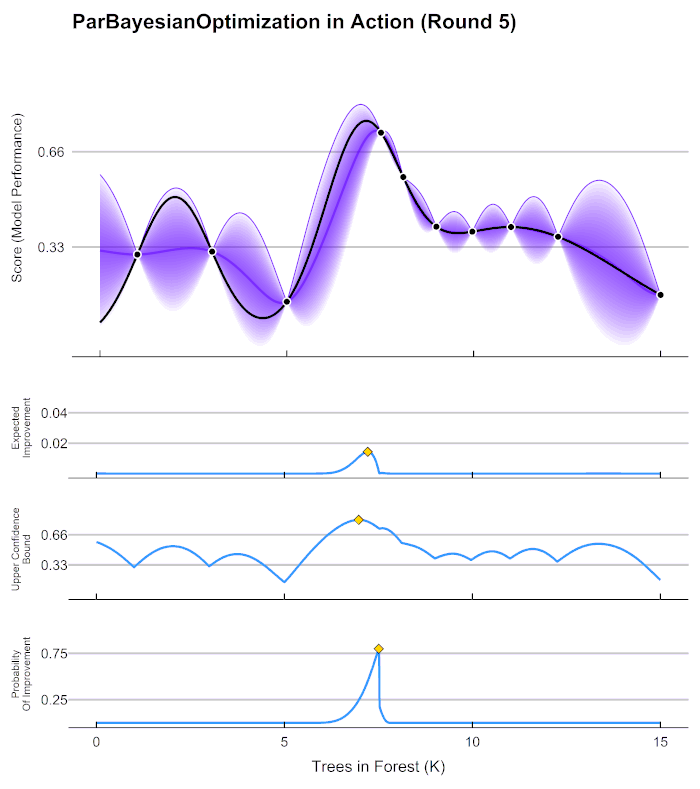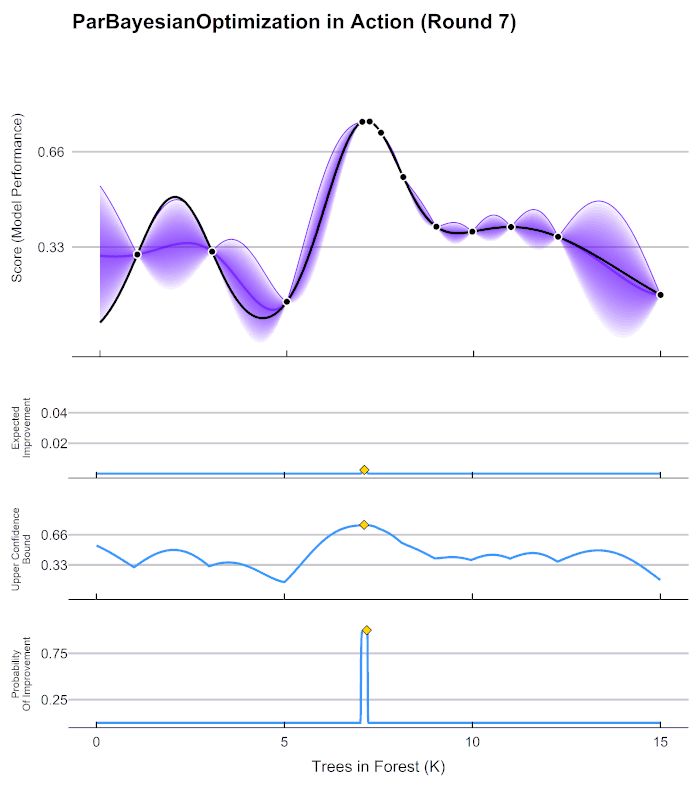
출처: https://commons.wikimedia.org/wiki/File:GpParBayesAnimationSmall.gif
대체 모델 = 사후 모델
획득 함수 = activation function
Step1: 최초에는 랜덤하게 하이퍼 파라미터들을 샘플링 하여 성능 결과를 관측
Step2: 관측된 값을 기반으로 대체 모델은 최적 함수를 예측 추정
Step3: 획득 함수에서 다음으로 관측할 하이퍼 파라미터 추출
Step4: 해당 하이퍼 파라미터로 관측된 값을 기반으로 대체 모델은 다시 최적 함수 예측 추정
HyperOpt 개요
베이지안 최적화를 구현한 주요 패키지
- HyperOpt
- Bayesian optimization
- Optuna
HyperOpt를 통한 최적화 예시
Search Space (입력 값 범위)
search_space = {'x': hp.quniform('x',5,15,1),'y':hp.uniform('y'0.01,0.1)}목적 함수
def objective_func(search_space):
x = search_space['x']
y = search_space['y']
print('x:', x, 'y:', y)
return {'loss': x ** 2 + y*20, 'status:' STATUS_OK }목적 함수 반환 최솟/최댓값 유추
best = fmin(fn=objective_func, space=search_sapce, algo=algo, max_evals=5, trials=trials)HyperOpt 실습
HyperOpt 설치
pip install hyperopthyperOpt 기본 실습
검색 공간을 설정함
- 검색 공간은 아래 나오는 목적 함수(objective_func())의 인자로 들어감
- x, y라는 입력 변수를 설정
- 이것들이 앞에서부터 순차적으로 들어가는 건 아님
from hyperopt import hp
# -10 ~ 10까지 1간격을 가지는 입력 변수 x 집합값 설정 (-10, -9, -8, ..., 0, 1, ... 9, 10)
# -15 ~ 15까지 1간격을 가지는 입력 변수 y 집합값 설정 (-15, -14, ... , 0, 1, ..., 14, 15)
search_space = {'x': hp.quniform('x', -10, 10, 1), 'y': hp.quniform('y', -15, 15, 1) }
search_space{'x': <hyperopt.pyll.base.Apply at 0x25aff0f8eb0>,
'y': <hyperopt.pyll.base.Apply at 0x25afc5ab3d0>}목적 함수를 설정함
- 검색 공간을 인자로 받음
- retval의 식은 최적 함수로 인데 이건 예제니까 이 식이 들어간 거임 (실제로는 평가 지표 기반으로 들어감)
- 저 식이 최소가 되려면 가 되어야 하는 사실을 기억
from hyperopt import STATUS_OK
# 목적 함수를 생성. 입력 변수값과 입력 변수 검색 범위를 가지는 딕셔너리를 인자로 받고 특정 값을 반환
def objective_func(search_space):
x = search_space['x']
y = search_space['y']
retval = x**2 - 20*y # 이건 예제니까 이 식이 들어간 거임
return retval # return {'loss': retval, 'status':STATUS_OK}베이지안 최적화 진행
trial_val객체는 아래에서 구조를 뜯어보기로 함
fmin함수를 이용해서 최적화 진행
tpe는 최적화 방식 중 하나 (가우시안 최적화, ... 등)
rstate실제로는 안 써주는 게 더 성능이 좋다고 함 (여기서는 결과값 고정을 위해 써줌)
max_evals= 5 만큼 돌면서 최적값 찾기
from hyperopt import fmin, tpe, Trials
import numpy as np
# 입력 결괏값을 저장한 Trials 객체값 생성.
trial_val = Trials()
# 목적 함수의 최솟값을 반환하는 최적 입력 변숫값을 5번의 입력값 시도(max_evals=5)로 찾아냄.
best_01 = fmin(fn=objective_func, space=search_space, algo=tpe.suggest, max_evals=5
, trials=trial_val, rstate=np.random.default_rng(seed=0)
)
print('best:', best_01)100%|█████████████████████████████████████████████████████████████| 5/5 [00:00<00:00, 357.22trial/s, best loss: -224.0]
best: {'x': -4.0, 'y': 12.0}목적함수의 최솟값을 반환하는 최적 입력 변숫값으로 가 나옴
딱 맞진 않았지만 어느정도 근사했음
max_evals = 20으로 늘려서 다시 해보자
trial_val = Trials()
# max_evals를 20회로 늘려서 재테스트
best_02 = fmin(fn=objective_func, space=search_space, algo=tpe.suggest, max_evals=20
, trials=trial_val, rstate=np.random.default_rng(seed=0))
print('best:', best_02)100%|██████████████████████████████████████████████████████████| 20/20 [00:00<00:00, 1176.21trial/s, best loss: -296.0]
best: {'x': 2.0, 'y': 15.0}로 훨씬 더 가까워짐
trial_val의 구조를 뜯어보자
HyperOpt 수행 시 적용된 입력 값들과 목적 함수 반환값 보기
# fmin( )에 인자로 들어가는 Trials 객체의 result 속성에 파이썬 리스트로 목적 함수 반환값들이 저장
# 리스트 내부의 개별 원소는 {'loss':함수 반환값, 'status':반환 상태값} 와 같은 딕셔너리
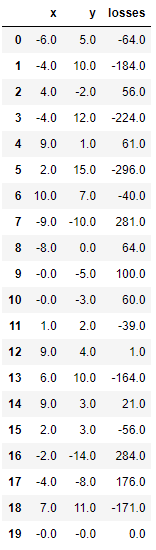
print(trial_val.results)[{'loss': -64.0, 'status': 'ok'}, {'loss': -184.0, 'status': 'ok'}, {'loss': 56.0, 'status': 'ok'}, {'loss': -224.0, 'status': 'ok'}, {'loss': 61.0, 'status': 'ok'}, {'loss': -296.0, 'status': 'ok'}, {'loss': -40.0, 'status': 'ok'}, {'loss': 281.0, 'status': 'ok'}, {'loss': 64.0, 'status': 'ok'}, {'loss': 100.0, 'status': 'ok'}, {'loss': 60.0, 'status': 'ok'}, {'loss': -39.0, 'status': 'ok'}, {'loss': 1.0, 'status': 'ok'}, {'loss': -164.0, 'status': 'ok'}, {'loss': 21.0, 'status': 'ok'}, {'loss': -56.0, 'status': 'ok'}, {'loss': 284.0, 'status': 'ok'}, {'loss': 176.0, 'status': 'ok'}, {'loss': -171.0, 'status': 'ok'}, {'loss': 0.0, 'status': 'ok'}]Trials 객체의 vals 속성에 {'입력변수명':개별 수행 시마다 입력된 값 리스트} 형태로 저장
print(trial_val.vals){'x': [-6.0, -4.0, 4.0, -4.0, 9.0, 2.0, 10.0, -9.0, -8.0, -0.0, -0.0, 1.0, 9.0, 6.0, 9.0, 2.0, -2.0, -4.0, 7.0, -0.0], 'y': [5.0, 10.0, -2.0, 12.0, 1.0, 15.0, 7.0, -10.0, 0.0, -5.0, -3.0, 2.0, 4.0, 10.0, 3.0, 3.0, -14.0, -8.0, 11.0, -0.0]}보기가 힘드니까 각 반복마다의 x값, y값, loss값을 묶어서 출력하는 함수를 만들어보자
import pandas as pd
# results에서 loss 키값에 해당하는 밸류들을 추출하여 list로 생성
losses = [loss_dict['loss'] for loss_dict in trial_val.results]
# DataFrame으로 생성.
result_df = pd.DataFrame({'x': trial_val.vals['x'],
'y': trial_val.vals['y'],
'losses': losses
}
)
result_dfindex:5 에서 최소의 losses가 찾아진 것 같음
5번만 돌렸을 때는 index:4 까지만 돌아서 index:3이 최소의 losses로 찾아진 듯
hyperOpt XGBoost 적용 실습
위스콘신 유방암 데이터 사용
train/val/test 데이터 분리까지 진행
import pandas as pd
import numpy as np
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
import warnings
warnings.filterwarnings('ignore')
dataset = load_breast_cancer()
cancer_df = pd.DataFrame(data=dataset.data, columns=dataset.feature_names)
cancer_df['target']= dataset.target
X_features = cancer_df.iloc[:, :-1]
y_label = cancer_df.iloc[:, -1]
# 전체 데이터 중 80%는 학습용 데이터, 20%는 테스트용 데이터 추출
X_train, X_test, y_train, y_test=train_test_split(X_features, y_label,
test_size=0.2, random_state=156 )
# 학습 데이터를 다시 학습과 검증 데이터로 분리
X_tr, X_val, y_tr, y_val= train_test_split(X_train, y_train,
test_size=0.1, random_state=156 )Search Space 설정
- max_depth:5에서 20까지 1간격
- min_child_weight는 1에서 2까지 1간격으로
- colsample_bytree: 0.5에서 1사이
- learning_rate는 0.01에서 0.2사이 정규 분포된 값으로 검색
- quniform: 정수형 값을 반환할 때 사용 (start, stop(포함), step)
- uniform: 최솟값부터 최댓값까지 정규분포 검색 공간 설정
- loguniform: exp(uniform(low,high)) 값 반환
from hyperopt import hp
xgb_search_space = {'max_depth': hp.quniform('max_depth', 5, 20, 1),
'min_child_weight': hp.quniform('min_child_weight', 1, 2, 1),
'learning_rate': hp.uniform('learning_rate', 0.01, 0.2),
'colsample_bytree': hp.uniform('colsample_bytree', 0.5, 1)
}목적 함수 (objective func) 설정
- fmin()에서 입력된 search_space값으로 입력된 모든 값은 실수형
- max_depth, min_child_weight는 정수가 들어와야 해서 int로 형변환
- 정확도는 높은 수록 더 좋은 수치니까 로 큰 정확도 값일 수록 최소가 되도록 변환
- 참고) 회귀에서 MAE, RMSE, ... 평가지표들은 작을수록 좋은거라 -1 안 곱해도 됨 ( 제외)
- 교차 검증의 평균 정확도에 -1을 곱한 값을 반환
- 위에 기본 실습에서는 임의의 식을 지정했었음, 그것과 비교해보기
def objective_func(search_space):
# 수행 시간 절약을 위해 n_estimators는 100으로 축소
xgb_clf = XGBClassifier(n_estimators=100, max_depth=int(search_space['max_depth']),
min_child_weight=int(search_space['min_child_weight']),
learning_rate=search_space['learning_rate'],
colsample_bytree=search_space['colsample_bytree'],
eval_metric='logloss')
accuracy = cross_val_score(xgb_clf, X_train, y_train, scoring='accuracy', cv=3)
return {'loss':-1 * np.mean(accuracy), 'status': STATUS_OK}베이지안 최적화 진행
algo = tpe.suggest는 거의 고정으로 생각하면 됨
max_evals=50으로 최대 반복 횟수 지정
from hyperopt import fmin, tpe, Trials
trial_val = Trials()
best = fmin(fn=objective_func,
space=xgb_search_space,
algo=tpe.suggest,
max_evals=50,
trials=trial_val, rstate=np.random.default_rng(seed=9))
print('best:', best)100%|███████████████████████████████████████████████| 50/50 [00:14<00:00, 3.52trial/s, best loss: -0.9670616939700244]
best: {'colsample_bytree': 0.5424149213362504, 'learning_rate': 0.12601372924444681, 'max_depth': 17.0, 'min_child_weight': 2.0}베이지안 최적화로 뽑은 최적의 하이퍼 파라미터 조합 출력
print('colsample_bytree:{0}, learning_rate:{1}, max_depth:{2}, min_child_weight:{3}'.format(
round(best['colsample_bytree'], 5), round(best['learning_rate'], 5),
int(best['max_depth']), int(best['min_child_weight'])))colsample_bytree:0.54241, learning_rate:0.12601, max_depth:17, min_child_weight:2여러 평가 지표를 출력하는 함수
get_clf_eval만듦
from sklearn.metrics import confusion_matrix, accuracy_score
from sklearn.metrics import precision_score, recall_score
from sklearn.metrics import f1_score, roc_auc_score
def get_clf_eval(y_test, pred=None, pred_proba=None):
confusion = confusion_matrix( y_test, pred)
accuracy = accuracy_score(y_test , pred)
precision = precision_score(y_test , pred)
recall = recall_score(y_test , pred)
f1 = f1_score(y_test,pred)
roc_auc = roc_auc_score(y_test, pred_proba)
print('오차 행렬')
print(confusion)
print('정확도: {0:.4f}, 정밀도: {1:.4f}, 재현율: {2:.4f},\
F1: {3:.4f}, AUC:{4:.4f}'.format(accuracy, precision, recall, f1, roc_auc))뽑은 최적의 파라미터들로 XGBClassifier에 넣어서 fit() 함
n_esimators=400으로 설정하고early_stopping_rounds=50설정
평가지표까지 출력
xgb_wrapper = XGBClassifier(n_estimators=400, learning_rate=round(best['learning_rate'], 5),
max_depth=int(best['max_depth']), min_child_weight=int(best['min_child_weight']),
colsample_bytree=round(best['colsample_bytree'], 5)
)
evals = [(X_tr, y_tr), (X_val, y_val)]
xgb_wrapper.fit(X_tr, y_tr, early_stopping_rounds=50, eval_metric='logloss',
eval_set=evals, verbose=True)
preds = xgb_wrapper.predict(X_test)
pred_proba = xgb_wrapper.predict_proba(X_test)[:, 1]
get_clf_eval(y_test, preds, pred_proba)[0] validation_0-logloss:0.58942 validation_1-logloss:0.62048
[1] validation_0-logloss:0.50801 validation_1-logloss:0.55913
[2] validation_0-logloss:0.44160 validation_1-logloss:0.50928
[3] validation_0-logloss:0.38734 validation_1-logloss:0.46815
[4] validation_0-logloss:0.34224 validation_1-logloss:0.43913
[5] validation_0-logloss:0.30425 validation_1-logloss:0.41570
[6] validation_0-logloss:0.27178 validation_1-logloss:0.38953
[7] validation_0-logloss:0.24503 validation_1-logloss:0.37317
...
[230] validation_0-logloss:0.01331 validation_1-logloss:0.22709
[231] validation_0-logloss:0.01329 validation_1-logloss:0.22694
[232] validation_0-logloss:0.01327 validation_1-logloss:0.22706
[233] validation_0-logloss:0.01325 validation_1-logloss:0.22676
[234] validation_0-logloss:0.01324 validation_1-logloss:0.22773
[235] validation_0-logloss:0.01322 validation_1-logloss:0.22743
오차 행렬
[[35 2]
[ 2 75]]
정확도: 0.9649, 정밀도: 0.9740, 재현율: 0.9740, F1: 0.9740, AUC:0.9944235에서 멈춘 것 보니까 235-50=185에서의 loss값보다 작은 값을
186~235에서 못 찾은 듯, 관련 설명은 이전 글 참고
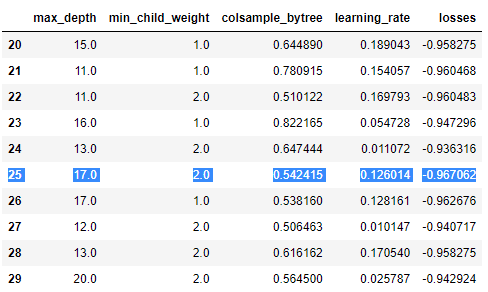
최종적으로 각 하이퍼 파라미터와 losses 값을 묶어서 DataFrame으로 출력
losses = [loss_dict['loss'] for loss_dict in trial_val.results]
result_df = pd.DataFrame({'max_depth': trial_val.vals['max_depth'],
'min_child_weight': trial_val.vals['min_child_weight'],
'colsample_bytree': trial_val.vals['colsample_bytree'],
'learning_rate': trial_val.vals['learning_rate'],
'losses': losses
}
)
result_df[20:30]50번의 반복 중에서 index:25에서 최소의 losses를 찾은 것 같음
솔직히 좀 헷갈린다. 다시 보기..