잘못된 내용 있으면 정정해주세요..
로지스틱 회귀 개요
로지스틱 회귀 개요
로지스틱 회귀는 이름은 회귀인데, 분류에 사용됨
종속변수가 범주형 변수일 때 사용하는 것이 로지스틱 회귀
시그모이드 함수
우선 시그모이드 함수를 먼저 알아보자 (로지스틱 함수라고도 함)
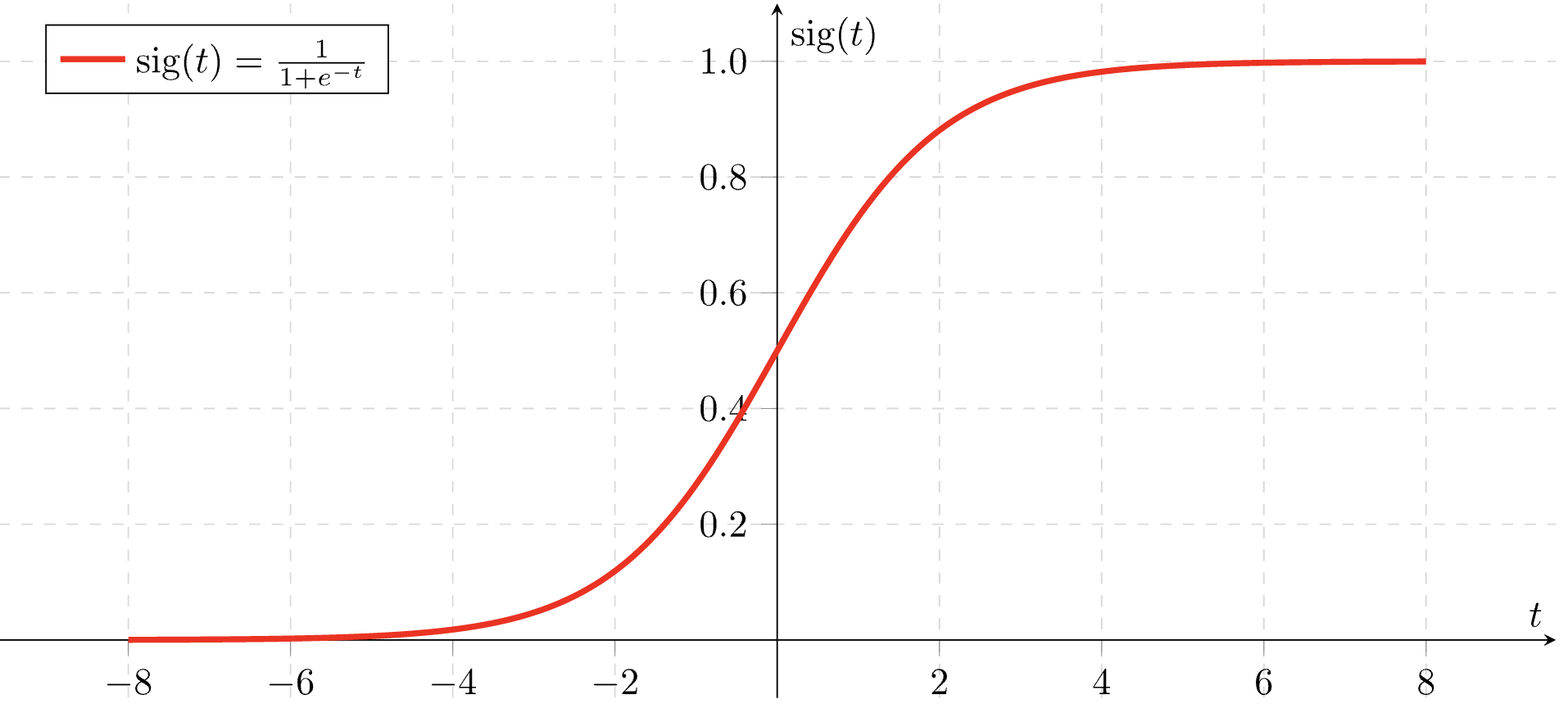
선형이 아닌 S-커브의 함수
보이는 것처럼 범위를 가짐
오즈 (odds)와 로짓 (logit)
오즈는 승산이라고도 함
오즈: 발생하지 않을 확률 대비 일어난 확률의 비율
발생할 확률을 라고 했을 때, 오즈는 다음과 같음
일 때,
일 때,
즉, 범위를 가짐
로짓(logit)은 odds에 log 변환한 것임
따라서, 범위를 가짐
그래서 이게 왜 필요한데?
다중 선형 회귀 식을 보자
여기서 가 범주형이면 좌변과 우변의 범위가 맞지 않음
(우변의 범위는 무한한데 좌변의 범위는 이진 분류이면 또는 )
그래서 를 으로 바꿔주고 최종적으로 확률에 대한 식으로 나타내는 것이 목표!
그럼 다음과 같이 나온다.
이걸 에 대한 식으로 변형하면 다음과 같다.
우리가 위에서 봤던 시그모이드 함수 꼴이다!
따라서, 독립변수와 회귀계수에 따라서 종속변수 가 0과 1사이 값으로 나오게 되고,
분류를 할 때는 일반적으로 0.5를 기준으로 설정하면 되는 것!
로지스틱 회귀 기타 특징
- 로지스틱 회귀는 주료 이진 분류로 사용 (다중도 가능)
- 가볍고 빠르며 희소한 데이터 세트 분류에 성능이 좋음 (텍스트 분류)
- 단, 요즘 XGBoost, LightGBM의 성능이 좋아서 주로 사용되진 않는 듯?
로지스틱 회귀 실습
scikit-learn에서는 LogisticRegression 클래스로 지원
주요 하이퍼 파라미터
- penalty: 규제 유형 설정 (l1, l2)
-> l1: L1 규제 적용
-> l2: L2 규제 적용 - solver: 회귀 계수 최적화 방식 지정
-> 이전에는 libnear가 기본, 지금은 lbfgs가 기본
-> lbfgs: 메모리 공간 절약, CPU 코어 수 많으면 최적화를 병렬로 수행
-> liblinear: 다차원이고 작은 데이터 세트에서 효과적으로 동작
-> 국소 최적화 (Local Minimum) 이슈 있음
-> 병렬로 최적화 불가
-> 그외 newtron-cg, sag, saga 있는데 성능에 큰 차이 없어서 변경할 일 없음 - C: 규제 강도 조절
-> 의 역수
-> 그러니까 작을수록 규제 강도가 큼
cf) lbfgs는 L2 규제만 됨
유방암 데이터 사용
import pandas as pd
from sklearn.datasets import load_breast_cancer
from sklearn.linear_model import LogisticRegression
cancer = load_breast_cancer()LogisticRegression은 스케일링이 선수되어야 함
from sklearn.preprocessing import StandardScaler
from sklearn.model_selection import train_test_split
scaler = StandardScaler()
feature_scaled = scaler.fit_transform(cancer.data)
X_train, X_test, y_train, y_test = train_test_split(feature_scaled, cancer.target, test_size = 0.2, random_state = 12)scikit-learn 1.0.2 기준으로
solver='lbfgs'가 기본값
from sklearn.metrics import roc_auc_score
lrc = LogisticRegression() # sklearn 1.0.2 기준 solver='lbfgs' 기본값
lrc.fit(X_train, y_train)
pred = lrc.predict(X_test)
score = roc_auc_score(y_test, pred)
print('ROC AUC Score: ', round(score,4))ROC AUC Score: 0.9792
solver간 Score 비교
solvers = ['lbfgs', 'liblinear', 'newton-cg', 'sag', 'saga']
for solver in solvers:
lrc = LogisticRegression(solver = solver, max_iter = 500)
lrc.fit(X_train, y_train)
pred = lrc.predict(X_test)
score = roc_auc_score(y_test, pred)
print(solver, 'ROC AUC Score: ', round(score,10))lbfgs ROC AUC Score: 0.9791666667
liblinear ROC AUC Score: 0.9791666667
newton-cg ROC AUC Score: 0.9791666667
sag ROC AUC Score: 0.9791666667
saga ROC AUC Score: 0.9791666667현재 환경에서는 solver간 ROC AUC Score 차이 없음
-> 실제로 solver가 크게 중요하지 않음
GridSearchCV 진행
from sklearn.model_selection import GridSearchCV
params = {'solver': ['liblinear', 'lbfgs'], 'penalty': ['l1', 'l2'], 'C': [0.01, 0.1, 1, 5, 10]}
lrc = LogisticRegression()
lrc_grid = GridSearchCV(lrc, param_grid = params, scoring='roc_auc', cv = 5)
lrc_grid.fit(feature_scaled, cancer.target)
print('최적 하이퍼 파라미터:', lrc_grid.best_params_, '\nROC AUC Score:', round(lrc_grid.best_score_, 4))최적 하이퍼 파라미터: {'C': 0.1, 'penalty': 'l2', 'solver': 'liblinear'}
ROC AUC Score: 0.9954
lbfgs는 l1 규제 적용이 안 돼서 오류 발생함
그외의 것들 중 최적 하이퍼 파라미터 조합을 찾은 듯
