[Paper Review] Handcrafted Histological Transformer (H2T): Unsupervised Representation of Whole Slide Images
Paper Review
Handcrafted Histological Transformer (H2T): Unsupervised Representation of Whole Slide Images

Introduction
HoverNet의 저자인 Quoc Dang Vu가 작성한 논문으로, SwAV 이라는 self-supervised learning 방식을 사용하여 WSI 의 각 patch 의 prototype vector 를 생성하고, 이 prototype vector 로 이루어진 representation space 에 각 WSI 의 patch 를 embedding 하여 feature 를 생성하였다. WSI 내 patch 간의 위치 정보도 활용할 수 있도록 pattern assignment map 을 활용하여 patch 정보를 WSI 단위로 aggregate 하였고, 이를 downstream analysis 에 활용하였다. patch 정보를 WSI 단위 정보로 aggregate 하는 과정에서 transformer 의 attention mechanism 과 유사한 방법이 사용되어, handcrafted histological transformer (H2T) 라 주장한다. 또한 interpretability 와 improvement in predictive power 사이의 trade-off 를 적절히 극복한 방법이라 주장한다.
Method
Handcrafted histological transformer (H2T)
Transformer 와 그 동작 원리를 밝힌 연구에 영감을 받아, H2T 를 설계하였다. H2T 는 two stage 로 이루어져 있다.
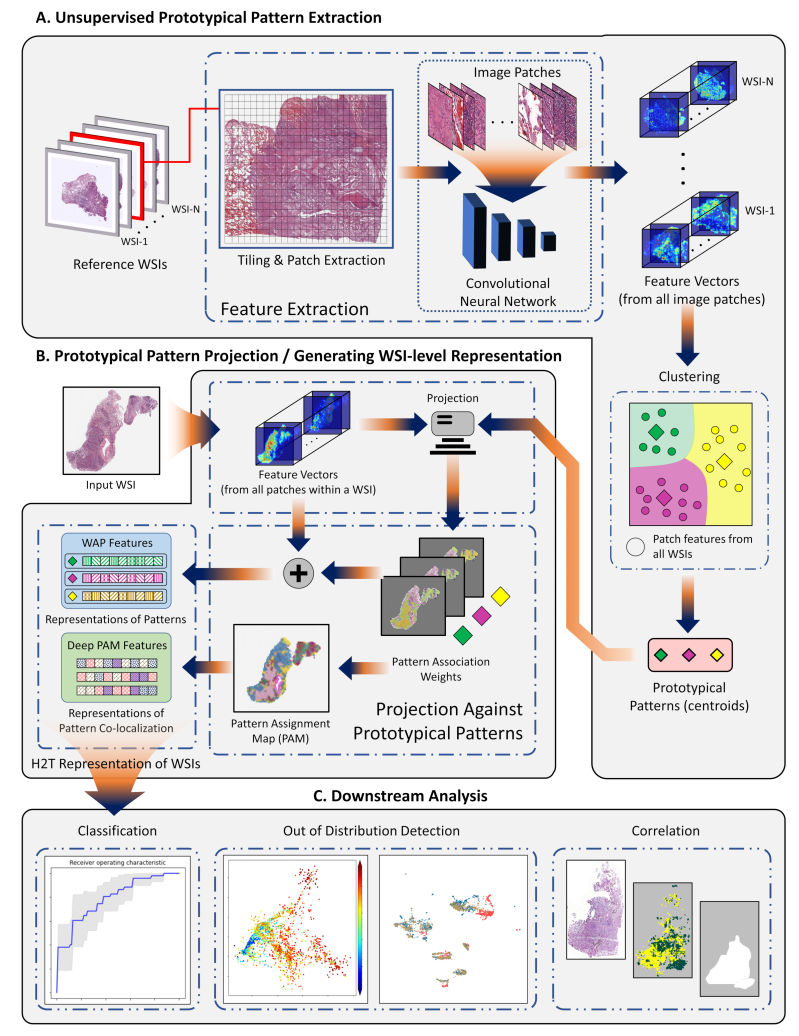
a. Construction of the prototypical patterns
b. Projection against these patterns
first stage 에서 여러 source 로부터 얻은 충분한 양의 reference WSI 를 patch 화 하여 prototypical pattern 을 추출한다. 특히 전에 리뷰했던 SwAV 라는 Self-supervised learning 방식, scalable online clustering, 을 활용하여 prototypical pattern (centroids) 를 계산한다. second stage 에서 새로운 WSI 를 patch 화 하여 prototypical pattern 에 대해 projection 하고, constituent instance 와 instance 가 assign된 pattern 간의 relationship 을 기반으로 한, WSI 단위의 summarized information 을 생성한다.
Multi head self-attention
transformer 의 multi head self-attention 에 대해 설명하고, 저자가 제안한 메커니즘과 synonuymous 임을 주장한다. 이번 섹션의 경우, 이해가 잘 되지는 않았다.. 다음은 K Q V relationship 을 나타내는 formulation 이다.
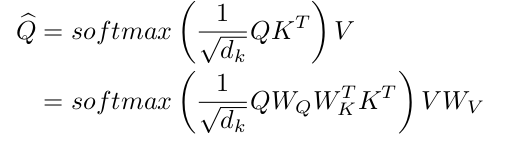
Hopfield is all you need 논문을 참조했을 때, MHA와 Hopfield neural network 는 큰 관련이 있고, K 와 V 에 대해 same input Y 와 Q 를 rename 한 R 을 적용하면 다음과 같은 식을 얻을 수 있고, 이는 몇가지 흥미로운 properties 를 보인다 주장한다. (음.. 1x1 convolution 으로 channel 수를 늘리고 이를 Q, K, V 로 활용하는 것이 아니었나? Q와 K의 attention를 V에 곱해주는게.. 아 Y(instance)랑 R(prototypical vector)와의 유사도를 보는건가? 흠..)
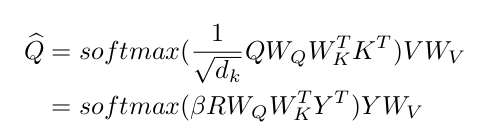
First, 위 식은 (R을 reference 삼아) inputs R과 Y 사이의 association 을 찾는 것과 동치이다. Second, scaling factor beta는 모델의 memorization 과 association capacity 를 결정 짓기에 중요한 요소이다. Finally, R이 trainable 이라 했을 때, traing set으로 부터 prototypical patterns P 의 집합을 배우는 아키텍쳐를 효과적으로 얻을 수 있다. (...)
Positional encoding
위에서 언급한 식을 보면, instance 간의 관계는 permutation invariant 하다. 하지만 instance 의 ordering 즉 position 은 중요한 정보이기에, MHA 에서 푸리에 인코딩이나 sine-encoding 방식을 사용하여 positional encoding을 해준다. (이후 sine, cosine 을 이용한 positional encoding 설명)
Handcrafted prototypical patterns
위 handcrafted histological transformer (H2T) 의 second stage 를 다시 두 가지로 나누면 다음과 같다.
1) WSI 를 patch 호 하여 prototypical pattern 에 projection 한다.
2) constituent instance 와 instance 가 assign 된 pattern 간의 relationship 을 기반으로 한 WSI 단위의 summarized info 를 생성한다.
1) representation from histological patterns
Prototypical patterns 를 SwAV 방식으로 뽑아내고, WSI 를 어떻게 projection 하느냐에 대한 설명이 담겨있다. (처음에 이해하느라 힘들었다)

WSI 를 patch 화 하여 (적절한 backbone 을 거친) feature vector 와 모든 prototypical vector 간의 distance 를 계산하여, minimum distance 를 가진 pattern 에 assign 된다. 우선 는 각 pattern 에 assign 된 instance 의 개수를 의미한다. 는 p와 psi 사이의 similarity 를 measure 해주는 attribution function 이다. 따라서 는 i 번째 pattern 에 속한 vector 들이 얼마나 centroid 와 가까운지를 보여주는 단합력을 기반으로 instance 끼리의 정보를 합한 pattern 의 weight, importance 라 생각할 수 있다. 이를 각 pattern 별로 concat 하여 WSI 단위의 H 를 생성하는 것이다.
이 때, , attribution function 을 다음과 같은 방식으로 다양하게 구성하여 실험을 진행하였다.


2) representation from co-localization of patterns
이제 patch instance 간의 spatial relation 을 기반으로 또 다른 WSI 단위의 정보를 뽑아낸다.
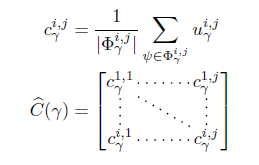
pattern co-localization matrix (PCM) 으로 정의된 다음 matrix 는 값 (radius) 에 따라 각 pattern 끼리 얼마나 가까이에 분포해 있는지에 대한 정보를 담는다. 다음 사진을 보면 더 쉽게 이해할 수 있다.
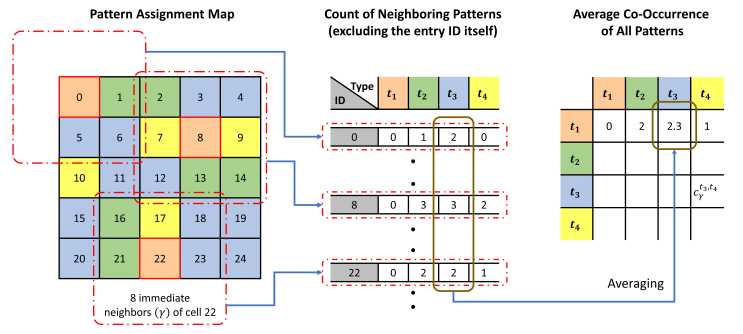
8개의 immediate neighbors 을 기준으로 어떤 pattern 이 몇개가 인접해 있는지를 PCM 으로 나타낸 것이다. 저자는 이 방법을 사용했을 때의 문제점도 지적하고 있다. gamma 값을 어느 수준까지만 높일 수 있다는 unscalable 한 특성을 지적하며, CNN 을 사용할 것도 제안한다. 각 patch instance 의 prototypical pattern 을 assign 했고 patch 의 위치도 알고 있으므로, 그 spatial relation 을 2D CNN 으로 보고 하나의 map, pattern assignment map (PAM) 을 얻을 수 있다고 주장한다. 또한, PAM 은 raw pixel intensity 를 가지지 않고 discrete value 를 가지기 때문에, PAM 을 directly learn 하도록 설계하는 것보다 CNN 을 학습함으로써 Deep PAM feature 를 얻고, 이를 WSI 별로 C 라는 feature 로 추출할 것을 제안하고 있다.
Experimental Results
Datasets
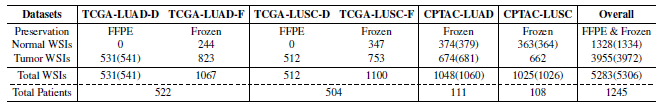
TCGA 와 CPTAC 의 2개의 암종 (LUAD, LUSC, Normal) 의 FFPE, Frozen slide 를 사용하여, 총 1245명의 환자자로부터 5306 장의 WSI 를 사용하였다.
Evaluation
여러 attribution function 을 사용해 WSI 로부터 추출한 H2T feature 는 linear probing 방식으로 downstream task 에 적용되어 성능을 측정하였다. CPTAC 와 TCGA 둘 중 하나를 training set 으로 사용하고, 나머지를 validation set 으로 활용하였다. 구체적으로 LUAD, LUSC, normal 사이를 classify 하는 task 를 5-fold crss validation 하여 수치를 기록하였다.
Implementation details
size 512x512 with 256x256 overlapping 방식으로 patch를 추출하였고, pretrained ResNet50 으로 기본 representation 을 뽑아내었다. 40x와 20x magnification 을 가진 WSI 를 사용하였고, SwAV 으로 하여금 16 x 2048 vector (16개의 cluster, 2048-dimensional) 를 학습하도록 설계하였다.
Prototypical patterns
further assessment 이전에 SwAV prototypical pattern 의 sanity check 를 수행하였다. 4개의 set (normal, LUAD, LUSC, all) 의 TCGA WSI 와 SwAV-ResNet50 을 이용하여 16 prototypical pattern 을 뽑아내었다. 이후 pattern assignment 를 수행하고, pathologist annotation 과 비교하였다. train 은 TCGA 로 했지만 PAM 은 ACDC dataset 을 이용하여 map 을 그려보았다.
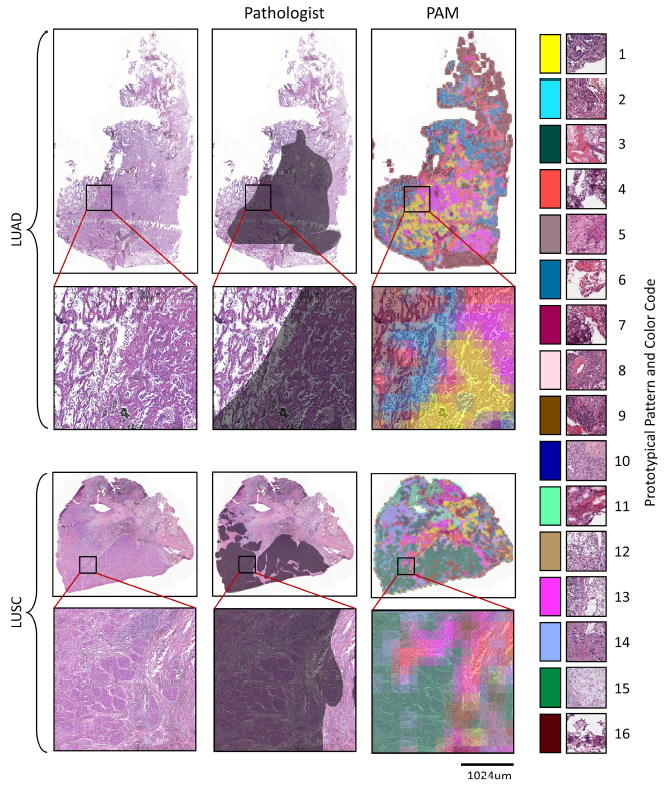
6번과 16번 pattern 이 normal 과 관련을 보였고, 1번, 13번 은 LUAD tumor, 15번은 LUSC tumor pattern 과 연관성을 보였다. LUAD, LUSC 와 pathologist 의 annotation 과 각각 0.6879, 0.8407 의 Pearson Correlation Coefficient 를 기록하였다.
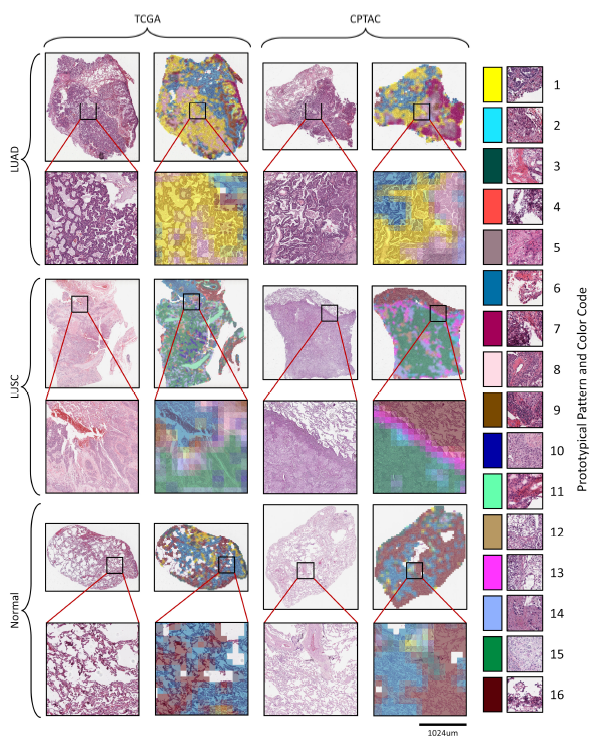
같은 prototypical pattern 으로 reference cohort TCGA, unseen cohort CPTAC setting 에서 3 dataset LUAD, LUSC, normal 을 visualization 하였다. 결과 tumorous, stromal region 간의 pattern consistency 를 확인할 수 있었다.
Comparative evaluation
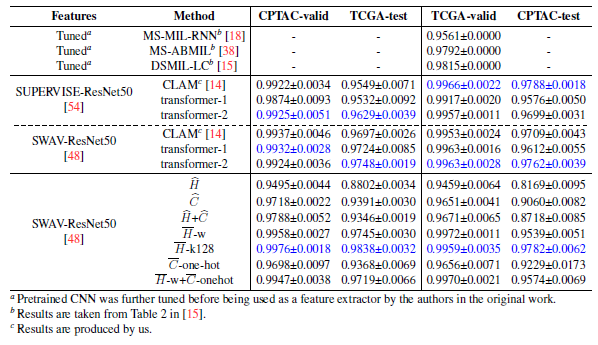
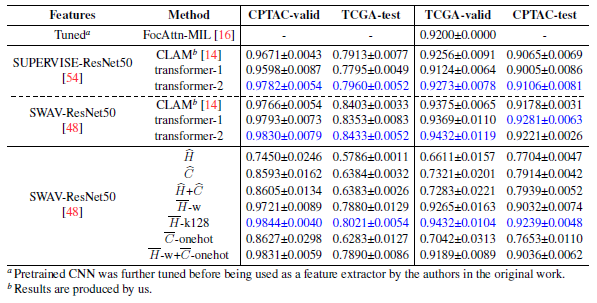
normal vs tumor, LUAD vs LUSC, Normal vs LUAD vs LUSC WSI classification task 결과 5 fold AUROC (or mAP) 을 나타낸 결과이다.
Abltaion study
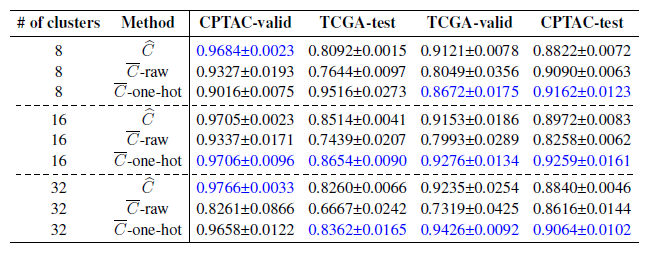
CNN 을 활용하기 전 C 를 활용하는 방법에 따른 성능 수치이다. Normal vs Tumor classification task 이다. C는 co-localization matrix of patterns within PAM, C-raw 는 PAM 을 CNN 으로 학습, C-one-hot 은 pattern 을 one-hot encoding 한 후 CNN 에 넣어 학습한 결과 이다.
Discovery experiments
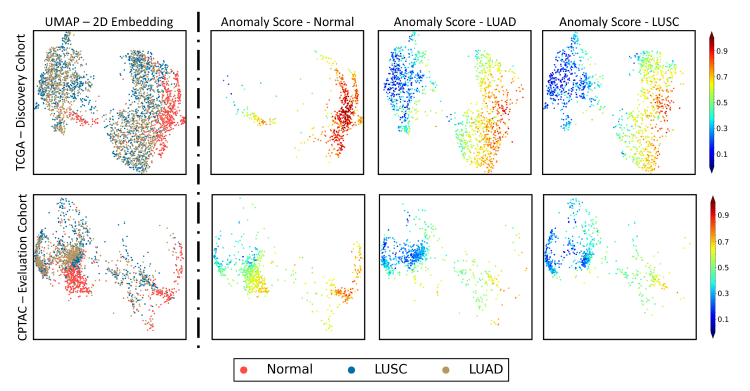
discovering anomalous WSI task 를 잘 수행하는 가도 추가 downstream task 로 진행하였다. 16 x 2048 feature, 즉 high-dimensional vector 를 visualization 하기 위해 UMAP 로 2D plane에 각 WSI 를 나타내었다. 결과 LUAD, LUSC 는 around 0.8 (orange) 에 머물렀지만, normal WSI 는 distinctly high anomaly score 를 가졌다. 또한 unsupervised clustering of WSI 측면에서도 LUAD, LUSC, normal 간 separation 을 관측할 수 있었다.
