이 블로그글은 2019년 조재영(Kevin Jo), 김승수(SeungSu Kim)님의 딥러닝 홀로서기 세미나를 수강하고 작성한 글임을 밝힙니다.
Github : 실습 코드 링크
Pytorch Regression (Linear Regression vs MLP)
💡목표 : Regression Problem을 pytorch로 해결해보기
- Linear Regression과 MLP를 둘 다 구현해보고 모델의 예측 데이터 분포를 시각화하기
# Pytorch 불러오기
import torch
print(torch.__version__)# 나머지 모듈 불러오기
import numpy as np
import matplotlib.pyplot as plt데이터 생성 (정의)
데이터셋 설명
- 아래의 데이터 분포를 따르고 관측시 발생한 오차 e가 더해져 있는 데이터
- sin(x) 함수와 log(x)함수는 모두 non-linear 함수
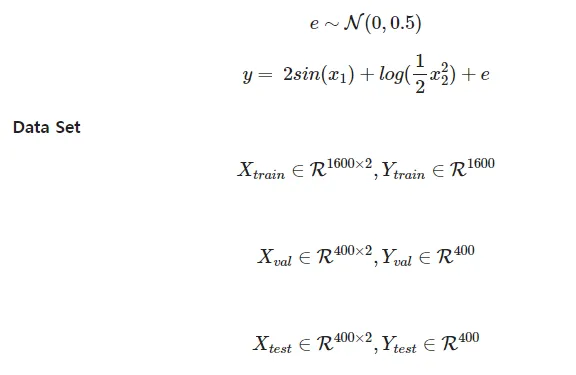
데이터셋 정의
num_data = 2400
x1 = np.random.rand(num_data) * 10
x2 = np.random.rand(num_data) * 10
e = np.random.noraml(0, 0.5, num_data)
X = np.array([x1, x2]).T
y = 2*np.sin(x1) + np.log(0.5*x2**2) + e- np.random.rand(num_data) : 0 ~ 1까지 uniform distribution에서 샘플링하는 함수
- np.random.normal(0, 0.5, num_data) : 평균이 0이고 표준편차가 0.5인 normal distribution에서 샘플링하는 함수
💡 참고
pytorch에서 데이터 셋은 첫 번째 차원은 샘플의 개수를 나타내도록 설계되어 있음
데이터셋 split (훈련, 검증, 테스트)
- train set만을 가지고 있는 경우, 학습한 모델을 평가할 방법이 없음
- 훈련 데이터 : 모델을 학습하시키는데 사용되는 데이터
- 검증 데이터 : 학습 도중에 모델의 성능을 평가하고 과적합 여부를 확인하고 하이퍼파라미터를 조정하는데 사용되는 데이터
- 테스트 데이터 : 최종적으로 학습된 모델의 성능을 평가하기 위해 사용되는 데이터
train_X, train_y = X[:1600, :], y[:1600]
val_X, val_y = X[1600:2000, :], y[1600:2000]
test_X, test_y = X[2000:, :], y[2000:]💡 참고
pytorch에서 데이터 셋은 첫 번째 차원은 샘플의 개수를 나타내도록 설계되어 있음
데이터셋 시각화
fig = plt.figure(figsize=(12, 5))
ax1 = fig.add_subplot(1, 3, 1, projection='3d')
ax1.scatter(train_X[:, 0], train_X[:, 1], train_y, c=train_y, cmap='jet')
ax1.set_xlabel('x1')
ax1.set_ylabel('x2')
ax1.set_zlabel('y')
ax1.set_title('Train set Distribution')
ax1.set_zlim(-10, 6)
ax1.view_init(40, -60)
ax1.invert_xaxis()
ax2 = fig.add_subplot(1, 3, 2, projection='3d')
ax2.scatter(val_X[:,0], val_X[:,1], val_y, c=val_y, cmap='jet')
ax2.set_xlabel('x1')
ax2.set_ylabel('x2')
ax2.set_zlabel('y')
ax2.set_title('Validation set Distribution')
ax2.set_zlim(-10, 6)
ax2.view_init(40, -60)
ax2.invert_xaxis()
ax3 = fig.add_subplot(1, 3, 3, projection='3d')
ax3.scatter(test_X[:,0], test_X[:,1], test_y, c=test_y, cmap='jet')
ax3.set_xlabel('x1')
ax3.set_ylabel('x2')
ax3.set_zlabel('y')
ax3.set_title('Test set Distribution')
ax3.set_zlim(-10, 6)
ax3.view_init(40, -60)
ax3.invert_xaxis()
plt.show()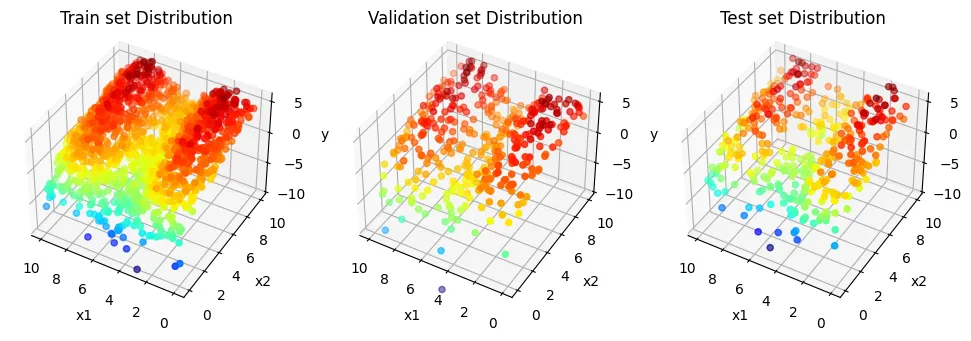
💡 cmap
데이터 시각화에서 색상을 매핑하여 데이터를 표현하는 방법
viridis: 균일하고 색맹 친화적인 그린-블루 계열plasma: 따뜻한 보라-노랑 계열의 그라디언트inferno: 어두운 보라-밝은 노랑, 강한 대비magma: 검정에서 노랑으로 이어지는 연속형 컬러맵cividis: 파랑-노랑, 색맹 친화적인 부드러운 전환
Hypothesis 정의 (모델 정의)
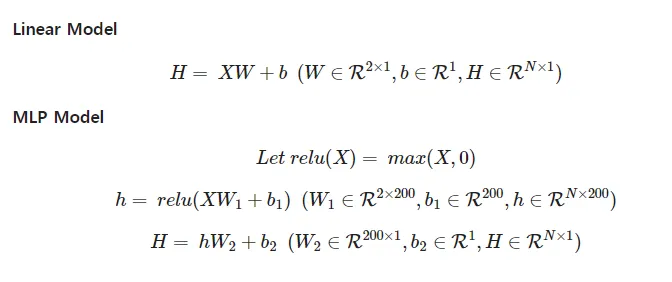
Linear Model
- PyTorch의
nn.module을 상속받아 새로운 신경망 모델 클래스를 정의 __init__은 클래스 초기화 메서드 (생성자)- super를 통해 부모 클래스의 초기화를 호출하여 상속받은 속성들을 초기화
- 선형 레이어 정의
- 입력 크기 : 2, 출력 크기 : 1, bias가 있음
forward는 입력 x를 받아 선형 변환을 적용하는 출력값을 반환하는 메서드
import torch.nn as nn
class LinearModel(nn.Module):
def __init__(self):
super(LinearModel, self).__init__()
self.linear = nn.Linear(in_features=2, out_features=1, bias=True)
def foward(self, x):
return self.linear(x)MLPModel
- PyTorch의
nn.Module을 상속받아 다층 퍼셉트론 모델 클래스를 정의 __init__은 클래스 초기화 메서드(생성자)- super를 통해 부모 클래스의 초기화를 호출하여 상속받은 속성들을 초기화
- 첫 번째 선형 레이어 정의
- 입력 크기 : 2, 출력 크기 : 200
- 두 번째 선형 레이어 정의
- 입력 크기 : 200, 출력 크기 : 1
- 활성화 함수로 ReLU 정의
forward는 입력 x를 받아 순전파(forward pass)를 수행하는 메서드- 첫 번째 선형 레이어를 통해 입력을 변환
- 변환된 값에 ReLU 활성화 함수를 적용하여 비선형성을 추가
- 두 번째 선형 레이어를 통해 최종 출력을 생성하고 반환
class MLPModel(nn.Module):
def __init__(self):
super(MLPModel, self).__init__()
self.linear1 = nn.Linear(in_features=2, out_features=200)
self.linear2 = nn.Linear(in_features=200, out_features=1)
self.relu = nn.ReLU()
def forward(self, x):
x = self.linear1(x)
x = self.relu(x)
x = self.linear2(x)
return xCost Function 정의
- Pytorch의 nn 모듈에 구현된 Loss Function이 이미 구현되어 있음
- 여기서는 MSE를 사용함
reg_loss = nn.MSELoss()💡 nn 모듈에 구현된 Loss Function 목록
MSELoss: 회귀 문제에서 평균 제곱 오차 계산.CrossEntropyLoss: 분류 문제에서 교차 엔트로피 계산.BCELoss: 이진 분류에서 이진 교차 엔트로피 계산.L1Loss: 예측 값과 실제 값의 절대 오차 계산.SmoothL1Loss: MSE와 L1 절충하여 작은 값은 MSE, 큰 값은 L1로 처리KLDivLoss: 예측 확률 분포와 실제 확률 분포 사이의 차이를 측정
학습 및 평가
- PyTorch의 loss.backward()라는 기능을 사용하면 알아서 loss를 계산
- 사용한 각 파라미터에 대한 loss의 편미분을 계산해 줌
- optimizer.step() 함수를 사용하여 각 파라미터의 그라디언트를 바탕으로 파라미터의 값을 조금씩 업데이트 해줌
- iteration마다 Train set으로 학습하면서 Validation Set으로 Loss를 비교함
- 최종적으로 모델을 Test Set을 넣고 MAE metric을 사용하여 평가함
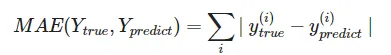
모델 생성
import torch.optim as optim
from sklearn.metrics import mean_absolute_error
#**model = LinearModel()
#print(model.linear.weight)
#print(model.linear.bias)**
model = MLPModel()
print(f'{sum(p.numel() for p in model.parameters() if p.requires_grad)} Parameters')Optimizer 생성
- SGD : 경사하강법 옵티마이저를 사용
- model.parameters()와 같이 업데이트할 파라미터를 넘겨줌
- 학습률 설정
lr = 0.005
optimizer = optim.SGD(model.parameters(), lr=lr)💡 optim 모듈에 구현된 옵티마이저 목록
SGD: 기본 확률적 경사 하강법, 단순하지만 효율적.Adam: 학습률과 모멘텀을 자동 조정, 빠르고 안정적.RMSprop: 학습률을 적응적으로 조정해 진동을 줄임.Adagrad: 학습 횟수에 따라 학습률 감소, 희소한 데이터에 유리.Adadelta: Adagrad의 개선형, 학습률을 무한히 감소하지 않도록 조정.AdamW: Adam과 비슷하지만 가중치 감쇠(weight decay) 적용.ASGD: SGD의 변형으로, 느리지만 더 안정적인 수렴을 목표로 함.
학습 & 검증
Train 부분
model.train(): 모델을 학습 모드로 전환optimizer.zero_grad(): 이전 기울기 초기화loss = reg_loss(pred_y.squeeze(), true_y): 예측 값과 실제 값의 손실 계산squeeze()는 텐서의 차원이 1인 축을 제거
loss.backward(): 역전파로 기울기 계산optimizer.step(): 가중치 업데이트list_train_loss.append(loss.detach().numpy()): 손실 값 저장detach(): 텐서를 연산 그래프에서 분리하여 기울기 계산(역전파)을 막음 (기울기를 추적하거나 업데이트하는 과정에서 제외하고 값만 사용함)
💡연산 그래프
- PyTorch가 텐서와 연산을 연결하여 기울기(gradient)를 계산하기 위해 만든 구조
- 모델이 데이터를 처리할 때 이 그래프가 만들어지고, 역전파를 통해 각 가중치에 대한 기울기를 자동으로 계산
Validate 부분
model.eval(): 모델을 평가 모드로 전환optimizer.zero_grad(): 기울기 초기화loss = reg_loss(pred_y.squeeze(), true_y): 검증 손실 계산list_val_loss.append(loss.detach().numpy()): 손실 값 저장
list_epoch = []
list_train_loss = []
list_val_loss = []
list_mae = []
list_mae_epoch = []
epoch = 4000
for i in range(epoch):
# Trian
model.train()
optimizer.zero_grad()
input_x = torch.Tensor(train_X)
true_y = torch.Tensor(train_y)
pred_y = model(input_x)
loss = reg_loss(pred_y.squeeze(), true_y)
loss.backward()
optimizer.step()
list_epoch.append(i)
list_train_loss.append(loss.detach().numpy())
# Validate
model.eval()
optimizer.zero_grad()
input_x = torch.Tensor(val_X)
true_y = torch.Tensor(val_y)
pred_y = model(input_x)
loss = reg_loss(pred_y.squeeze(), true_y)
list_val_loss.append(loss.detach().numpy())
if i % 200 == 0:
model.eval()
optimizer.zero_grad()
input_x = torch.Tensor(test_X)
true_y = torch.Tensor(test_y)
pred_y = model(input_x).detach().numpy()
mae = mean_absolute_error(true_y, pred_y)
list_mae.append(mae)
list_mae_epoch.append(i)
fig = plt.figure(figsize=(15,5))
ax1 = fig.add_subplot(1, 3, 1, projection='3d')
ax1.scatter(test_X[:, 0], test_X[:, 1], test_y, c=test_y, cmap='jet')
ax1.set_xlabel('x1')
ax1.set_ylabel('x2')
ax1.set_zlabel('y')
ax1.set_zlim(-10, 6)
ax1.view_init(40, -40)
ax1.set_title('True test y')
ax1.invert_xaxis()
ax2 = fig.add_subplot(1, 3, 2, projection='3d')
ax2.scatter(test_X[:, 0], test_X[:, 1], pred_y, c=pred_y[:,0], cmap='jet')
ax2.set_xlabel('x1')
ax2.set_ylabel('x2')
ax2.set_zlabel('y')
ax2.set_zlim(-10, 6)
ax2.view_init(40, -40)
ax2.set_title('Predicted test y')
ax2.invert_xaxis()
input_x = torch.Tensor(train_X)
pred_y = model(input_x).detach().numpy()
ax3 = fig.add_subplot(1, 3, 3, projection='3d')
ax3.scatter(train_X[:, 0], train_X[:, 1], pred_y, c=pred_y[:,0], cmap='jet')
ax3.set_xlabel('x1')
ax3.set_ylabel('x2')
ax3.set_zlabel('y')
ax3.set_zlim(-10, 6)
ax3.view_init(40, -40)
ax3.set_title('Predicted train y')
ax3.invert_xaxis()
plt.show()
print(f'epoch : {i}, mae = {mae}')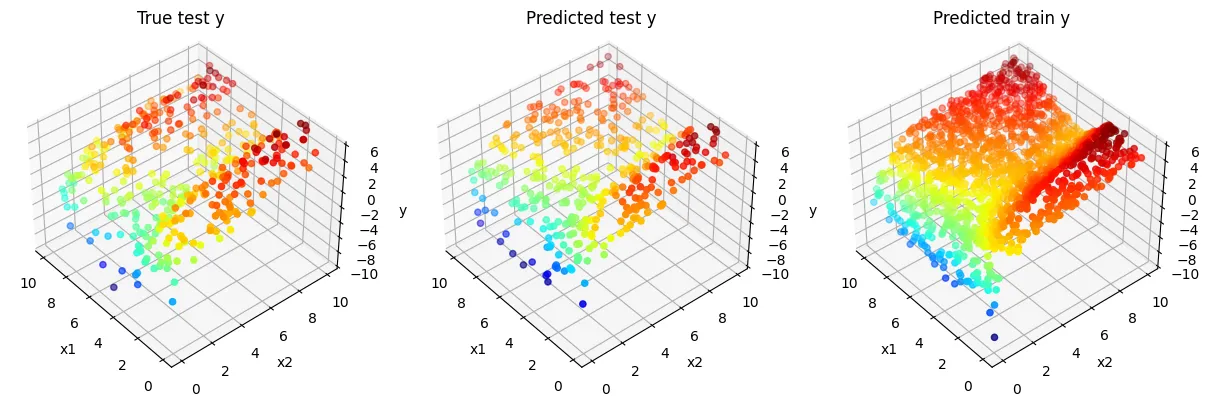
💡 epoch vs iteration
- Epoch: 전체 훈련 데이터를 한 번 다 사용해 학습하는 과정
- Iteration: 미니 배치 단위로 한 번 학습하는 과정
- 1 Epoch = 여러 Iterations, 데이터 크기와 배치 크기에 따라 결정됨
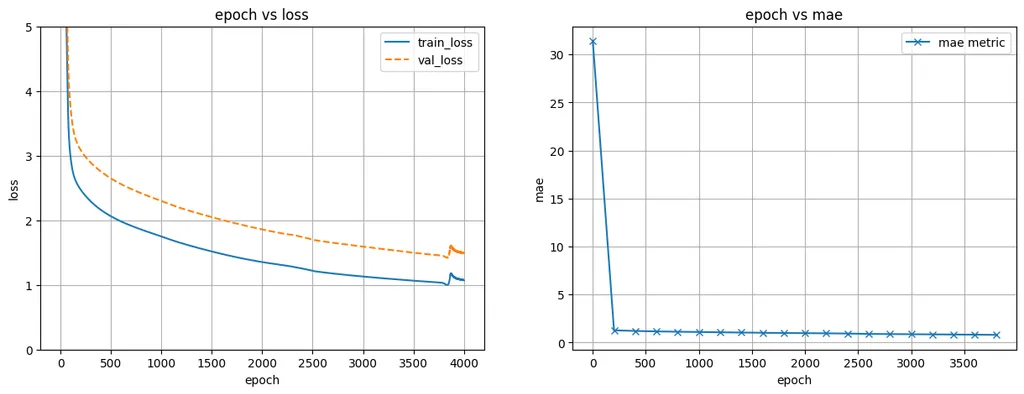
결과
- 첫 번째 그래프는 epoch에 따른 train_loss와 val_loss를 그림
- epoch에 따라 loss가 꾸준히 줄어듦
- 이때 val_loss가 증가한다면 모델의 성능이 나빠짐을 의미
- 두 번째 그래프는 epoch에 따른 mae을 그림
- epoch에 따라 모델의 성능이 얼마나 되는지 평가 가능
# 결과 출력
fig = plt.figure(figsize=(15, 5))
ax1 = fig.add_subplot(1, 2, 1)
ax1.plot(list_epoch, list_train_loss, label='train_loss')
ax1.plot(list_epoch, list_val_loss, '--', label='val_loss')
ax1.set_xlabel('epoch')
ax1.set_ylabel('loss')
ax1.set_ylim(0, 5)
ax1.grid()
ax1.legend()
ax1.set_title('epoch vs loss')
ax2 = fig.add_subplot(1, 2, 2)
ax2.plot(list_mae_epoch, list_mae, marker='x', label='mae metric')
ax2.set_xlabel('epoch')
ax2.set_ylabel('mae')
ax2.grid()
ax2.legend()
ax2.set_title('epoch vs mae')
plt.show()