(2023)SAM-Med3D
Paper Review
0. Abstract
SAM(Segment Anything Model)은 2D natural image segmentation에서는 좋은 성능을 보였지만, 3D volume image에 적용하면 성능이 좋지 못하고 많은 prompt point가 필요하다. SAM의 2D structure가 3D spatial 정보를 반영하지 못하기 때문이다.
본 논문에서는 위와 같은 문제를 해결하기 위해 3D architecture로 재구성하여 large-scale volumetric medical dataset으로 학습한 SAM-Med3D를 제안한다.
1. Introduction
이전 연구에 따르면 SAM을 medical domain에 적용하는 것은 medical image에 대한 knowledge가 부족하기 때문에 제한이 있다.
이 문제의 간단한 해결 방법은 fine-tuning이다. MedSAM은 110만개의 medical image의 mask로 fine-tuning을 하였다. SAM-Med2D는 adapter와 2천만개의 mask를 사용해 fine-tuning을 함으로 좋은 결과를 얻었다.
그러나 이러한 방법들은 volumetric image를 slice-by-slice로 접근한다. 3D data를 2D slice로 나누고 독립적으로 분석한 다음 2D 결과를 3D로 예측하는 방식이다.
몇몇 연구들에서는 2D에서 3D로 적용하기 위한 시도를 했다. 2D layer를 freeze 후, 3D adapter를 학습하여 3D image를 model이 학습할 수 있도록 하였다.
그러나 이 방법에도 두 가지 한계가 있다. 첫 번째로 dataset의 크기와 다양성 측면에서 부족하다. 두 번째로 여전히 2D design paradigm을 고수하여 3D spatial information을 활용하지 못하고 있다. 이러한 한계를 극복하기 위해 본 논문에서는 fully learnable 3D architecture에 large-scale volumetric medical dataset으로 학습한 SAM-Med3D를 제안하였다.
SAM-Med3D의 장점은 두 가지가 있다. 첫 번째로 적은 prompt point가 필요하다. 2D 방법과 비교했을 때 사용자가 좀 더 쉽게 활용할 수 있다. 두 번째로 이전 연구들과 비교했을 때 SAM-Med3D는 다양한 target과 modality에서 좋은 성능을 보여준다.
2. Related Works
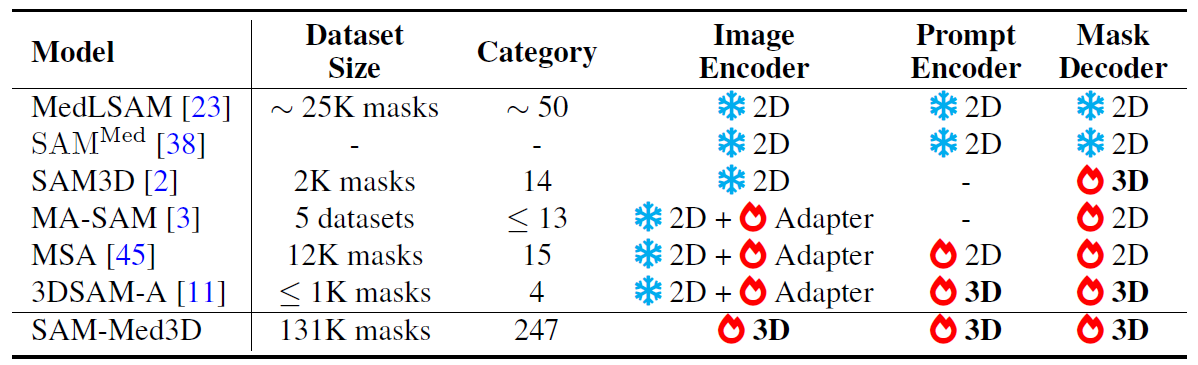
- MedLSAM: Prompt를 정확하게 주기 위해서 localization model을 사용한 two-stage model
- SAM3D: SAM의 2D image encoder를 사용하고 3D decoder를 대신 사용함
- SAMMed: 2D point로부터 3D prompt를 생성함
- 3DSAM-Adapter, MA-SAM: SAM의 component를 3D convolution을 변환, prompt encoder에 3D adapter 적용
- MSA: SAM의 weight는 유지한 채 space, depth adapter를 적용해 3D spatial information을 활용함, 그러나 정해진 특정 parameter로만 학습이 가능하기 때문에 fine-tuning에 한계가 있음
3. Large-Scale Volumetric Medical Dataset
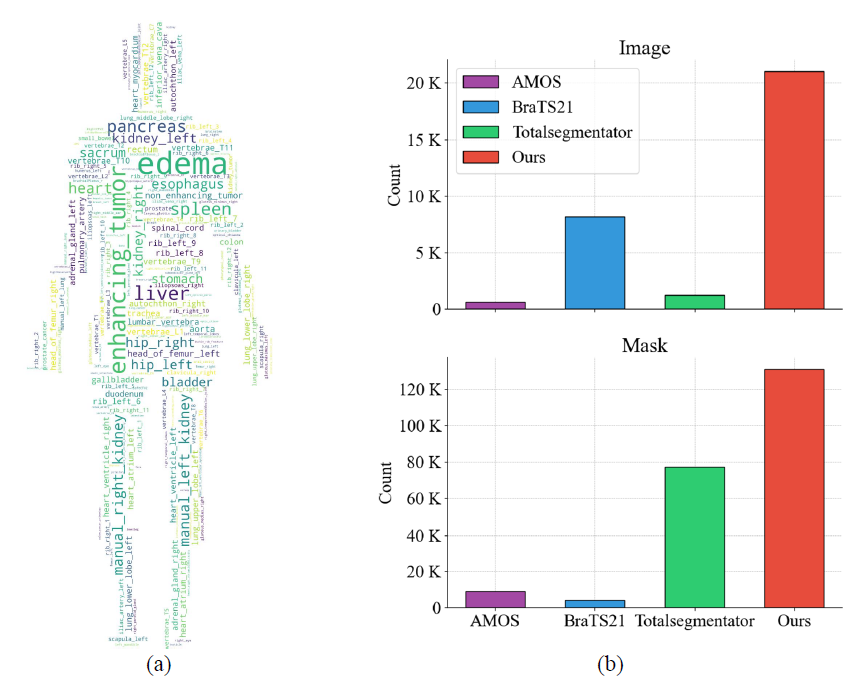
학습을 위한 3D medical image dataset으로 21000개의 medical image와 이에 대응되는 131000개의 mask를 준비하였으며 현재 가장 큰 volumetric medical image segmentation dataset일 것이다.
평가를 위해서 13개의 public benchmark dataset을 사용했다. MICCAI 2023 challenge에서 2개의 dataset을 추가했고 이는 thorax, abdominal organ, brain, bone 등이 포함된다. 또한 CT, US, 8개의 MRI sequence를 포함한 다양한 volumetric modality가 사용이 되었다. 그리고 다양한 범주에 걸쳐 153개의 target이 있다.
4. Method
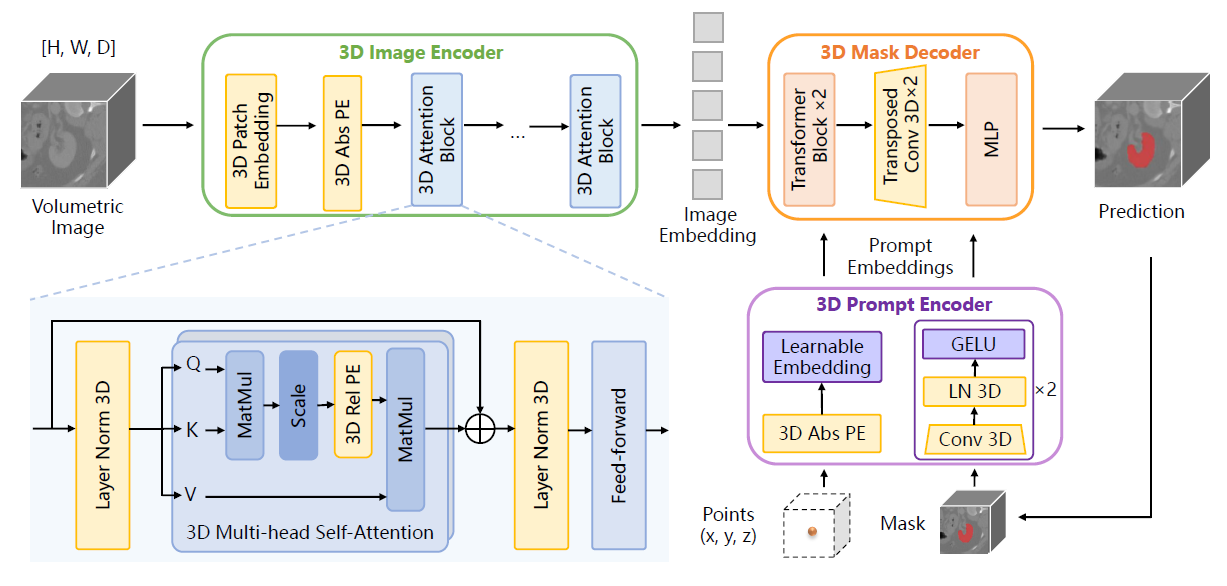
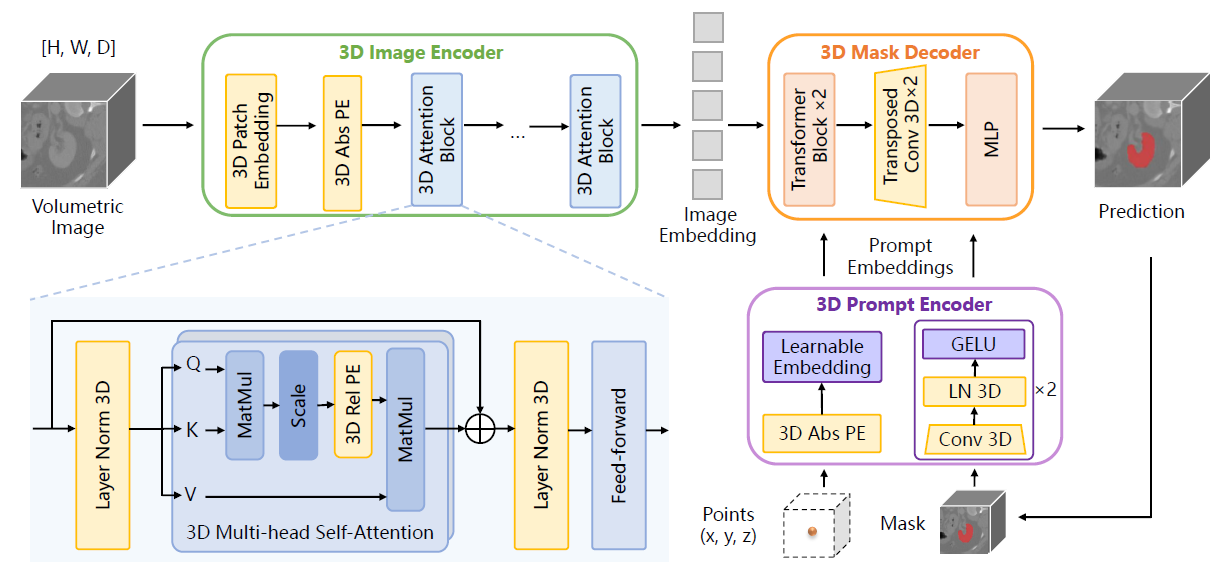
본 논문의 목적은 기존 SAM의 2D components를 수정함으로 volumetric medical images에 맞추는 것이다. SAM-Med3D는 3D spatial 정보를 활용하기 위해 전체 structure를 3D로 구성하였다.
3D Image Encoder는 patch 기반으로 3D convolution layer (16, 16, 16)와 Positional Encoding (PE)으로 embedding을 얻는다. 그리고 embedding patch들은 3D attention block에 입력된다. 그리고 Multi-Head Self-Attention (MHSA) module에 3D relative PE를 incorporate 함으로 spatial 정보의 detail을 학습할 수 있도록 했다. Prompt encoder에서 sparse prompt는 3D spatial nuance를 represent 하는 반면, dense prompt는 3D convolution으로 representation을 얻는다. 3D Mask Decoder는 3D transposed convolution으로 up-scaling을 수행한다.
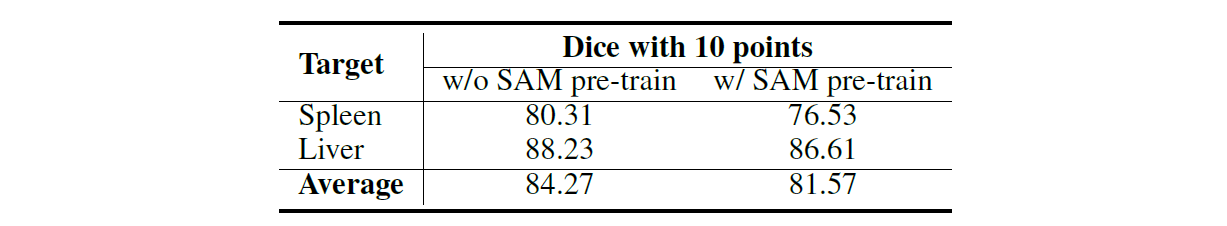
2D natural image와 3D medical image는 domain gap이 상당히 크기 때문에 SAM의 pre-trained weights를 사용하는 것의 영향을 비교하기 위해 실험을 하였다. 적용하는 방법은 2D convolution kernel을 복제해 3D kernel로 쌓았다. 위 표를 보면 알 수 있듯이 SAM pre-trained weights는 성능의 향상에 도움을 주지 못한다.
5. Experiments
Pytorch로 구현 되었으며 NVIDIA Tesla A100 GPU 8대로 학습하였다. Optimizer는 Adam을 사용하였으며 learning rate는 8e-4이다. Epoch은 800 epoch이다. Learning rate는 120, 160, 190 epoch에 1/10 씩 낮췄으며 200 epoch에 초기 값으로 복원했다. 모든 image는 crop 또는 padding을 적용해 128x128x128 shape으로 맞췄다. loss function으로는 DiceCELoss를 사용했다. Batch size는 12이며 20 step마다 gradient accumulation을 수행했으며 weight decay는 0.1이다. Augmentation은 RandomFlip과 ZNormalization만 적용을 했다.
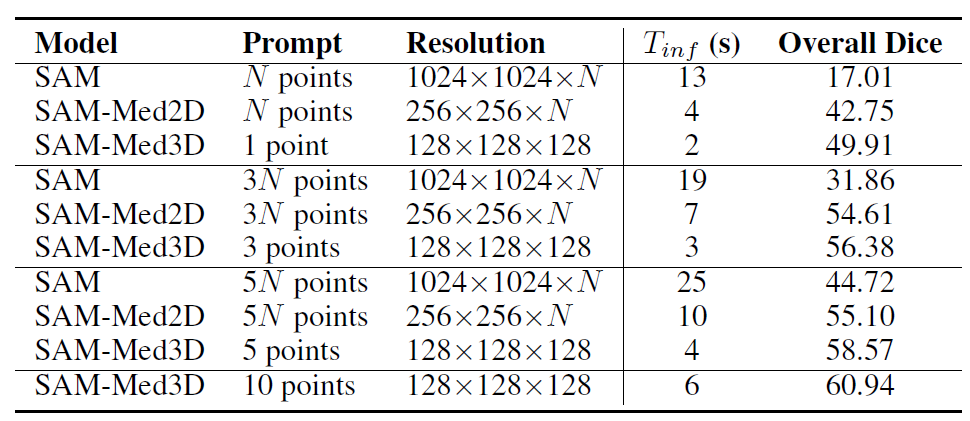
위 표는 SAM, SAM-Med2D, SAM-Med3D의 성능을 비교하고 있다. SAM-Med3D가 입력 prompt point 개수가 적음에도 가장 좋은 성능을 보이고 있다.
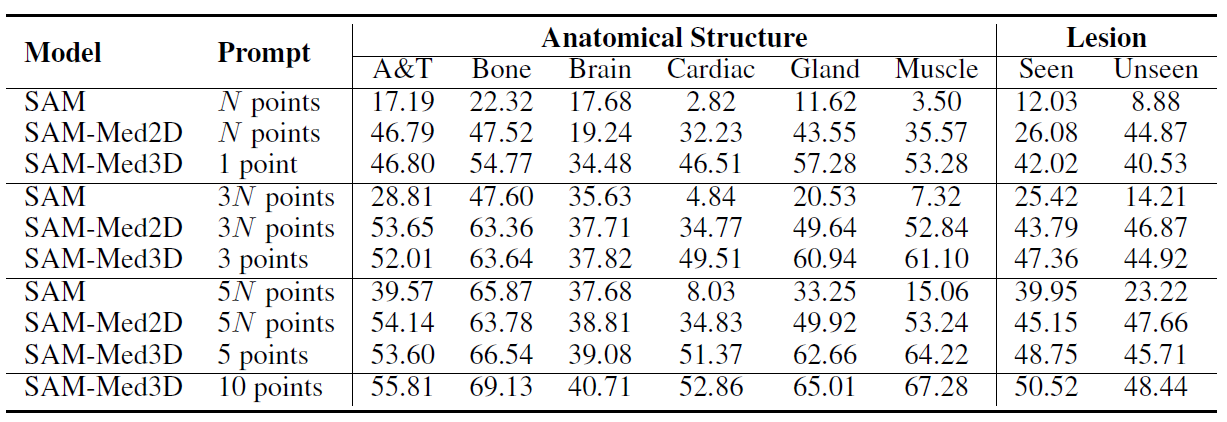
다양한 organ과 anatomical structure에 대한 검증도 하였다. 모든 anatomical structure에서 SAM-Med3D가 가장 좋은 성능을 보이고 있다.
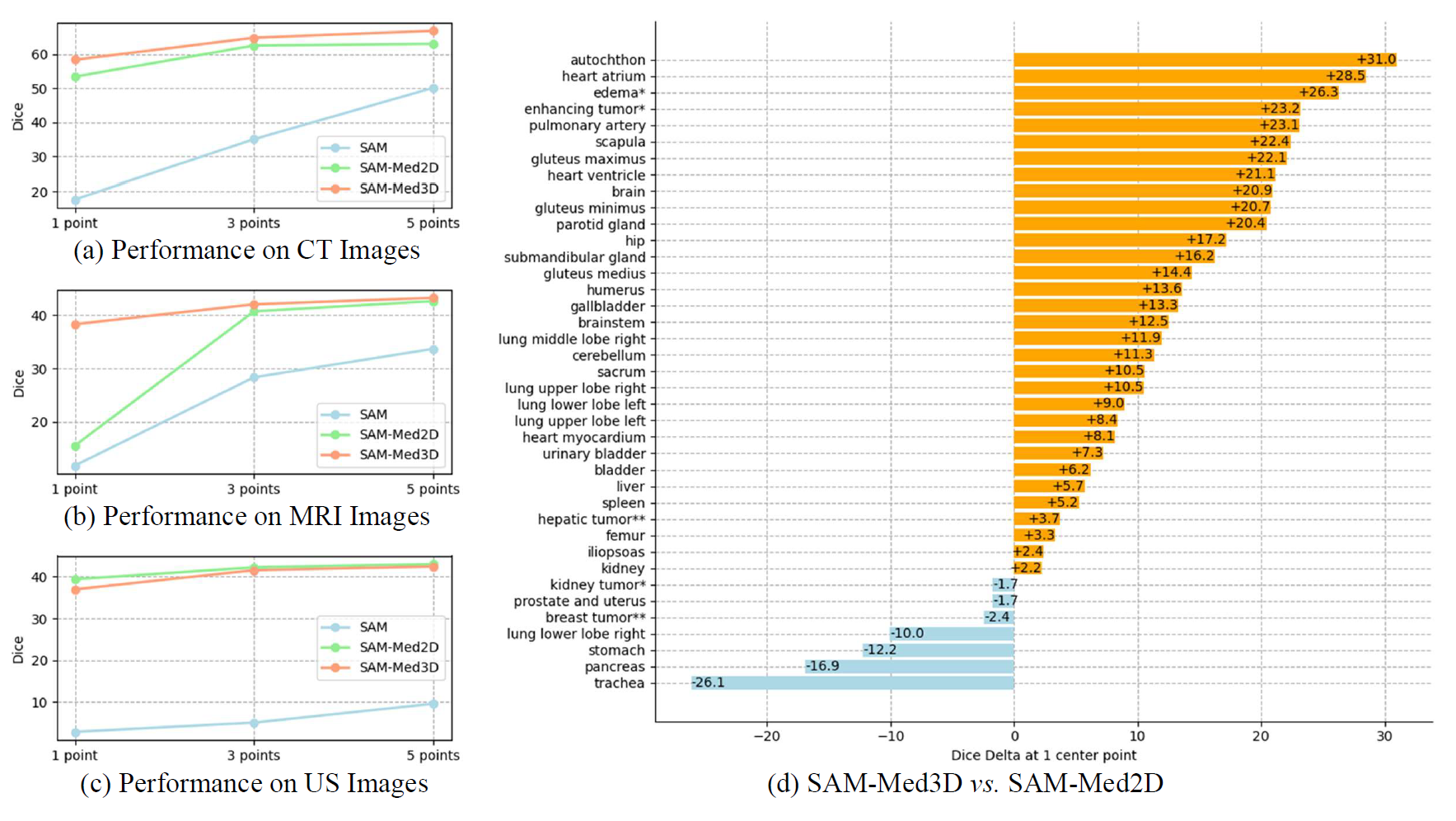
CT, MR, US 세 가지 modality 마다 성능도 prompt point 개수에 따라 비교를 하였다. SAM-Med3D와 SAM-Med2D의 성능이 큰 차이가 나지는 않지만 적은 prompt point 개수에서도 SAM-Med3D는 좋은 성능을 보였다. 특히 단일 prompt point일 때 MRI에서 SAM-Med2D의 성능이 현저히 낮았다.
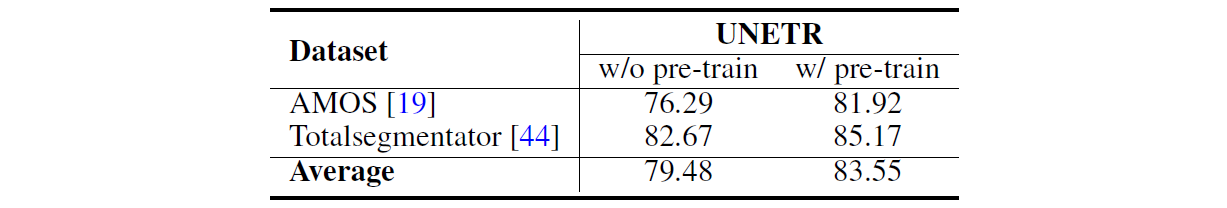
위 표는 UNETR 모델에서 SAM-Med3D의 pre-trained ViT encoder를 사용해 pre-training을 수행한 유무에 따른 성능을 비교한 결과이다. 결과적으로 SAM-Med3D는 3D 의료 영상 분석을 위한 foundation model로 자리 잡을 수 있을 만한 potential이 있다.
6. Conclusion
본 논문에서는 volumetric medical image를 위한 3D SAM model인 SAM-Med3D를 제안하였다. Large-scale 3D medical image dataset을 scratch로 학습을 했으며 3D structure로 모델을 재구성 하였다. Volume당 1 prompt point가 입력될 때를 기준으로 SAM보다 32.90%의 성능 향상이 있었다. 또한 SAM-Med3D는 transferability도 우수하며 3D medical image task에서 foundation model로 자리를 잡을 potential이 있다. 추후에는 3D context에 더 적합한 3D prompt form을 찾는 것과 좀 더 적합한 training strategy를 찾는 과제가 남아 있다.