
본 포스팅은 제가 읽었던 논문을 간단하게 정리하는 글입니다. 논문의 모든 내용을 작성하는 것이 아닌, 일부분만 담겨 있으므로 자세한 내용은 원본 논문을 확인해 주시기를 바랍니다. 또한, 논문을 잘못 이해한 부분이 있을 수 있으므로, 양해 바랍니다.
Overview
Segmentation을 위한 foundation model SAM(Segment Anything)은 medical specific knowledge의 부족으로 인해 medical image segmentation의 성능이 저조한 것으로 알려져 있음.
Medical image의 경우, low image contrast, ambiguous tissue boundaries, tiny lesion regions 등 다양한 문제가 존재함. Medical 분야에 맞도록 MedSAM 등 SAM을 medical image에 fully fine-tuning 한 기존의 연구가 존재하나, full fine-tuning은 시간 및 하드웨어 리소스 등에서 매우 큰 비용이 필요함.
본 논문에서는 medical 분야에서의 segmentation 성능을 높이기 위해 fine-tuning 대신 adapter 기반의 Medical SAM Adapter(Med-SA)를 제안함. 해당 방법을 통해 medical specific knowledge를 모델에 전달할 수 있음.
Adapter의 주 아이디어는 large pre-trained model의 가중치는 동결하고, 훈련할 수 있는 파라미터를 조금 더 추가한 뒤 해당 파라미터를 통하여 domain-specific knowledge를 학습하는 것임. 이러한 Adaption은 매우 효율적이나, Medical 분야에 바로 적용하기에는 문제가 있을 수 있음.
대표적인 문제 예시로 3D 이미지가 있음. CT, MRI 등의 이미지는 3D로 저장되기 때문에, 2D SAM 모델을 3D로 적용하기 위해서는 추가적인 처리가 필요함.
본 논문에서는 medical 분야에 적합한 Med-SA와 함께, 3D data를 처리할 수 있는 Space-Depth Transpose(SD-Trans), 사용자가 제공한 prompt의 중요성을 고려하는 Hyper-Prompting Adapter(HyP-Adpt)를 제안함.
Method
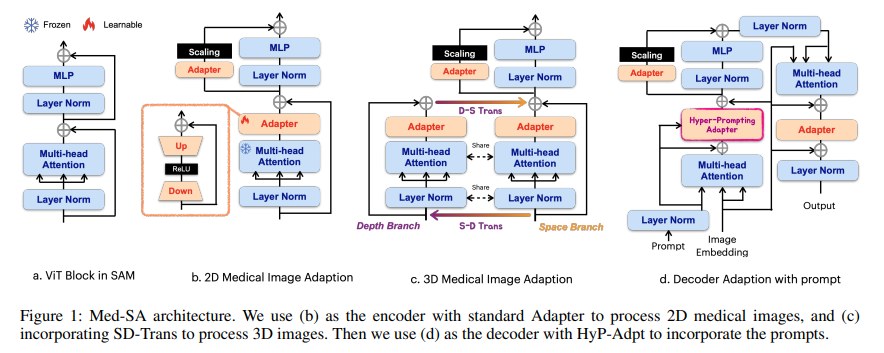
아래의 Figure 1을 통해 본 논문에서 제안하는 아키텍처를 확인할 수 있음.
Preliminary: SAM architecture
SAM은 image encoder, prompt encoder, mask decoder로 구성됨. 이미지 인코더는 MAE로 pre-trained 된 ViT를 사용하며, 본 논문에서는 ViT-H/16 variant를 사용하였음.
Prompt encoder의 경우 sparse(points, boxes) prompt와 dense(mask) prompt를 받을 수 있는데, 본 논문에서는 sparse prompt만 고려하였음.
Med-SA architecture
Pre-trained SAM의 파라미터는 변경하지 않고, Transformer 모듈 중간 사이사이에 adapter를 추가하여 해당 adapter를 훈련시킴.
Adapter의 경우 down-projection, ReLU activation, up-projection으로 구성되며, 각 projection은 MLP를 통해 수행됨.
SAM 인코더의 경우 각 ViT block에 두 개의 adapter를 추가하였음. 해당 위치는 Figure 1의 (b)에서 확인할 수 있음. MLP 출력과 연결되는 두 번째 adapter의 경우 embedding을 scaling 하며, 이때 scale factor 를 사용함.
SAM 디코더의 경우 각 ViT block에 세 개의 adapter를 추가하였음. 그중 하나는 Hyper-Prompting Adapter로, 아래에서 설명함.
SD-Trans
MRI, CT 등과 같은 여러 개의 slice를 가지는 3D 이미지로 저장됨. 각 Slice마다의 correlation을 이해하는 것은 정확한 의사 결정에 있어서 필수적임.
Figure 1의 (c)에서 3D 이미지를 위한 adaption을 확인할 수 있음. Attention operation을 두 갈래로 나누어 처리함. 깊이가 인 3D 이미지는 크기의 벡터로 임베딩 됨. 은 임베딩 벡터의 개수, 은 임베딩 벡터의 차원을 의미함.
Space branch의 경우 기존의 attention과 동일하게 각 patch 사이의 관계를 학습함. Depth branch의 경우, 입력 데이터를 로 변환하여 깊이와 임베딩 벡터 사이의 interaction을 학습함. 이러한 depth branch를 사용하여 3D 이미지의 depth information을 학습할 수 있음.
HyP-Adpt
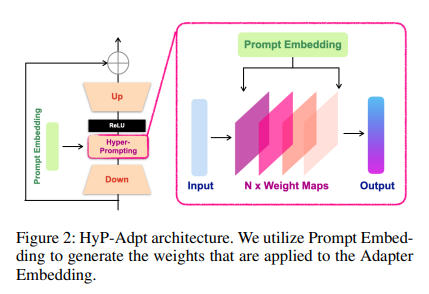
Source task의 interactive behavior와 downstream task에서의 interactive behavior는 서로 다를 수 있음. 따라서, downstream task에서의 성능을 보다 높일 수 있도록 visual prompt 정보를 adapter에 추가하고자 함.
HyP-Adpt에서는 간단한 projection과 reshaping 연산으로 prompt embedding에 대한 weight map을 생성함.
Hyper-prompting은 adapter 의 reduced embedding에 적용됨. 사용자의 클릭 위치, box 위치와 같은 prompt 정보는 로 축소되며, 다음의 연산을 통해 sequence of weights를 생성함.
는 reshape 연산을 의미하고, 은 을 으로 변환시키는 MLP layer를 의미함. 은 의 차원과 동일하고, 은 출력의 target length와 동일함.
Reshape 연산을 통해 1D embedding 는 2D weight 으로 변환되고, 해당 2D 가중치는 에 다음과 같이 적용됨.
Training Strategy
Click prompt와 bounding box prompt를 고려함. Bbox prompt 생성은 origial SAM과 동일한 방법을 사용하고, click prompt generation은 새로운 방법을 사용함.
Click prompt generation의 핵심은 positive click은 foreground region을, negative click은 background region을 의미한다는 것임. Random & iterative click sampling 기법을 통해 모델을 훈련할 prompt를 생성하였음.
Experiments
Dataset
모델의 일반적인 segmentation 성능을 확인할 수 있도록 abdominal multi-organ segmentation을 위한 BTCV dataset을 사용함.
추가로, 다른 modalities에서의 성능 또한 확인할 수 있도록 아래의 4가지 task에서도 실험하였음.
- optic disc and optic cup segmentation over fundus images
- brain tumor segmentation over brain MRI images
- thyroid nodule segmentation over ultrasound images
- melanoma or nevus segmentation from dermoscopic images
Implementation Details
4가지의 prompt setting을 비교함. “1-point”는 임의의 positive point 1개, “3-points”는 임의의 positive point 3개, “BBox 0.5”는 target과 50% 정도 겹쳐지는 bbox, “BBox 0.75”는 target과 75% 정도 겹쳐지는 bbox를 의미함.
훈련 에포크 및 하드웨어 등은 논문 참고.
Comparing Results
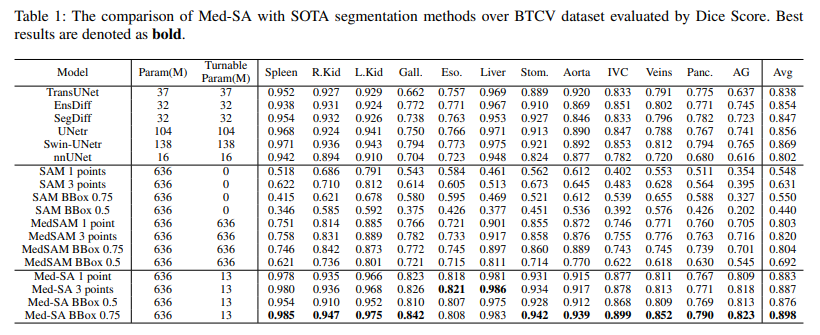
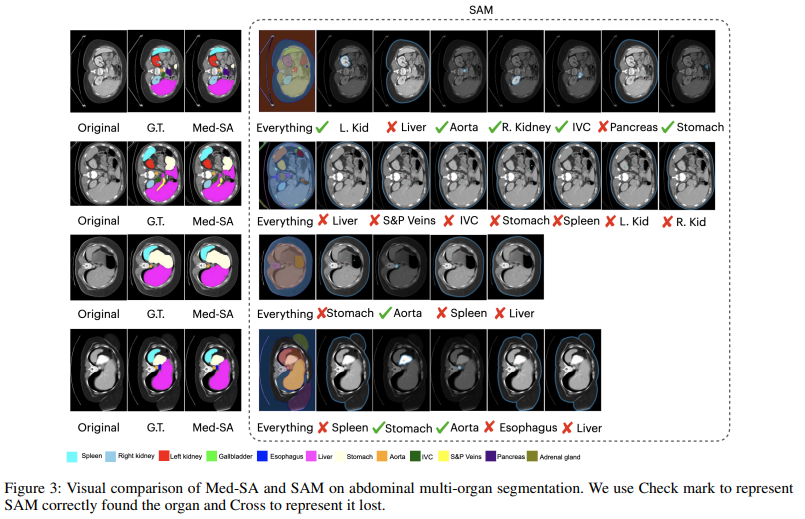
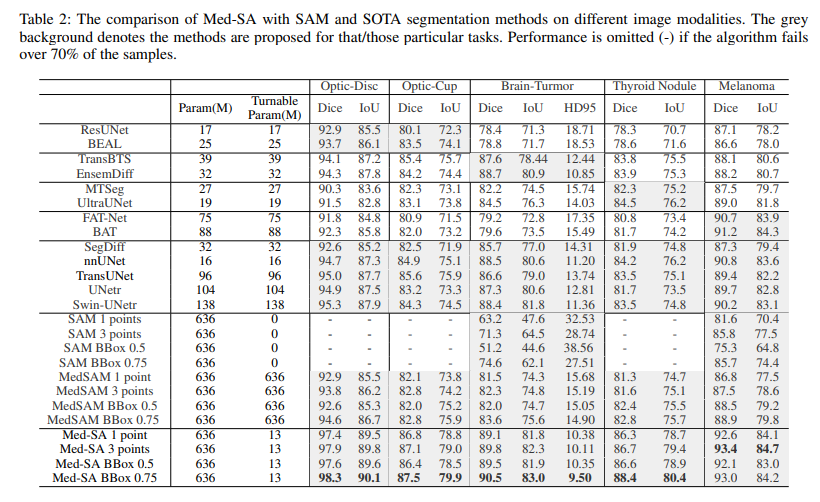
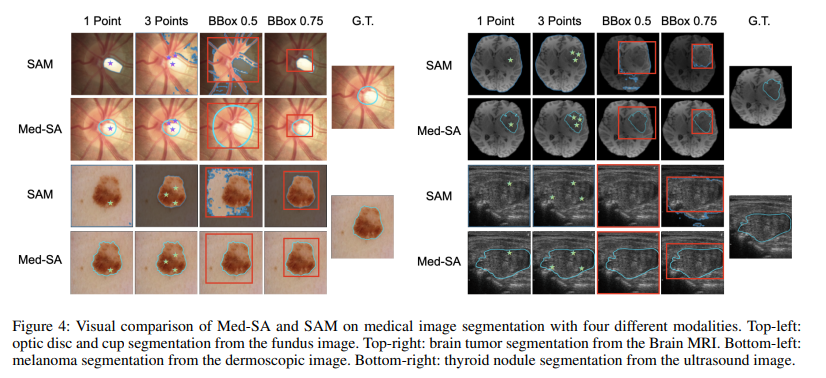
실험을 통해, Med-SA가 다른 segmentation method에 비해 더 높은 성능을 달성함을 확인하였음. 추가로, 매우 적은 수인 13M 개의 파라미터 학습을 통해 fully fine-tuned MedSAM 모델보다 더 높은 성능을 얻음.
Ablation Study 등 추가적인 실험 결과는 논문을 참고.
