AI_Tech부스트캠프 week13...[6] High Performance를 자랑하는 U-Net 계열의 모델들 U-Net, U-Net++, U-Net 3+
AI_tech_CV트랙 여정
1. U-Net
1.1 U-Net Intro
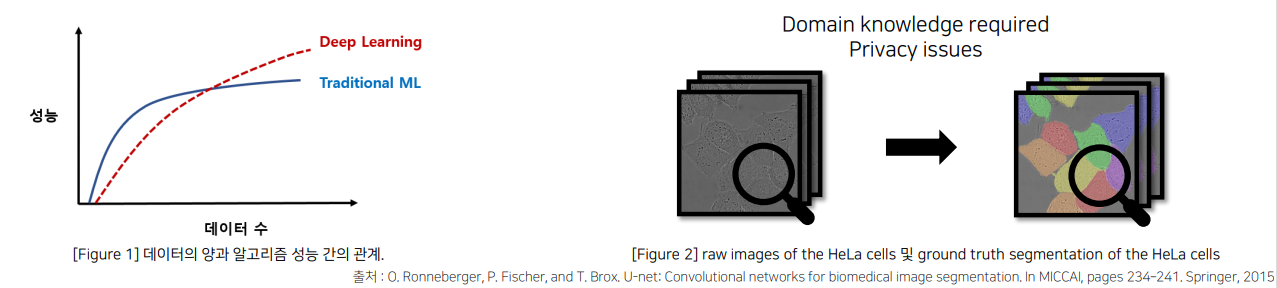
의료 계열에서의 문제 상황 I (데이터 부족)
• 기본적인 Deep Learning은 파라미터 수가 많고 네트워크가 깊어서 train data가 많이 필요
• 특히, biomedical의 특성상 data의 수가 많이 부족할 수 밖에 없음
• 이유 : 전문가에 의한 labeling 작업 및 환자 개인 정보 등의 문제로 원활한 train data 수집 어려움
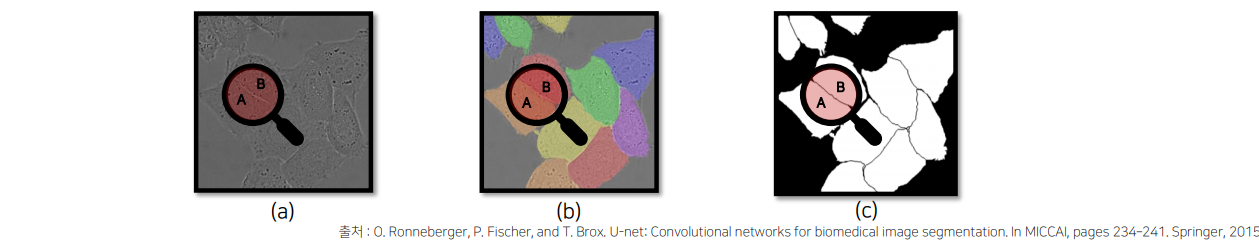
의료 계열에서의 문제 상황 II (같은 클래스가 인접한 셀 구분이 어려움)
특히, Cell Segmentation 작업의 경우, 같은 클래스가 인접해 있는 셀 사이의 경계 구분 필요
-> 아래 그림 (a)에서 “A” 와 “B”는 같은 클래스지만, (b)처럼 서로 다른 인스턴스(셀)로 구분을 요구하며, 이를 구분하기 위해 (c) 처럼 인스턴스(셀)의 경계를 테두리로 만들어야 하는 작업 필요 그러나, 일반적인 “Semantic Segmentation” 작업에서는 불가능
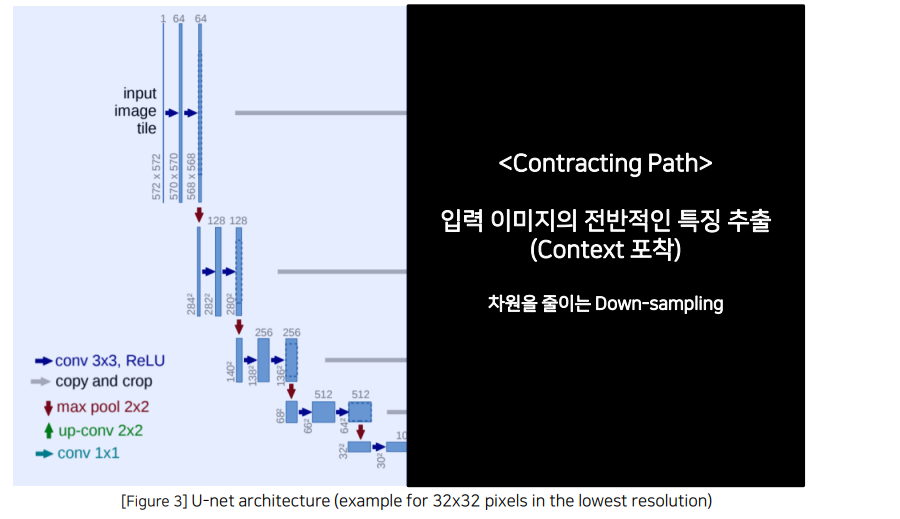
1.2 U-Net Architecture (구조)
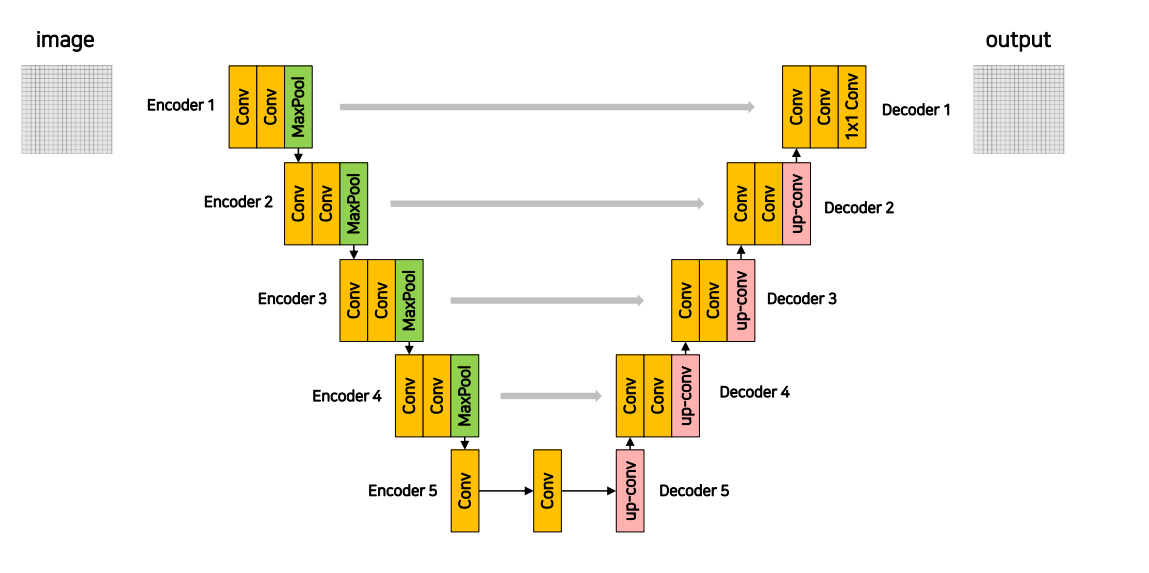
Contracting Path 와 Expanding Path 가 대칭인 U자 형을 이룸

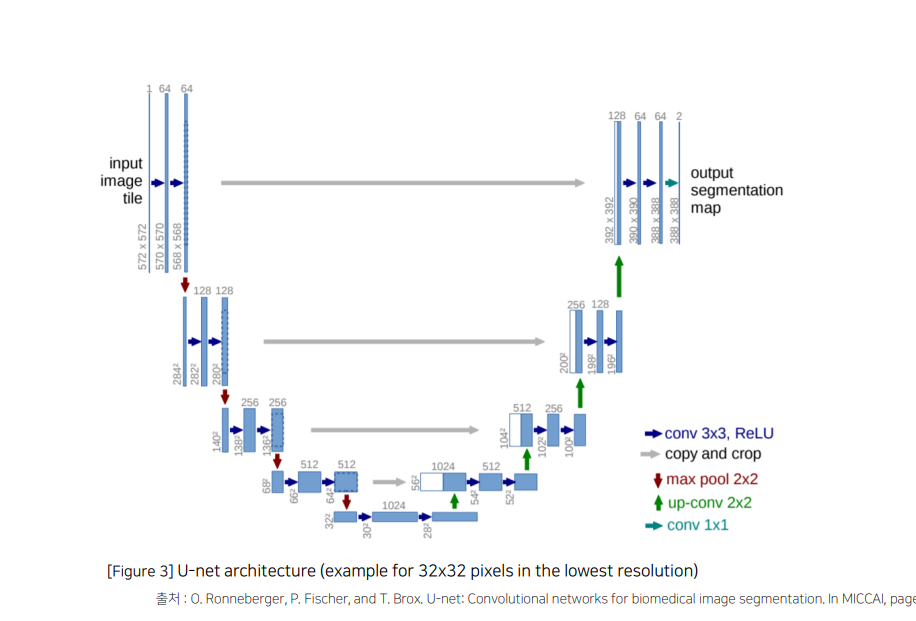
- 3x3 Conv – BN - ReLU가 2번씩 반복되는 구조
- Zero-Padding을 적용하지 않아서 특징맵의 크기가 감소
- Contracting Path에서는 채널의 수가 2배로 증가하고 Expanding Path에서는 채널의 수가 2배로 감소
- 같은 계층(Level)의 Encoder 출력물과 Up-Conv의 결과를 Concatenate하는 형태
Contracting Path : 이미지 특징 추출
- (3 x 3 convolution Network + BN + ReLU) X 2
- No zero-padding 으로 patch-size 감소
- 2 x 2 Max pooling (stride = 2)
- Feature Map의 크기가 절반으로 감소
- Max pooling 이후 채널 수 2배 증가
🤔No zero-padding 이면 특징맵(patch-size)의 크기가 감소하는 이유?
👉 Convolution 연산 시에 padding을 추가하여 입력과 동일한 크기의 출력을 얻을 수 있기 때문
ex) 입력이 크기라고 하고, Zero-padding 없이 커널을 사용하여 Convolution 연산을 하면, 특징맵의 크기가 로 줄어듭니다. 이를 반복하면 점차적으로 특징맵 크기(또는 patch size)가 줄어드는 것입니다.
Expanding Path : localization을 가능하게 함
- 2 x 2 Up-Conv 사용 (Transposed Convolution)
- Feature Map 크기가 2배 증가
- 채널의 수를 절반으로 감소 (1/2배 )
- Contracting 에서 얻은 Feature Map 과 Concat 진행
- 1 x 1 convolution 진행 (output channel은 class 수)
U-Net 네트워크의 Contribution
- Encoder가 확장함에 따라 채널의 수를 1024까지 증가시켜 좀 더 고차원에서 정보를 매핑
- 각기 다른 계층의 Encoder의 출력을 Decoder와 결합시켜서 이전 레이어의 정보를 효율적으로 활용
1.3 U-Net 에 적용된 Techniques
Data Augmentation
Random Elastic deformations을 통해서 Augmentation을 수행
-> model이 invariance와 robustness를 학습할 수 있도록 하는 방법
Pixel-wise loss weight를 계산하기위한 Weight map을 생성
같은 클래스를 가지는 인접한 셀을 분리하기 위해 해당 경계부분에 가중치를 제공
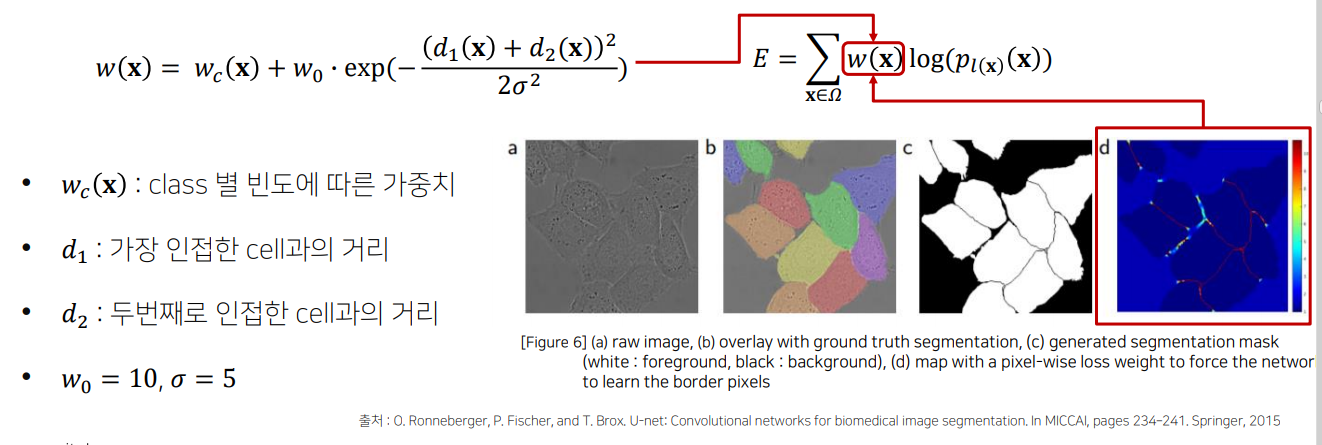
: 거리 값을 조절하는 파라미터
이 항이 큰 값을 갖게 되는 경우는, 두 개의 객체 경계 사이에 위치한 픽셀일 때입니다. 이 경우 𝑑1과 d2 가 작아서 지수 함수 값이 커지므로 해당 픽셀에 더 높은 가중치를 부여하게 됩니다. 이를 통해 네트워크가 경계에 있는 픽셀을 더 잘 학습하게 됨!
가만히 보면 크로스엔트로피랑 매우 유사하다.
크로스엔트로피 :
: 전체 손실 함수로, 각 픽셀의 손실을 가중합한 값입니다.
: 이미지 내의 모든 픽셀 위치 집합입니다.
: 위에서 정의한 가중치 맵으로, 특정 픽셀 위치 에서의 가중치를 나타냅니다.
: 에서 예측된 클래스 의 확률입니다. 네트워크가 특정 클래스에 대해 예측한 확률로, 픽셀별로 올바른 클래스를 예측할 확률 값을 갖습니다.
=>이 손실 함수는 각 픽셀의 손실에 𝑤(𝑥)가중치를 부여하여 경계에 있는 픽셀과 중요도가 높은 픽셀에 대한 학습을 강화합니다.
1.4 코드 실습
import torch
import torch.nn as nn
# CBR2d 정의
def CBR2d(in_channels, out_channels, kernel_size=3, stride=1, padding=1, bias=True):
return nn.Sequential(
nn.Conv2d(in_channels=in_channels,
out_channels=out_channels,
kernel_size=kernel_size,
stride=stride,
padding=padding,
bias=bias),
nn.BatchNorm2d(num_features=out_channels),
nn.ReLU()
)
# U-Net 네트워크의 일부로 인코더와 디코더 정의
class UNet(nn.Module):
def __init__(self):
super(UNet, self).__init__()
# Encoder 1
self.enc1_1 = CBR2d(1, 64, kernel_size=3, stride=1, padding=0, bias=True)
self.enc1_2 = CBR2d(64, 64, kernel_size=3, stride=1, padding=0, bias=True)
self.pool1 = nn.MaxPool2d(kernel_size=2)
# Encoder 2
self.enc2_1 = CBR2d(64, 128, kernel_size=3, stride=1, padding=0, bias=True)
self.enc2_2 = CBR2d(128, 128, kernel_size=3, stride=1, padding=0, bias=True)
self.pool2 = nn.MaxPool2d(kernel_size=2)
# Encoder 3
self.enc3_1 = CBR2d(128, 256, kernel_size=3, stride=1, padding=0, bias=True)
self.enc3_2 = CBR2d(256, 256, kernel_size=3, stride=1, padding=0, bias=True)
self.pool3 = nn.MaxPool2d(kernel_size=2)
# Encoder 4
self.enc4_1 = CBR2d(256, 512, kernel_size=3, stride=1, padding=0, bias=True)
self.enc4_2 = CBR2d(512, 512, kernel_size=3, stride=1, padding=0, bias=True)
self.pool4 = nn.MaxPool2d(kernel_size=2)
# Encoder 5 and Decoder 5
self.enc5_1 = CBR2d(512, 1024, kernel_size=3, stride=1, padding=0, bias=True)
self.enc5_2 = CBR2d(1024, 1024, kernel_size=3, stride=1, padding=0, bias=True)
self.upconv4 = nn.ConvTranspose2d(1024, 512, kernel_size=2, stride=2, padding=0, bias=True)
# Decoder 4
self.dec4_2 = CBR2d(1024, 512, kernel_size=3, stride=1, padding=0, bias=True)
self.dec4_1 = CBR2d(512, 512, kernel_size=3, stride=1, padding=0, bias=True)
self.upconv3 = nn.ConvTranspose2d(512, 256, kernel_size=2, stride=2, padding=0, bias=True)
# Decoder 3
self.dec3_2 = CBR2d(512, 256, kernel_size=3, stride=1, padding=0, bias=True)
self.dec3_1 = CBR2d(256, 256, kernel_size=3, stride=1, padding=0, bias=True)
self.upconv2 = nn.ConvTranspose2d(256, 128, kernel_size=2, stride=2, padding=0, bias=True)
# Decoder 2
self.dec2_2 = CBR2d(256, 128, kernel_size=3, stride=1, padding=0, bias=True)
self.dec2_1 = CBR2d(128, 128, kernel_size=3, stride=1, padding=0, bias=True)
self.upconv1 = nn.ConvTranspose2d(128, 64, kernel_size=2, stride=2, padding=0, bias=True)
# Decoder 1
self.dec1_2 = CBR2d(128, 64, kernel_size=3, stride=1, padding=0, bias=True)
self.dec1_1 = CBR2d(64, 64, kernel_size=3, stride=1, padding=0, bias=True)
# Output Segmentation map
self.score_fr = nn.Conv2d(in_channels=64, out_channels=2, kernel_size=1, stride=1, padding=0, bias=True)
# 이미지 크기 맞추기
def crop_img(self, in_tensor, out_size):
dim1, dim2 = in_tensor.size()[2:]
out_tensor = in_tensor[:, :,
int((dim1 - out_size) / 2): int((dim1 + out_size) / 2),
int((dim2 - out_size) / 2): int((dim2 + out_size) / 2)]
return out_tensor
def forward(self, x):
# Encoder 1
enc1_1 = self.enc1_1(x)
enc1_2 = self.enc1_2(enc1_1)
pool1 = self.pool1(enc1_2)
# Encoder 2
enc2_1 = self.enc2_1(pool1)
enc2_2 = self.enc2_2(enc2_1)
pool2 = self.pool2(enc2_2)
# Encoder 3
enc3_1 = self.enc3_1(pool2)
enc3_2 = self.enc3_2(enc3_1)
pool3 = self.pool3(enc3_2)
# Encoder 4
enc4_1 = self.enc4_1(pool3)
enc4_2 = self.enc4_2(enc4_1)
pool4 = self.pool4(enc4_2)
# Encoder 5
enc5_1 = self.enc5_1(pool4)
enc5_2 = self.enc5_2(enc5_1)
upconv4 = self.upconv4(enc5_2)
# Decoder 4
crop_enc4_2 = self.crop_img(enc4_2, upconv4.size()[2])
cat4 = torch.cat([upconv4, crop_enc4_2], dim=1)
dec4_2 = self.dec4_2(cat4)
dec4_1 = self.dec4_1(dec4_2)
upconv3 = self.upconv3(dec4_1)
# Decoder 3
crop_enc3_2 = self.crop_img(enc3_2, upconv3.size()[2])
cat3 = torch.cat([upconv3, crop_enc3_2], dim=1)
dec3_2 = self.dec3_2(cat3)
dec3_1 = self.dec3_1(dec3_2)
upconv2 = self.upconv2(dec3_1)
# Decoder 2
crop_enc2_2 = self.crop_img(enc2_2, upconv2.size()[2])
cat2 = torch.cat([upconv2, crop_enc2_2], dim=1)
dec2_2 = self.dec2_2(cat2)
dec2_1 = self.dec2_1(dec2_2)
upconv1 = self.upconv1(dec2_1)
# Decoder 1
crop_enc1_2 = self.crop_img(enc1_2, upconv1.size()[2])
cat1 = torch.cat([upconv1, crop_enc1_2], dim=1)
dec1_2 = self.dec1_2(cat1)
dec1_1 = self.dec1_1(dec1_2)
# Output
output = self.score_fr(dec1_1)
return output
# 모델 생성
model = UNet()
print(model)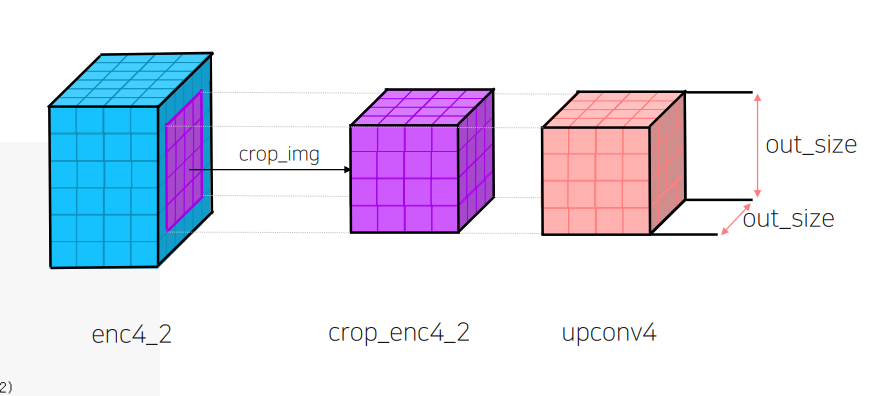
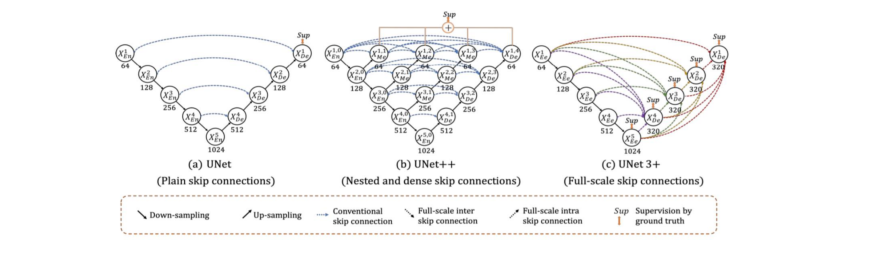
1.5 한계점
- U-Net은 기본적으로 깊이가 4로 고정됨
• 데이터셋마다 최고의 성능을 보장하지 못함
• 최적 깊이 탐색 비용 ↑ - 단순한 Skip Connection
• 동일한 깊이를 가지는 Encoder와 Decoder 만 연결되는 제한적인 구조
2. U-Net++
2.1 U-Net++ Intro
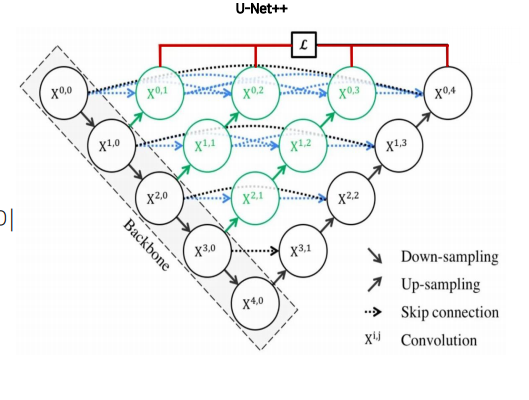
U-Net의 두 가지 한계점을 극복하기 위한 새로운 형태의 아키텍처 제시
- Encoder를 공유하는 다양한 깊이의 U-Net을 생성
- Encoder 𝑑𝑒𝑝𝑡ℎ = 1 ~ Encoder 𝑑𝑒𝑝𝑡ℎ = 4
2.2 Dense Skip Connection / Ensemble / Deep Supervision
Dense Skip Connection
- Skip Connection을 동일한 깊이에서의 Feature Maps이 모두 결합되도록 유연한 Feature Map 생성
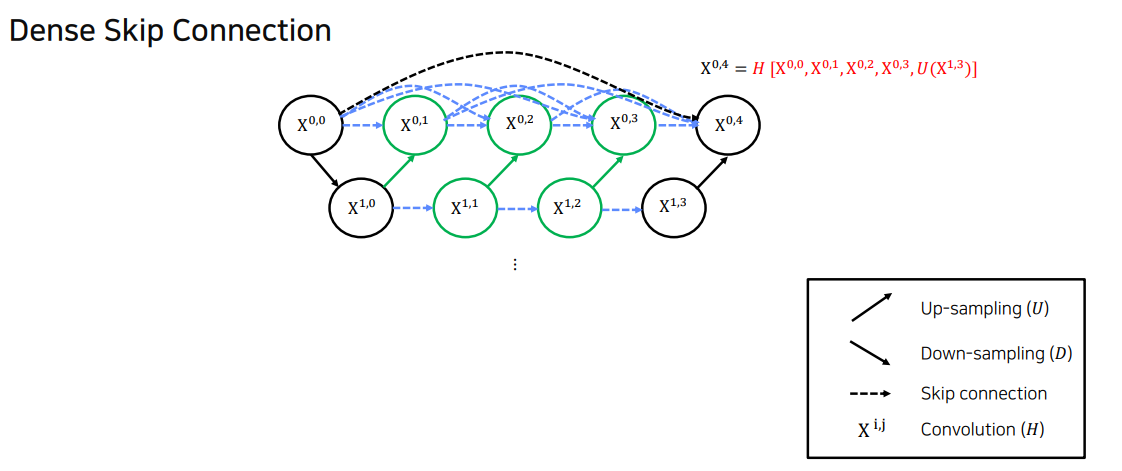
Concatnate를 함. 모든 곳에. 단순한 skip connection 극복
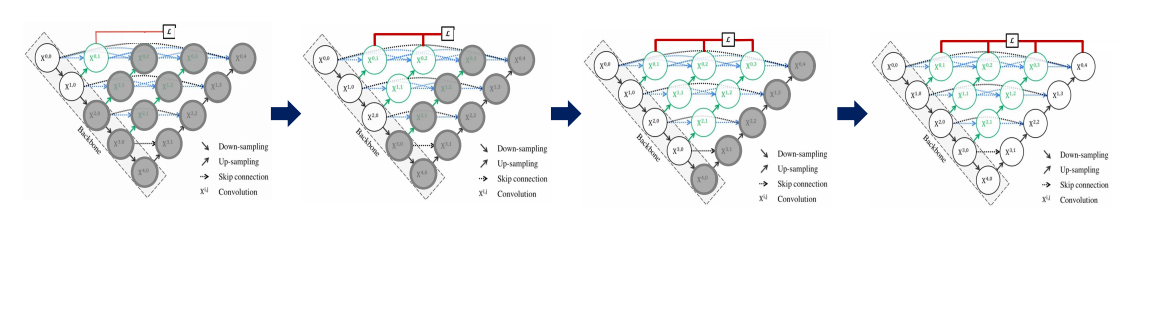
Ensemble
위 두가지 기법을 활용해서 ensemble 한 효과가 있는것임!
depth 1 + depth 2 + depth 3 + depth 4 인 모델들을 ensemble 하는 효과
Hybrid Loss
hybrid loss = Pixel wise Cross Entropy + Soft Dice Coefficient
이 수식은 하이브리드 손실 함수로, 크로스 엔트로피 손실과 다른 손실 항을 결합하여 네트워크가 특정 목표를 더 잘 학습하도록 설계
수식의 의미
- : 배치 크기 내의 픽셀 개수를 나타냅니다. 즉, 이미지에서의 각 픽셀에 대해 손실을 계산하고 평균화할 것입니다.
- : 클래스의 개수입니다. 예를 들어, 3개의 클래스가 있다면 C=3이 됩니다. 손실 함수는 모든 클래스에 대해 계산됩니다.
- : n에서 클래스 c의 타겟 레이블입니다. 이 값은 일반적으로 원-핫 인코딩된 형태로, 실제 클래스에 해당하는 위치에서는 1, 그 외의 위치에서는 0을 갖습니다.
- :n에서 클래스 c에 대해 예측된 확률 값입니다. 모델의 출력으로, 각 클래스에 속할 확률을 나타냅니다.
첫번째 항: 크로스엔트로피 손실
타겟 클래스와 예측 확률 간의 차이를 측정하며, 정확한 클래스를 예측할수록 손실이 낮아짐 -> 모델이 실제 타겟 클래스에 더 높은 확률로 예측하게끔 유도
두번째 항: 유사성 측정항
분자는 예측 값과 실제 값의 곱이고, 분모는 예측 값과 실제 값의 제곱합.
목적은 모델이 타겟 레이블에 가까운 값을 더 많이 예측하도록 유도!
👉 즉, 하이브리드 손실 함수는 크로스 엔트로피 손실을 기본으로 하면서, 두 번째 항을 추가하여 예측 값과 실제 값의 유사성을 더욱 강조합니다. 이를 통해 모델이 타겟 클래스뿐만 아니라 확률 분포 상에서 실제 값과 더 유사한 예측을 하도록 학습합니다
Deep Supervision
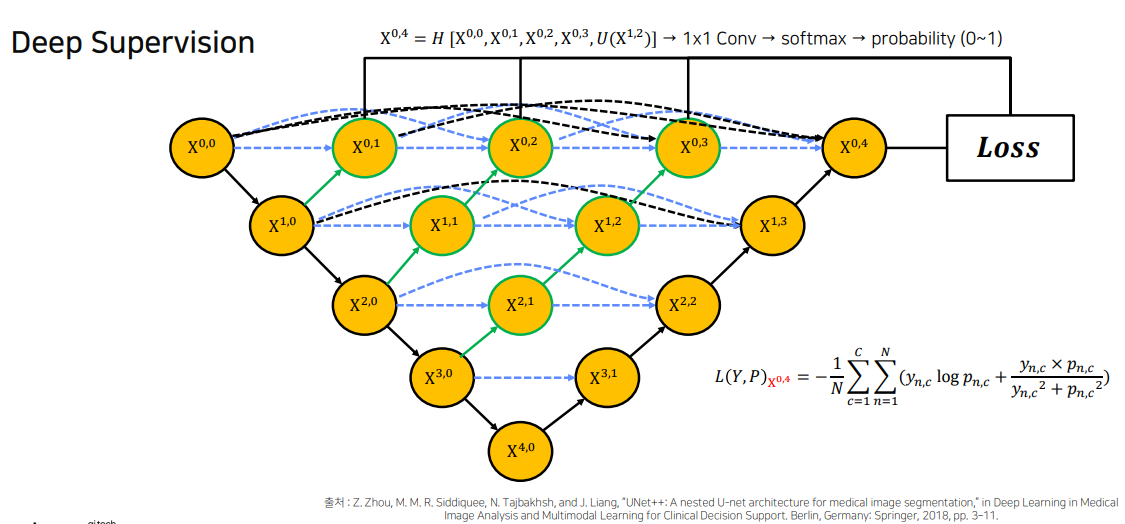
Deep Supervision은 네트워크의 중간 레이어에서도 손실(Loss)을 계산하여 학습하는 방식(다단계 손실함수를 계산함으로써 더 segmentic 하게 잘 하도록 함)
2.3 한계점
- 복잡한 Connection 으로 인한 Parameter ↑
- 많은 Connection으로 인한 Memory ↑
- Encoder-Decoder 사이에서의 Connection이 동일한 크기를 갖는 Feature map 에서만 진행됨
- 즉, Full Scale에서 충분한 정보를 탐색하지 못해 위치와 경계를 명시적으로 학습하지 못함
3. U-Net 3+
3.1 U-Net, U-Net++의 한계점
• U-Net에서의 decoder를 구성하는 방법은 같은 level의 encoder layer로 부터 feature map을 받는 simple skip connection 사용
• U-Net ++에서는 nested and dense skip connection을 사용하여 encoder–decoder 사이의 semantic gap을 줄임
-> 하지만 위에 parameter가 많고 메모리사용량이 높아지고 full scale에 명시적으로 학습을 못한다는 한계점이 있었음. 이걸 극복하기 위해서 U-Net 3+ 탄생
3.2 U-Net 3+ 에 적용된 Techniques
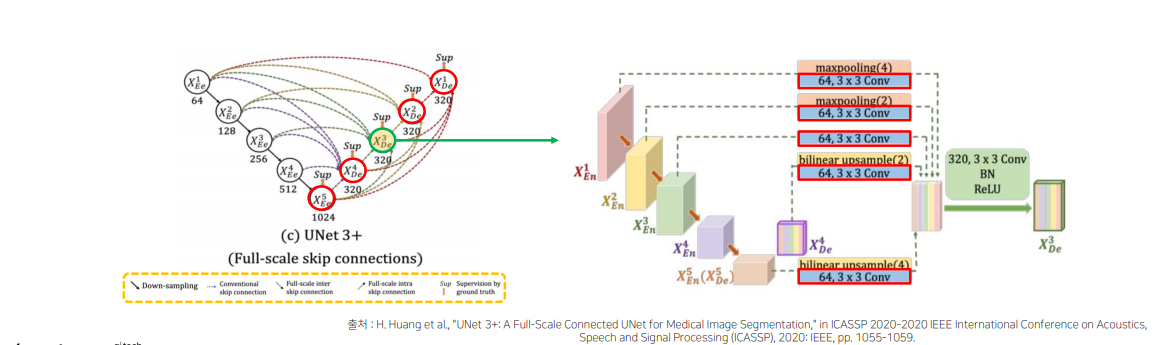
Full-scale Skip Connections : (conventional + inter + intra) skip connection
- decoder의 feature map 구성 방법
- Conventional: encoder layer로부터 same-scale의 feature maps 받음
- inter: encoder layer로부터 smaller-scale의 low-level feature maps 받음
- 풍부한 공간 정보를 통해 경계 강조
- intra: decoder layer로부터 larger-scale의 high-level feature maps 받음
- 어디에 위치하는지 위치 정보 구현
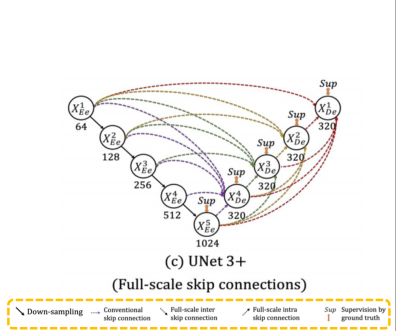
Full-scale Skip Connections with 64 Channel, 3x3 Convolutions
• Parameter를 줄이기 위해 모든 decoder layer의 channel 수 320 통일
• 모든 encoder layer 에서 skip connection 과정에서 64 channel, 3 x 3 conv 동일하게 적용
• U-Net, U-Net++, U-Net 3+ Architecture의 parameter 비교 (backbone : Vgg-16 / ResNet-101)
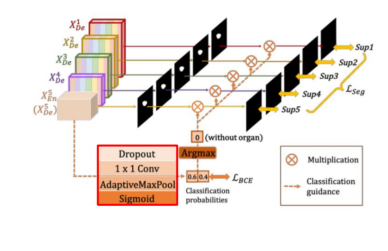
Classification-guided Module (CGM)
low-level layer에 남아있는 background의 noise발생하여, 많은 false-positive 문제 발생
• 정확도를 높이고자, extra classification task 진행
• high-level feature maps인 𝑋𝑑𝑒5 를 활용
- Dropout, 1x1 Conv, AdaptiveMaxPool, Sigmoid 통과
- Argmax를 통해 Organ이 없으면 0, 있으면 1으로 출력
- 위 에서 얻은 결과와 각 low-layer 마다 나온 결과를 곱
Full-scale Deep Supervision (loss function)
• 경계 부분 잘 학습하기 위해서 Loss 여러가지 결합

Loss 결합: 경계 부분을 잘 학습하기 위해 다양한 손실 함수를 결합하여 최종 손실 함수 𝐿seg 를 정의합니다
구성요소
①
Focal Loss로, 학습이 어려운 샘플에 더 큰 가중치를 부여하여 모델이 불균형 데이터에 대해 더 잘 학습할 수 있게 합니다
②
Multi-Scale Structural Similarity (MS-SSIM) 손실로, 구조적 유사성을 높이며 경계 부분의 디테일을 잘 반영할 수 있게함(boundary 인식 강화)
③
IoU (Intersection over Union) 손실로, 예측된 경계와 실제 경계의 겹치는 비율을 나타내며, IoU 값을 최대화하여 정확도를 높입니다(IoU : 픽셀의 분류 정확도를 상승)
3.3 결론
4. Another version of the U-Net
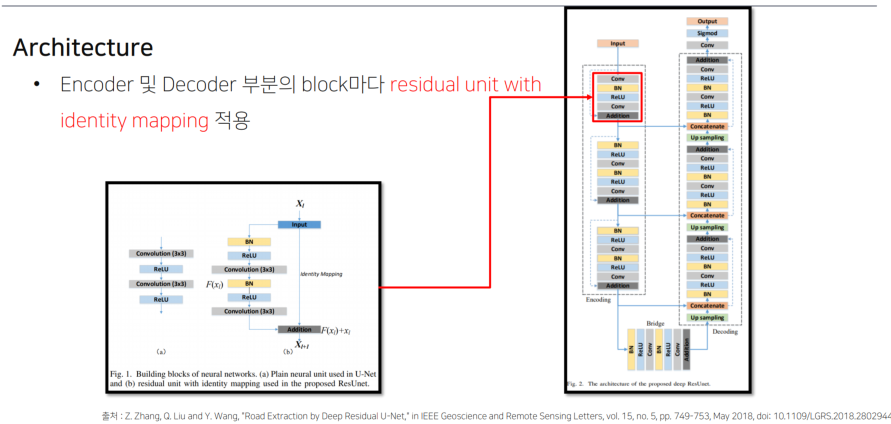
4.1 Residual U-Net
4.2 Mobile-UNet
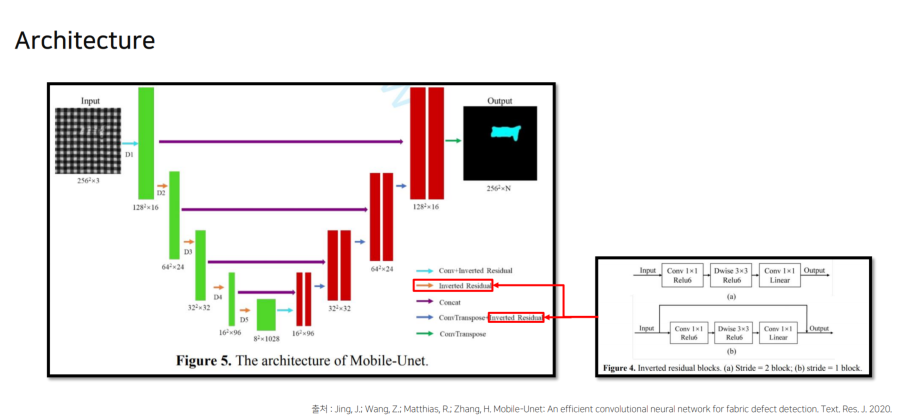
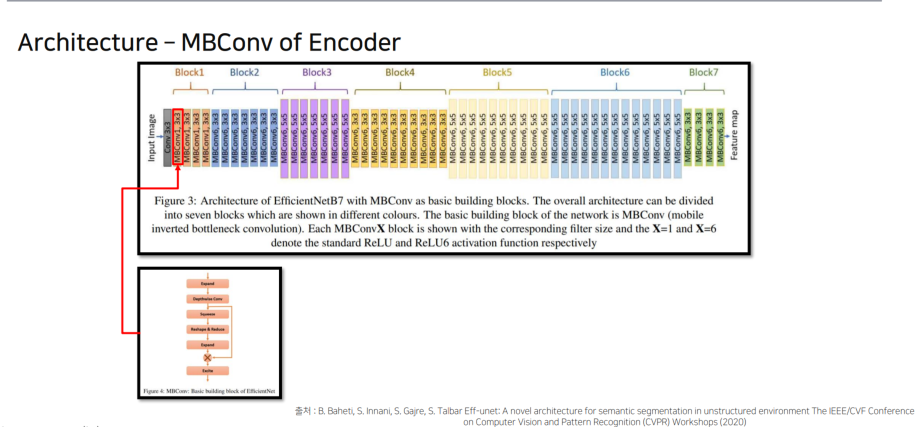
4.3 Eff-UNet
5. 결론
5.1 정리
6. 참고자료
U-Net: Convolutional Networks for Biomedical Image Segmentation
https://arxiv.org/abs/1505.04597
UNet++: Redesigning Skip Connections to Exploit
Multiscale Features in Image Segmentation
https://arxiv.org/pdf/1912.05074
UNet 3+: A Full-Scale Connected UNet for Medical Image Segmentation
https://arxiv.org/abs/2004.08790
Eff-UNet: A Novel Architecture for Semantic Segmentation in Unstructured Environment
https://openaccess.thecvf.com/content_CVPRW_2020/papers/w22/Baheti_Eff-UNet_A_Novel_Architecture_for_Semantic_Segmentation_in_Unstructured_Environment_CVPRW_2020_paper.pdf
The One Hundred Layers Tiramisu: Fully Convolutional DenseNets for Semantic Segmentation
https://arxiv.org/abs/1611.09326
Road Extraction by Deep Residual U-Net
https://arxiv.org/pdf/1711.10684
