교차분석
- 성별에 따라 생존자 숫자 (비율)이 다른가?
- pd.crosstab ( 변수1, 변수2, normalize=True or False) : normalize = 비율
titanic= sns.load_dataset("titanic")
pd.crosstab(titanic.sex, titanic.survived)
pd.crosstab(titanic.sex, titanic.survived, normalize=True)
# 시각화까지
ct=pd.crosstab(titanic.sex, titanic.survived)
ct.plot.bar(stacked=True) # seaborn lib
통계적 유의성(카이제곱)
- (= 모집단에서도 그럴 것인가 = 지금 샘플의 결과에서의 차이가 통계적으로 유의미한 차이인가)
pvalue < 0.05 이면 (남녀 생존율이) 통계적으로 유의미한 차이가 있다.
from scipy import stats
stats.chi2_contingency(ct) # 2번째 값이 p value
2번째 값이 p값T-test
- Nominal인 독립변수 값의 종류가 2가지 일 때
독립적 2 sample 비교
- 서로 다른 group의 값을 비교하는 경우 (예: 남 vs.여)
순서
- 우선, 분산 비교(F-test: 등분산성, 분산이 차이가 있는지 없는지 )를 해야 함
- levene ( sample1, sample2) : p-value < 0.05 이면, 통계적 유의성이 있게 분산의 차이가 있음
- ttest_ind ( sample1, sample2, equal_var=True of False)
male= titanic[titanic.sex=="male"]
female= titanic[titanic.sex=="female"]
levene(male.fare,female.fare)
#levene test에서 p값을 보자
ttest_ind(male.fare, female.fare, equal_var=False)
#equl_var가 다르니까 false. 이는 두 샘플의 분산이 같은지 여부
- p값 낮으면
성별에 따른 요금의 차이가 통계적 유의성이 있다. (=통계적으로 유의미한 수준의 차이가 있다)paired 2 sample 비교
- 같은 대상의 서로 다른 값을 비교하는 경우 (예: 약먹기전의 수치 vs. 약먹은 후의 수치)
- 2개 변수는 같은 종류 (예, 몸무게)
- 벡터1, 2의 길이가 같아야.. => 같은 값 비교
- NA 가 없어야 => 짝이 맞아야하니까
순서
ttest_rel ( 벡터1, 벡터2)
ttest_rel ( sl.height, sl.height2) #두개에 유의미한 차이가 있는지 보기. 변수 두개를 줘야함3 가지 이상의 값을 갖는 독립변수의 경우 => ttest를 못써. 이건 두개일때만
one way anova (분산분석)
- m=ols ('종속변수~독립변수', df).fit () : 모델 생성
- anova_lm (m) : 모델에 대한 정보 조회
from statsmodels.formula.api import ols
from statsmodels.stats.anova import anova_lm
model=ols('fare~pclass', titanic).fit()
print (anova_lm(model))
여기서는 p-값(PR(>F))은 0.529369. 0.05 보다 큼으로 유의미한 차이가 없다.상관분석(numeric-numeric)
- 상관관계: 같이 증가, 같이 감소 (선형적 상관관계)
- 독립변수: 종속 변수 같은 순서가 없음.
- 수치화된 계수+ 시각화로 안보면 상관관계 찾기 어려움
목표
- 상관도가 높은 변수 pair를 찾아내기
상관계수
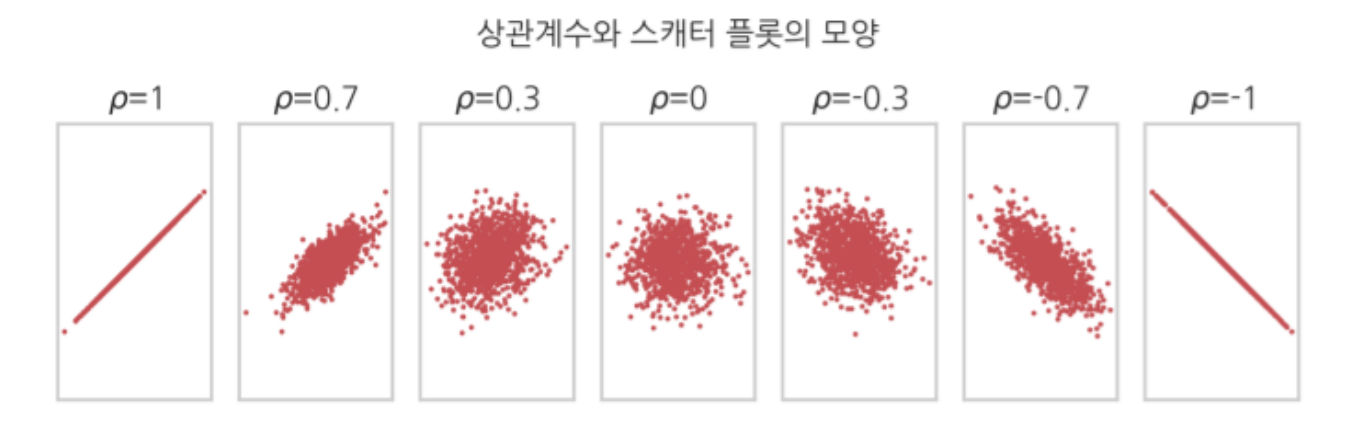
df.corr() 사용 <= 방향성이 없음 #순서가 없음
titanic[['age','fare']].corr()sns.pairplot(df, hue=키변수) , 키 변수 : 제3의 변수 추가 분석
- 그림 여러개 그리는 라이브러리
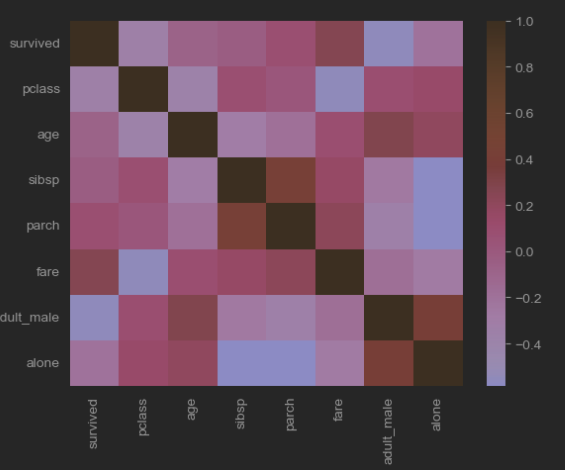
- 상관계수의 강도 보기 : heatmap(), 밝을 수록 높은 상관관계
T - 분포
- 샘플 사이즈가 작을때 쓰기 유리해. => 정규분포를 가성비 있게 만드는법.
카이스퀘어 분포
-
카이가 표준정규분포라고 할 때
-
카이 스퀘어 값의 크고 작음을 알려주는
-
분산의 분포
-
가설의 검증
Pearson & Spearman
Pearson 상관계수
이게 기본. 선형관계 (값의 크기 자체가 의미가 있는 numeric : 곱하기 나누기가 의미를 갖는 숫자)
-
수치형 변수 종류 (3가지)
- 순서만 의미있는 수치 : 학력 (고졸, 대졸, 대학원졸.. )
- +/- 가 가능한 수치 : 섭씨온도, 지능지수
- */ / 가 의미있는 수치 : 중량
-
범위는 -1에서 1까지
-
(-1)은 강한 음의 선형 관계를 나타내고, 1은 강한 양의 선형 관계를 나타내고, 0은 선형 관계가 없음
-
이상값에 민감하며 데이터가 정규 분포를 따른다고 가정한다.
Spearman 상관계수
값의 크기 대신 순위로 계산 (순서가 있는 nominal 데이터).
- 선형이든 비선형이든 두 변수 사이의 단조로운 관계를 측정합니다.
- 범위는 -1에서 1까지 동일.
- 데이터 순위를 기반으로 하며 이상치에 민감하지 않다.
- 데이터가 정상적으로 분포되어 있다고 가정하지 않습니다.
상관계수 주의사항
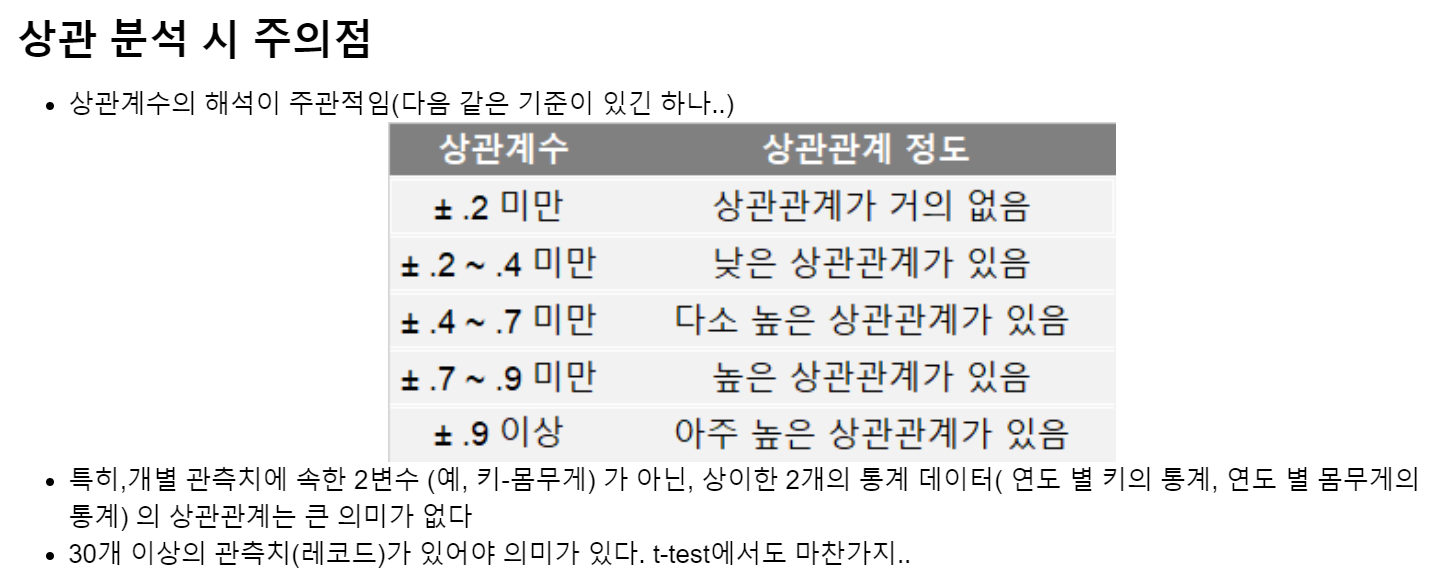
-
상이한 2개의 통계 데이터( 연도 별 키의 통계, 연도 별 몸무게의 통계) 의 상관관계는 큰 의미가 없다
-
30개 이상의 관측치(레코드)가 있어야 의미가 있다. t-test에서도 마찬가지..
회귀분석(numeric-numeric)
-
상관관계 : 2 변수가 같이 증가, 같이 감소 (선형적 상관관계, 직선에 얼마나 몰려있는지)
-
회귀분석 : 독립변수 -> 종속변수 선형적 상관관계의 정도(기울기)까지 분석
- 단순 선형회귀 : 하나의 독립변수 -> 하나의 종속변수
- 모델 생성 : 모델=ols( '종속변수~독립변수', data=데이터).fit()
- 모델 정보 조회 : 모델.summary(), 모델.params
from statsmodels.formula.api import ols, glm
m1 = ols ( 'quality~alcohol', data=wine).fit()
m1.summary()해석
- coef : 선형 모델 (1차함수 직선)의 기울기 (선형적으로 어느 정도 영향을 주었는지 분석)
- intercept: 절편
- y(종속변수)= coef(기울기)*x(독립변수) + intercept
예측
- 기울기와 절편이 데이터의 관계를 나타내는 하나의 모형이 됨
red_df= pd.read_csv("winequality-red.csv", sep=";")
m1 = ols ( 'quality~alcohol', data=wine[:3000]).fit() #3000개만 써서 모델을 만듬
m1.predict( wine[3000:].alcohol) # 3000개 이후의 데이터를 집어넣어서 예측(predict)중선형 회귀
-
독립변수가 여러개 일때 => 각각에 계수가 붙어
-
모델 생성 : 모델=ols( '종속변수~독립변수1+독립변수2+ ... ', data=데이터).fit()
-
각 독립변수의 영향이 출력됨
-
p < 0.05 작은 경우만 유의성을 가짐
wine.columns = [c.replace(' ', '_') for c in wine.columns]
wineForm = 'quality~fixed_acidity+volatile_acidity+citric_acid+residual_sugar+chlorides+free_sulfur_dioxide+total_sulfur_dioxide+density+pH+sulphates+alcohol'
m3 = ols(wineForm,data=wine[:5000]).fit()
prediction = m3.predict(wine[5000:])
