Practical Aspects of Deep Learning
Setting up your Machine Learning Application
Applied ML A highly iterative process
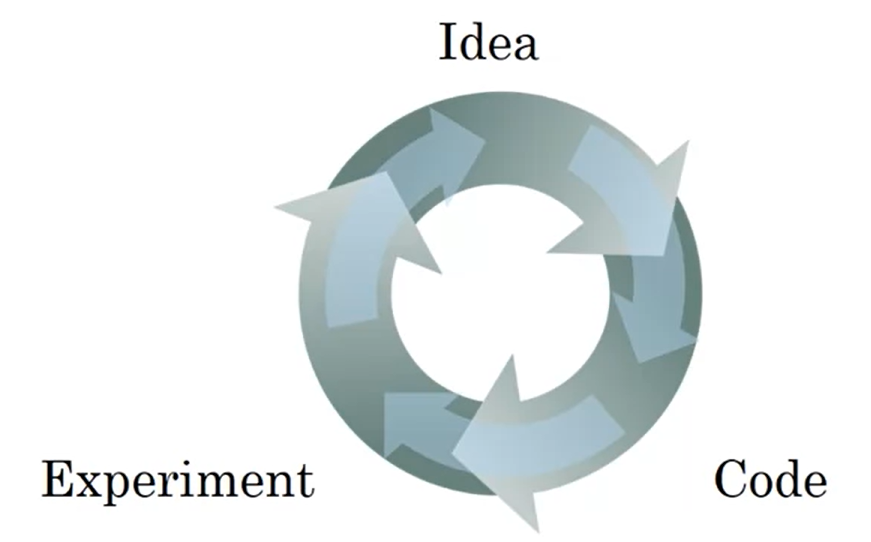
- 한 application이 다른 application에 곧바로 transfer 되는 것은 아니다.
- 각 분야에 맞는 things가 있고, iterative cycle을 통해 good choice를 찾게 되면 quick faster하게 만들 수 있다.
Train / Dev / Test sets
-
Previous era (traditional ML)
- train 70% / test 30% : all of them like 100-1000-10000
- train 60% / dev 20% / test 20%
-
Big data (in DL) : 1000000
- train 98% / dev 1% / test 1% : 1% is 100000
- train 99.5% / dev 0.4% / test 0.1% ← even more than 1 million data
Mismatched train/test distribution
-
Mismatched distribution 상황
- 만약 training set이 webpage에서 가져온 cat pictures고, dev/test set이 유저들이 app을 활용하며 올린 cat pictures라면,
Dev와 Test set은 반드시 같은 distribution을 따라야 한다!
- 만약 training set이 webpage에서 가져온 cat pictures고, dev/test set이 유저들이 app을 활용하며 올린 cat pictures라면,
-
보통 "test" set이라 불렸던 set은 dev가 더 올바른 표현일 수 있으므로 dev라 칭하고자 한다.
Bias and Variance
-
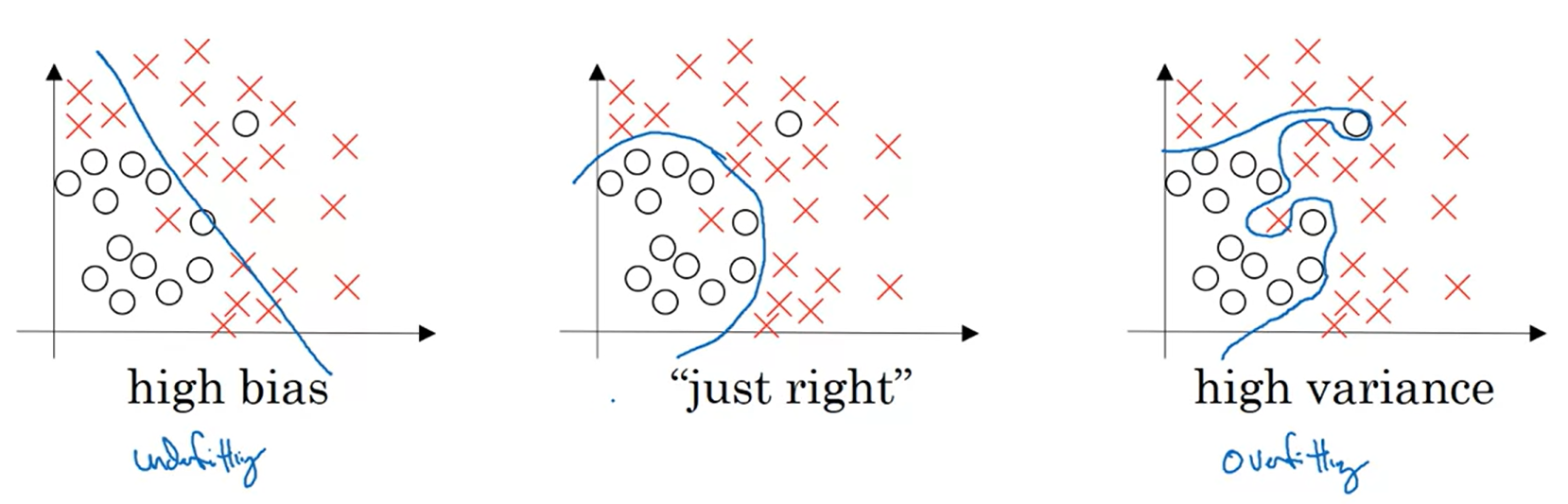
아래 그림과 같이 왼쪽처럼 모델이 너무 단순해서 분류 성능이 좋지 않으면 underfitting, 오른쪽처럼 너무 복잡해서 좋지 않으면 overfitting이라 한다.
- 가운데와 같이 적절한 지점을 찾아 분류하는 모델을 학습시키는 것이 중요하다!
-
예를 들어, Cat classification을 수행한다고 하자.
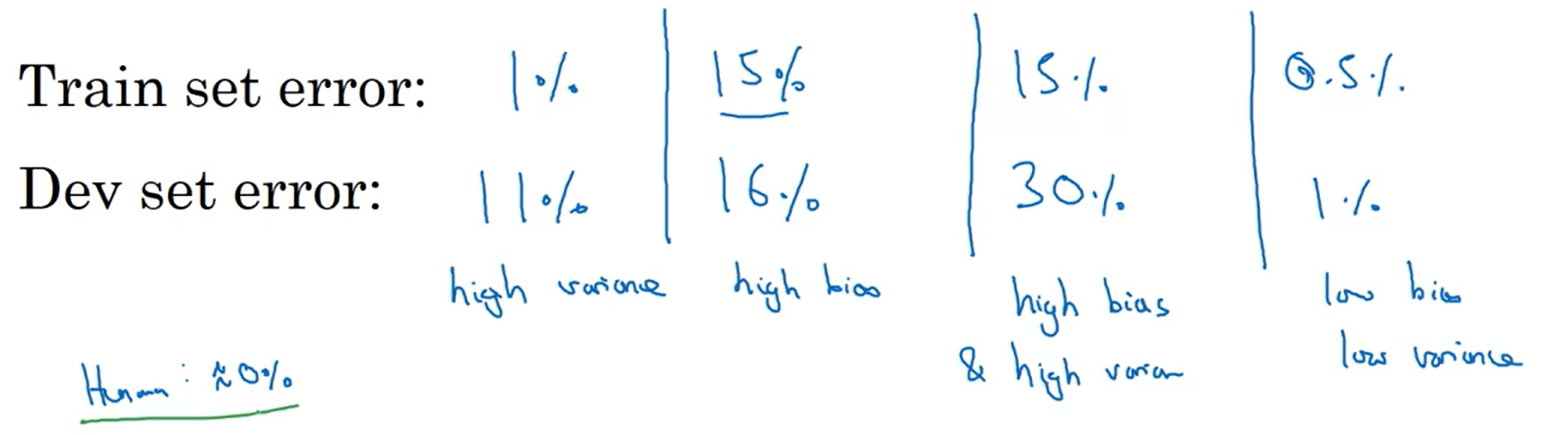
-
Human level performance가 0%일 때,
- Train / Dev error가 1% / 11%일 경우 : high variance ← "overfitting"
- Train / Dev error가 15% / 16%일 경우 : high bias ← "underfitting"
- Train / Dev error가 15% / 30%일 경우 : high bias & high variance
- Train / Dev error가 0.5% / 1%일 경우 : low bias & low variance
-
정리해 보면
- 첫 번째 상황은 overfitting, 두 번째 상황은 underfitting이다.
- 맨 마지막 상황이 가장 best인 상황에 해당한다.
- Variance가 높으면 Train 성능을 Dev(/Test) 성능이 따라가지 못한다는 뜻이고, Bias가 높으면 Train과 Dev(/Test) 모두 성능이 저평가되었다는 것을 뜻한다.
-
만약, Human level performance가 15%일 때는
- 기존 high bias라 판단했던 두 번째 상황이 좀 더 fit한 결과라고 봐도 무방하다.
- Blurry image가 껴있는 상황에서 Optimal (Bayes) error를 측정했을 때 이와 같은 상황이 벌어질 수 있다.
- 즉, 각 상황과 task에 맞는 performance를 잘 보고 판단하는 것이 중요하다.
-
-
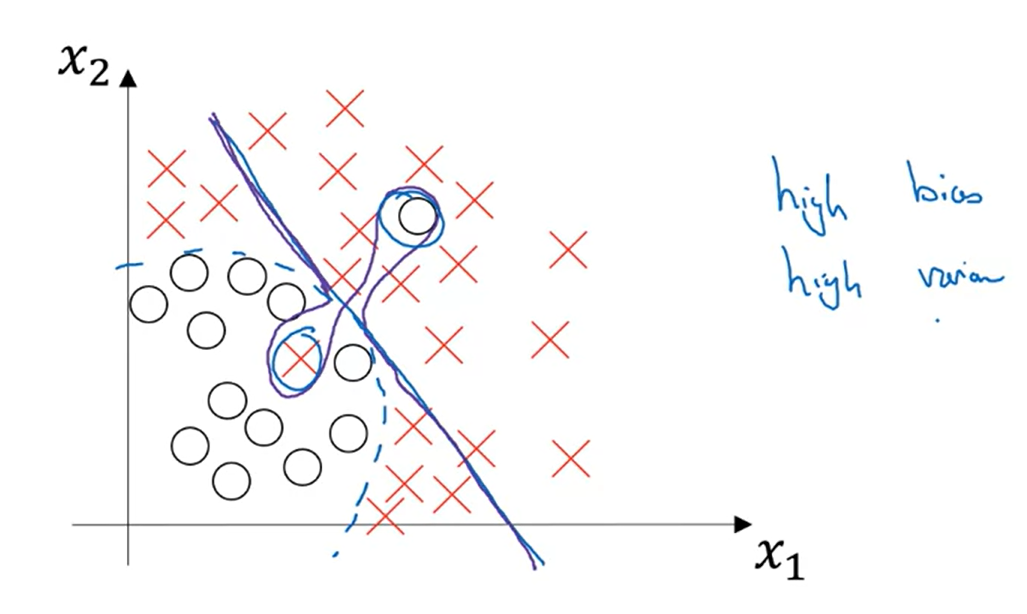
High bias and high variance 상황은 어떤 것인지 보자.
-
위 그림에서 보라색으로 표시된 부분이 high bias & high variance 상황이다.
-
파란색으로 표시된 직선 분류기가 underfitting 상황이고, 보라색이 overfitting이다.
-
즉, 훨씬 더 복잡한 curve function을 이루고 있고 특정 outlier들을 너무 많이 고려하는 유연성을 가지고 있다면 이런 문제가 발생할 수 있다.
-
특히나 차원이 많아질수록 이 현상은 문제를 크게 만들 수 있으므로 주의하도록 하자!
-
Basic Recipe for Machine Learning
- Underfitting : What if? High bias? (training data performance)
- Bigger network : layers ⇑
- Train longer (NN architecture search) : epoches ⇑
↓ N (repeat)
- Overfitting : What if? High variance? (dev set performance)
- More data
- Regularization (NN architecture search) ← after next video
↓ N
- Done
-
"Bias(↑↓) Variance(↓↑) trade off" : 서로 상반되는 영향을 끼친다!
-
'Bigger network' and 'More data' solved by appearing Deep learning
Regularizing your Neural Network
Regularization
-
일반적인 Logistic regresstion의 Cost function에서 더해지는
- ← 이 항을 말한다.
-
L2 norm으로 수식이 정리되기 때문에 L2 regularzation이라 하며,
- L1 norm을 적용한 L1 regularization도 있다.
-
parameter에 대하여 정규화 식을 적용할 수도 있지만 이는 생략한다.
- 는 일종의 parameter로서 norm의 scale을 조정한다.
- 를 프로그래밍 언어(python)으로 작성할 때 lambd로 쓰겠다.
- 기존에 lambda라는 내장 함수가 존재하기 때문이다.

-
Neural network 내에서는 layer들이 쌓여있기 때문에 notation이 다음과 같다.
-
참고로 아래 그림에 나와있는 것을 수정했다.
- W의 shape은 , current layer , previous layer
-
우리가 2 norm이라 부르는 것들은 관행적으로 Frobenius norm이라 한다.
-
-
Regularization을 적용한 수식은 derivative될 때 로 정리된다.
- Gradient descent 시 이 항에 넣어주면 되며, 이를 정리하면 Regularization이 Weight decay의 역할을 한다는 것을 알게 된다.
- Update 이후 항의 변화가 로 정리되기 때문이다!

Why Regularization Reduces Overfitting?
-
그렇다면 왜 Regularization이 Overfitting을 방지한다는 것인가?
-
기존 Cost function에 항을 추가하면 weight decay 효과를 낼 수 있다는 것을 알게 되었다.
- 즉, 의 원소 값을 줄일수록 overfitting을 방지할 수 있다는 직관을 얻을 수 있다.
-
만약, 항의 크기가 커지면 weight 항의 원소를 0에 가깝게 만들 수 있다.
- 이는 hidden unit의 영향을 없애는 과정과도 같아진다.
- 결국 몇몇 layer들의 영향만 가진 작고 단순한 신경망이 완성되는 것이다.
-
의 값을 크게 키울수록 신경망의 크기가 작아지면서 아래 그림과 같이 underfitting의 상황으로까지 이어질 수 있다.
- 적절한 "just rignt" 값을 찾는다면 좋은 성능을 가진 신경망이 완성될 것이다.
-
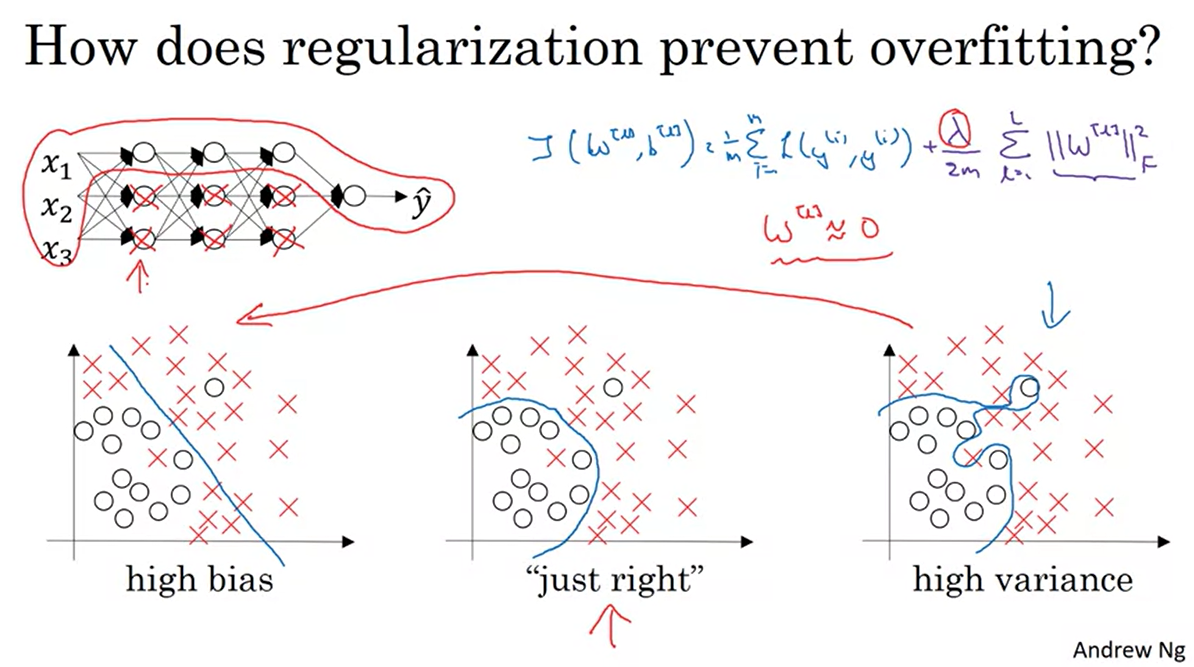
-
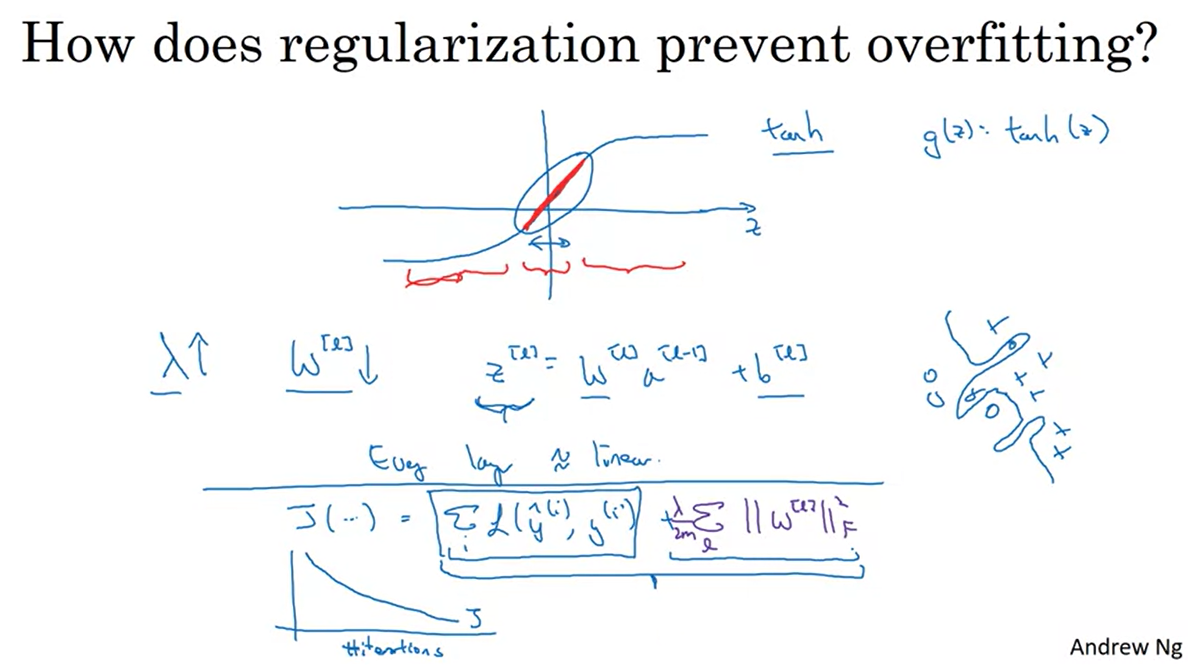
또 다른 예시를 살펴보자.
-
함수와 같은 경우 0과 가까운 부분에서는 linear한 function 꼴을 갖지만, 그 외의 z가 너무 작거나 너무 큰 경우는 그렇지 않다.
- Gradient 값이 미세해지면서 vanishing 문제에 빠질 수 있다.
-
의 크기를 키우면 weight 의 값도 줄어든다.
- 모든 layer가 linear function이라면 의 값이 작아질 때 값도 작아진다.
- 즉, z의 범위를 0에 가까운 값으로 보냄으로써 gradient update가 dynamic해질 수 있도록 만들 수 있는 것이다.
-
Debugging을 위해 plot 함수를 작성할 때에는 L2 panalty까지 추가한 Cost function을 그려야 한다.
- 기존 Cost function만으로는 이제 단조로운 감소 함수가 만들어지지 않을 것이다.
-
Dropout Regularization
-
Dropout이란 layer의 일부를 turn off하는 시스템이라고 이해하면 된다.
- 0.5만큼의 prob.로 layer가 dropout된다고 가정하면, 아래 그림과 같이 4개의 layer중 2개의 layer만 남기고 forward & backward 영향력이 사라진다는 것을 뜻한다.
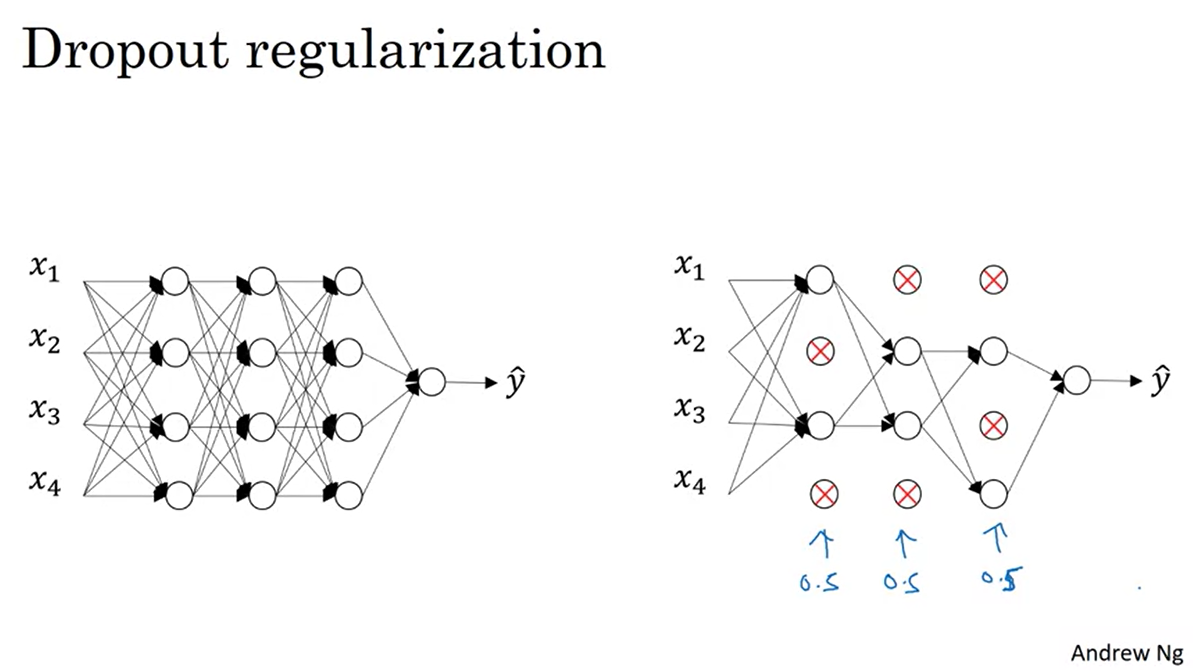
-
Dropout을 코드로 구현할 때는 "Inverted dropout" 테크닉을 사용한다.
keep_prob값을 0.8로 지정한다면 0.2 비율의 layer를 turn off한다는 것을 의미한다.keep_prob보다 작은 값을 True로 설정하는d3배열을 지정하고a3layer와의 곱으로 0.8 비율의 layer만 남긴다.- 이 때,
keep_prob으로 최종 결과값을 나눠주는 이유는 최종 layera3의 실제 수치가 20%만큼 떨어졌다고 할 때, 정규화를 시켜주는 방법은 0.8을 나눠주는 것이기 때문이다.

-
만약 dropout이 없다면 layer가 깊게 쌓일 때 수치가 exploding하거나 vanishing하게 될 위험성이 있다.
- 따라서 weight 수치를 안정화 시키기 위한 technique 중에 하나로 쓰인다.

Understanding Dropout
-
그렇다면 어째서 Dropout이 효과가 있는 것일까?
- 직관적으로 말하자면, dropout은 gradient에 L2 panalty를 주는 것과 비슷한 효과를 가진다.
- 몇몇 layer의 영향력을 꺼버림으로 인해 weights shrinking이 일어난다고 볼 수 있으며, Weights decay(L2) Shrink weights(Dropout)이기 때문이다.
-
Dropout은 Computer Vision task에 매우 효과적이다.
- 데이터의 수치값이 매우 많기 때문이며, 몇개의 layer를 꺼버림으로 인해서 여러 features의 영향력으로 분산시켜줄 수 있다는 장점이 있다.
-
또한, Dropout을 적용한 순간부터는 Cost function의 수치가 단조로울 수 없기 때문에 디버깅을 위해서라면 dropout을 끄고 plot하는게 좋다.
- 이 때에는
keep_prob의 값을 1로 놓고 plot하면 된다.
- 이 때에는

Other Regularization Methods
-
지금까지 했던 모든 것들은 Overfitting을 방지하는 것이었다.
-
Data augmentation 기법 또한 데이터 수집 비용을 줄이고, 효과적으로 training할 수 있는 기법 중에 하나로 손꼽힌다.
- 크게 Flip, Rotation, Shearing 기법들이 있다.
-
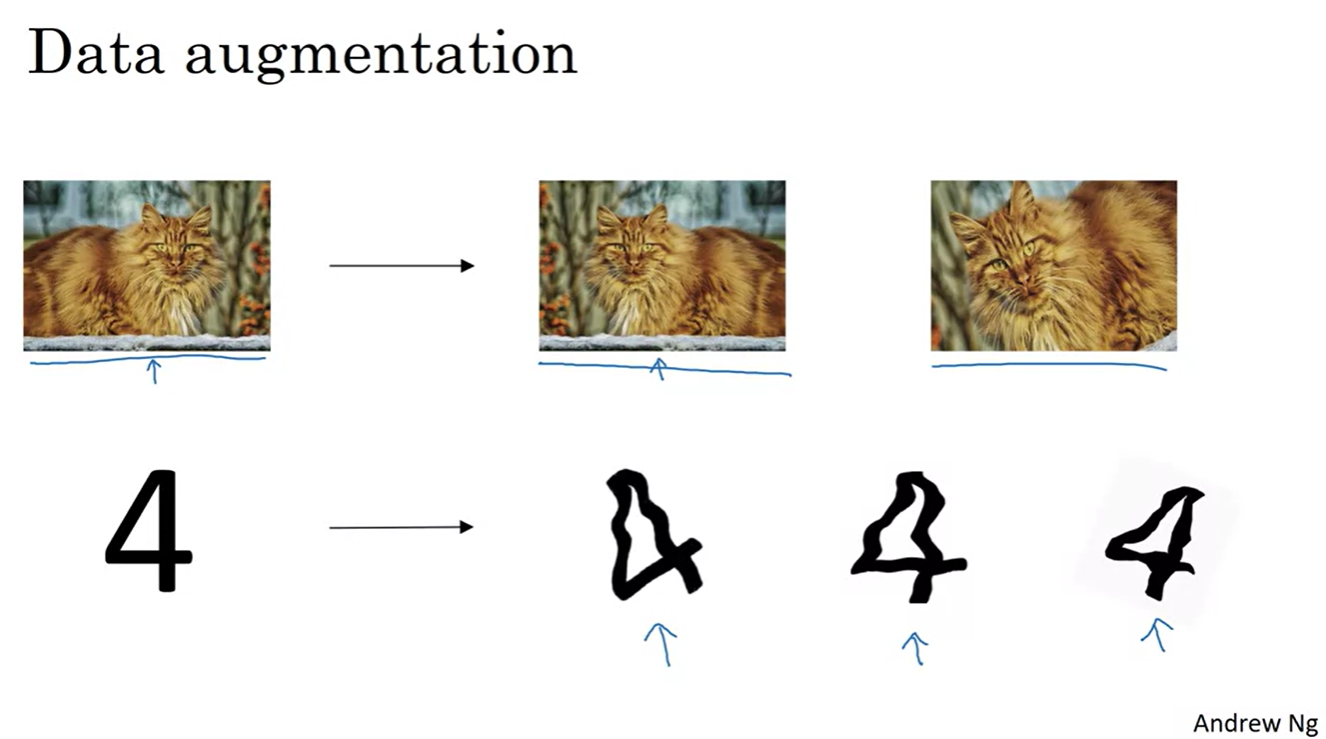
-
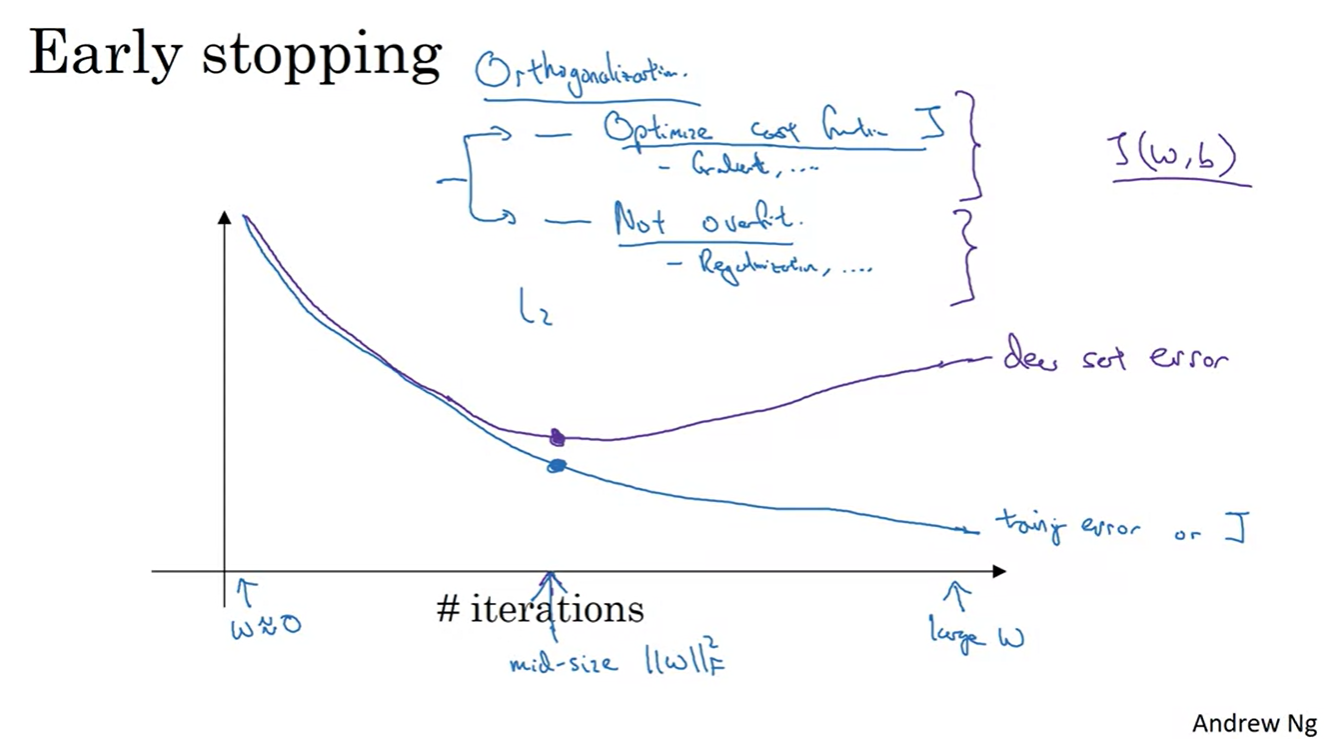
Early stopping 기법 또한 Overfitting을 막는 방법 중에 하나다.
-
만약 training error와 dev set error가 어느 순간을 기점으로 크게 차이가 나기 시작할 때, 훈련을 일찌감치 stop하는 기법이다.
- 예를 들어, error의 원인이 weight가 계속 커지기 때문이라면 적당한 mid-size 일 때 훈련을 멈추는 것이다.
-
그리고 Early stopping은 L2 norm panalty 기법에 비해서 계산 효율적인 방법론이라 할 수 있다.
- 특별한 계산 없이 적당한 시점에 훈련을 멈춰주기만 하면 되기 때문이다.
-
-
우리가 지금까지 보았던 훈련 기법들은 크게 두 가지로 나눠볼 수 있었다.
- Cost function 를 optimizing하는 기법: Gradient descent, ...
- Overfitting을 방지하는 기법: Regularization, L2 norm...
-
Andrew는 위의 두 가지 방법론이 Orthogonal한 성질을 가지고 있다고 했다.
- 둘 중 하나만 고려해서 문제를 해결할 것이 아니라, 두 가지 영향력을 모두 고려했을 때 최적의 solution을 찾아낼 수 있다는 의미로 받아들이면 된다.
Setting Up your Optimization Problem
Normalizing Inputs
-
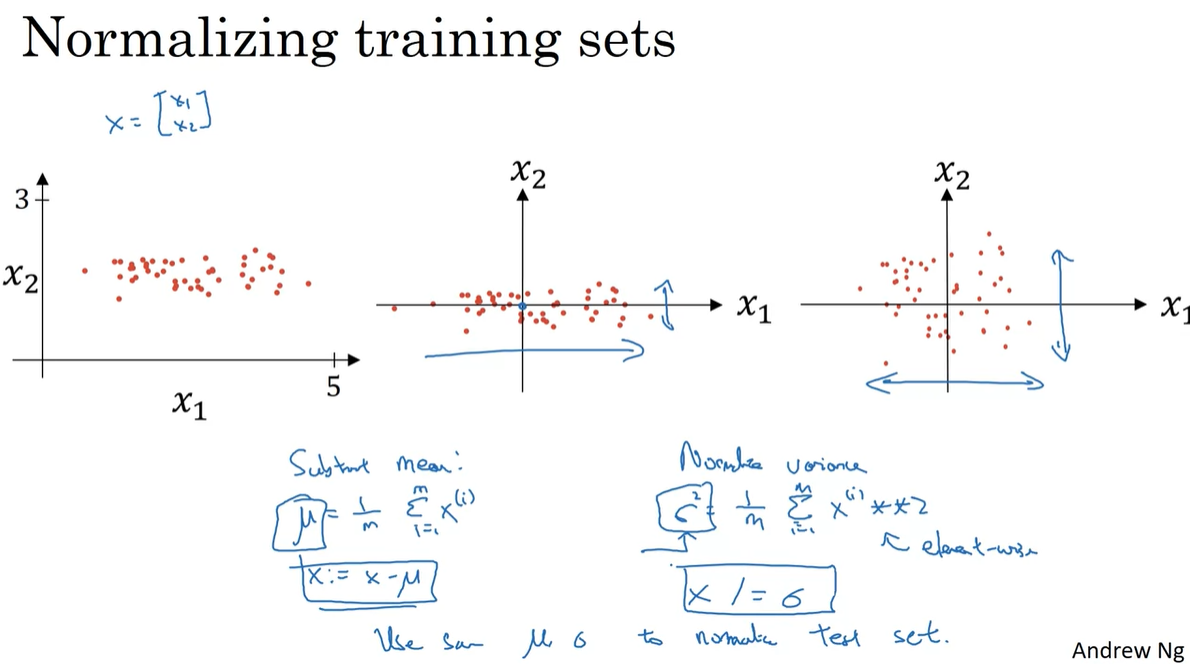
Training data가 로 주어진 상황을 가정해보자.
-
Vector 의 mean 를 구하여 로 업데이트 하자.
-
이제 Variance 를 구하여 로 업데이트 한 결과가 맨 오른쪽 plot과 같다.
- ←
-
이러한 과정을 Normalizing이라 한다.
- 최종 plotting 결과, 데이터 분포가 중심에 많이 모이게 되었음을 알 수 있다.
-
-
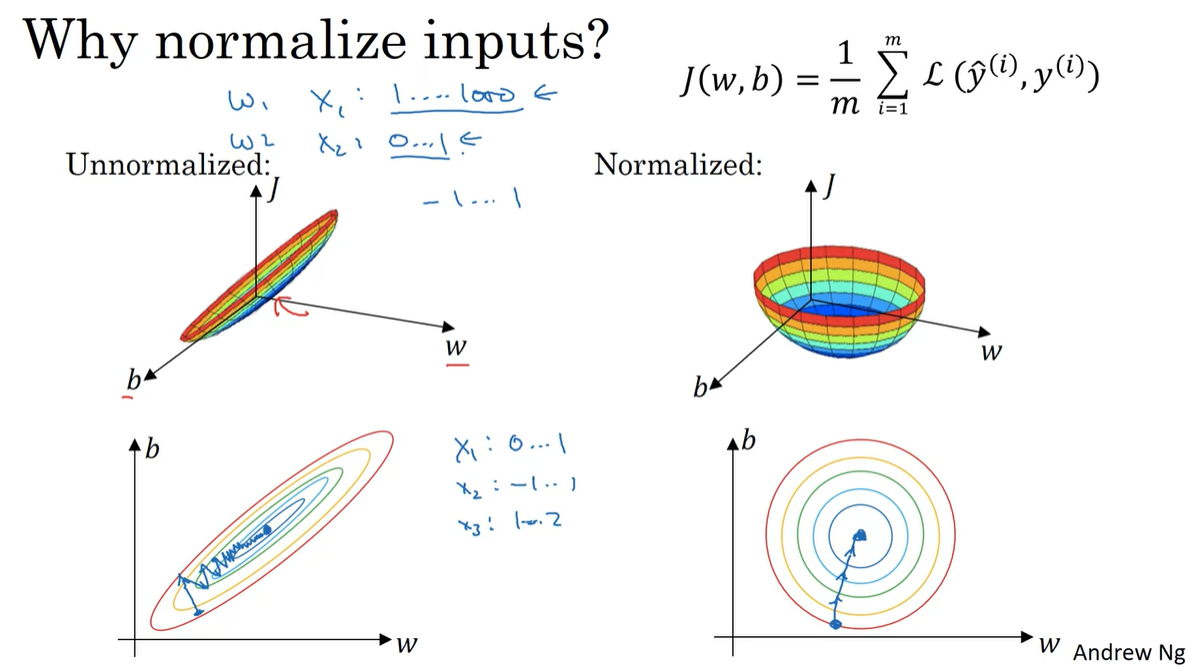
그렇다면 Normalizing은 왜 필요한 것일까?
-
Unnormalizing 상황에서의 cost function 그래프를 살펴보면 ex. 과 값의 분포 차이가 큰 이유로 weight 와 분포가 다소 치우칠 수 있다.
-
이러한 input을 Normalizing하면 Symmetric한 cost function을 얻을 수 있고, Unnormalizing 상황에 비해 bouncing하지 않는 매끄러운 학습이 이어진다.
-
Vanishing / Exploding Gradients
-
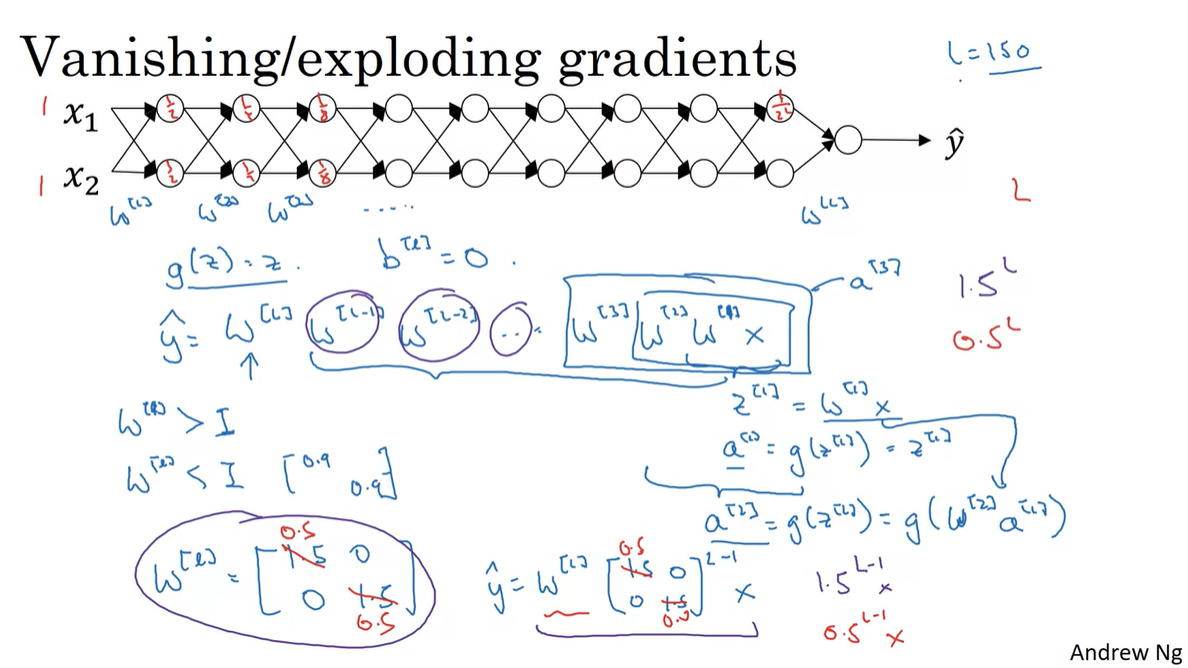
만약 아래와 같은 deep layer로 쌓인 neural network가 존재한다고 해보자.
-
추론값 은 weights s와 input data 의 연쇄적인 곱으로 계산된다.
-
한 층마다 가 곱해지고 나면, 는 activation function에 의해 로 계산이 이어진다.
-
의 한 value가 전부 이거나 일 때 개라고 가정한다면, 나 의 값은 매우 크거나 매우 작은 값을 갖게 된다.
-
Layer를 깊게 쌓으면 이러한 연산이 늘어나기 때문에, weight 의 원소가 1보다 클 때와 작을 때 모두 Exploding/Vanishing 문제를 일으킬 수 있다.
-
-
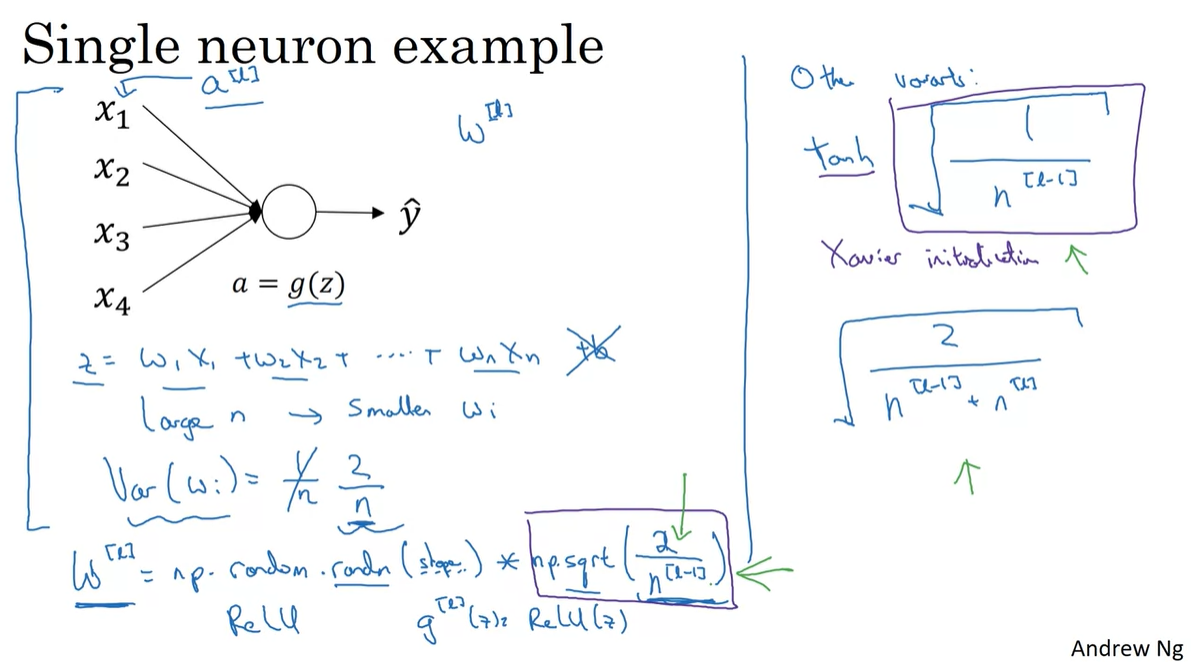
Weight Initialization for Deep Networks
-
한 layer를 통과하는 값은 linear transform으로 엮인 의 합으로 정의된다.
-
만약 layer 개수 이 늘어날 수록 값은 커지기 때문에 는 작은 value일수록 좋다.
- 따라서 의 분산을 로 맞춰주는 initialize 방법을 주로 사용한다.
-
코드로는 을 사용한다.
-
Activation이 ReLU일 경우 보다 가 더 유효하다는 검증에 의해, 를 대부분 사용한다.
-
Activation이 라면 를 보라색 박스에 교체시켜 사용한다.
← Xavier initialization
-
Bengio 교수님과 그의 제자들이 쓴 논문에 의해 ReLU activation에서 가 곱해진 값이 더 유용하다고 검증한 바 있다.
← He initialization
-
-
Numerical Approximation of Gradients
-
Gradient를 numerical하게 approximation하는 방법에 대해 소개한다.
-
우리는 매우 작은 범위 내에 있는 을 원래의 에 하여, { y축 값의 차이 / x축 값의 차이 }를 gradient라 정의한다.
-
Parameter Optimizing을 통해 계산한 와 numerical derivative로 계산한 가 같아지는지 확인하기 위한 debugging 과정에 이러한 기법을 사용할 것이다.
-
를 로 설정하면 의 compute 연산이
들어가기 때문에 으로 계산하여 의 복잡도로 해결한다.
-

Gradient Checking
-
Gradient Checking을 하기 위해서는 다양한 parameters를 와 로 통합하는 과정이 필요하다.
- ex. 와 를 하나의 큰 vector로 만들기 위해 concaternate한다.

-
각 항들(
i)에 대해서 정도의 값을 설정하여, Cost function의 gradient를 계산한 를 구한다.-
이 값이 와 얼마나 차이가 있는지 알아보기 위해 를 계산한다.
- 만약 이 차이가 정도를 만족한다면 great, 이면 worry라고 판단하도록 하여 gradient 계산이 잘 이루어지고 있는지 debugging하면 된다.
-

Gradient Checking Implementation Notes
-
Gradient Checking 시 주의해야 할 점이 몇 가지 존재한다.
-
Training 시에 사용하지 말고 debugging 시에만 사용한다.
-
만약 grad check 시 실패하였다고 뜬다면, bug를 찾아내기 위해 직접 와 를 비교해보라.
-
Cost function에 Regularization 하는 것 또한 잊지 말아야 한다. ← ~ ...
-
Dropout과 함께 작업하지 말아야 한다. ←
keep_prob을 1.0으로 설정한다. -
Random initialization을 반복하며 training을 지속적으로 시도해보자.
-

Assignment
W1A1
Initialization
-
Model differed by initialization
- Zeros
- Random
- He
def model(X, Y, learning_rate = 0.01, num_iterations = 15000, print_cost = True, initialization = "he"):
"""
Implements a three-layer neural network: LINEAR->RELU->LINEAR->RELU->LINEAR->SIGMOID.
Arguments:
X -- input data, of shape (2, number of examples)
Y -- true "label" vector (containing 0 for red dots; 1 for blue dots), of shape (1, number of examples)
learning_rate -- learning rate for gradient descent
num_iterations -- number of iterations to run gradient descent
print_cost -- if True, print the cost every 1000 iterations
initialization -- flag to choose which initialization to use ("zeros","random" or "he")
Returns:
parameters -- parameters learnt by the model
"""
grads = {}
costs = [] # to keep track of the loss
m = X.shape[1] # number of examples
layers_dims = [X.shape[0], 10, 5, 1]
# Initialize parameters dictionary.
if initialization == "zeros":
parameters = initialize_parameters_zeros(layers_dims)
elif initialization == "random":
parameters = initialize_parameters_random(layers_dims)
elif initialization == "he":
parameters = initialize_parameters_he(layers_dims)
# Loop (gradient descent)
for i in range(num_iterations):
# Forward propagation: LINEAR -> RELU -> LINEAR -> RELU -> LINEAR -> SIGMOID.
a3, cache = forward_propagation(X, parameters)
# Loss
cost = compute_loss(a3, Y)
# Backward propagation.
grads = backward_propagation(X, Y, cache)
# Update parameters.
parameters = update_parameters(parameters, grads, learning_rate)
# Print the loss every 1000 iterations
if print_cost and i % 1000 == 0:
print("Cost after iteration {}: {}".format(i, cost))
costs.append(cost)
# plot the loss
plt.plot(costs)
plt.ylabel('cost')
plt.xlabel('iterations (per hundreds)')
plt.title("Learning rate =" + str(learning_rate))
plt.show()
return parameters- Zero initialization
def initialize_parameters_zeros(layers_dims):
"""
Arguments:
layer_dims -- python array (list) containing the size of each layer.
Returns:
parameters -- python dictionary containing your parameters "W1", "b1", ..., "WL", "bL":
W1 -- weight matrix of shape (layers_dims[1], layers_dims[0])
b1 -- bias vector of shape (layers_dims[1], 1)
...
WL -- weight matrix of shape (layers_dims[L], layers_dims[L-1])
bL -- bias vector of shape (layers_dims[L], 1)
"""
parameters = {}
L = len(layers_dims) # number of layers in the network
for l in range(1, L):
parameters['W' + str(l)] = np.zeros((layers_dims[l], layers_dims[l-1])) * 0.01 # 1-0
parameters['b' + str(l)] = np.zeros((layers_dims[l], 1))
return parameters
>>> W1 = [[0. 0. 0.]
[0. 0. 0.]]
b1 = [[0.]
[0.]]
W2 = [[0. 0.]]
b2 = [[0.]]-
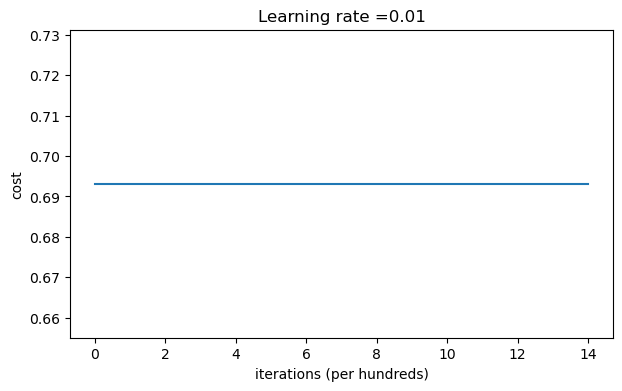
사실상 아주 작게 진동하는 그래프다.
-
Loss가 계속해서 0.6931.. 값을 나타내는 이유는 0 weights에 의해 을 만들어버린 후, 가 계속적으로 0.5를 만족하기 때문에 생긴 결과다.
-
-
>>> Cost after iteration 0: 0.6931471805599453
Cost after iteration 1000: 0.6931471805599453
Cost after iteration 2000: 0.6931471805599453
Cost after iteration 3000: 0.6931471805599453
Cost after iteration 4000: 0.6931471805599453
... 
- Prediction 결과 두 경계선을 거의 구분하지 못한다.
>>>
predictions_train = [[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 ...]]
predictions_test = [[0 0 0 0 0 0 0 ...]] 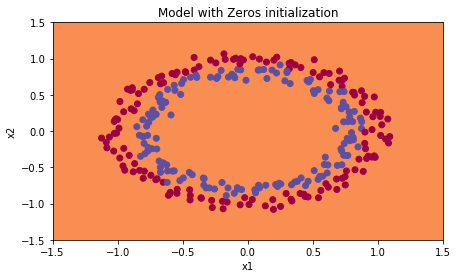
- Random Initialization
def initialize_parameters_random(layers_dims):
"""
Arguments:
layer_dims -- python array (list) containing the size of each layer.
Returns:
parameters -- python dictionary containing your parameters "W1", "b1", ..., "WL", "bL":
W1 -- weight matrix of shape (layers_dims[1], layers_dims[0])
b1 -- bias vector of shape (layers_dims[1], 1)
...
WL -- weight matrix of shape (layers_dims[L], layers_dims[L-1])
bL -- bias vector of shape (layers_dims[L], 1)
"""
np.random.seed(3) # This seed makes sure your "random" numbers will be the as ours
parameters = {}
L = len(layers_dims) # integer representing the number of layers
for l in range(1, L):
parameters['W' + str(l)] = np.random.randn(layers_dims[l], layers_dims[l-1]) * 10 # scaled by *10
parameters['b' + str(l)] = np.zeros((layers_dims[l], 1))
return parameters
>>> W1 = [[ 17.88628473 4.36509851 0.96497468]
[-18.63492703 -2.77388203 -3.54758979]]
b1 = [[0.]
[0.]]
W2 = [[-0.82741481 -6.27000677]]
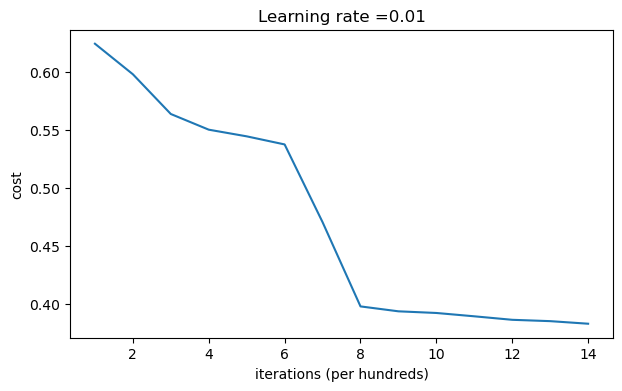
b2 = [[0.]]- Prediction 결과 안정적인 Loss curve를 가지게 되었음을 알 수 있다.
>>> Cost after iteration 0: inf
Cost after iteration 1000: 0.6242305622140936
Cost after iteration 2000: 0.5978835450878724
Cost after iteration 3000: 0.5636222799576053
Cost after iteration 4000: 0.5501000257265779
... 
-
Boundary는 zero initialization보다는 향상되었다.
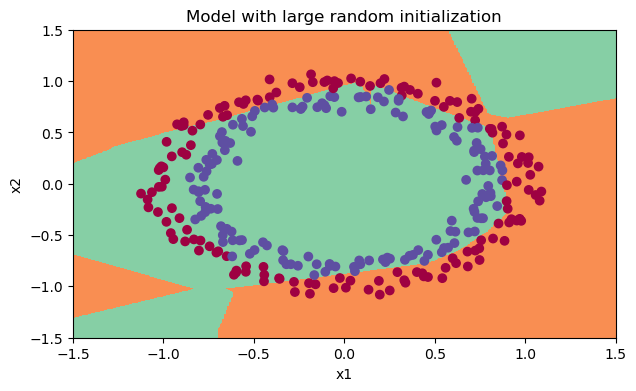
-
He initialization
-
를 기대하기 때문에
- 코드가 핵심이다.
-
def initialize_parameters_he(layers_dims):
"""
Arguments:
layer_dims -- python array (list) containing the size of each layer.
Returns:
parameters -- python dictionary containing your parameters "W1", "b1", ..., "WL", "bL":
W1 -- weight matrix of shape (layers_dims[1], layers_dims[0])
b1 -- bias vector of shape (layers_dims[1], 1)
...
WL -- weight matrix of shape (layers_dims[L], layers_dims[L-1])
bL -- bias vector of shape (layers_dims[L], 1)
"""
np.random.seed(3)
parameters = {}
L = len(layers_dims) - 1 # integer representing the number of layers
for l in range(1, L + 1):
parameters['W' + str(l)] = np.random.randn(layers_dims[l], layers_dims[l-1]) * np.sqrt(2./layers_dims[l-1])
parameters['b' + str(l)] = np.zeros((layers_dims[l], 1))
return parameters
>>> W1 = [[ 1.78862847 0.43650985]
[ 0.09649747 -1.8634927 ]
[-0.2773882 -0.35475898]
[-0.08274148 -0.62700068]]
b1 = [[0.]
[0.]
[0.]
[0.]]
W2 = [[-0.03098412 -0.33744411 -0.92904268 0.62552248]]
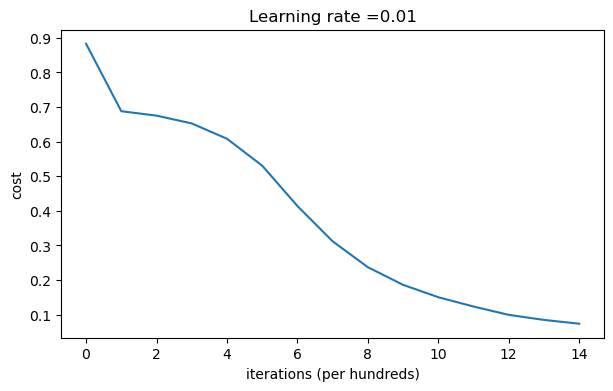
b2 = [[0.]]- Prediction 결과 가장 매끄럽다.
>>> Cost after iteration 0: 0.8830537463419761
Cost after iteration 1000: 0.6879825919728063
Cost after iteration 2000: 0.6751286264523371
Cost after iteration 3000: 0.6526117768893807
Cost after iteration 4000: 0.6082958970572938
... 
-
Boundary 또한 매우 잘 형성되었음을 볼 수 있다.
- 이러한 결과로 인해 알 수 있는 건 ReLU activation을 쓸 때 He initialization을 이용하는 것이 가장 적합하다는 점이다.
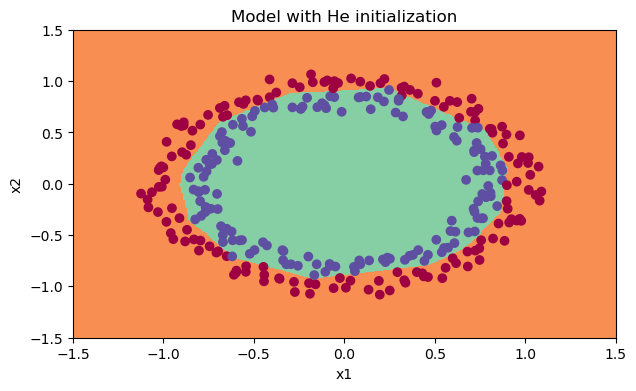
Regularization
-
아래와 같은 축구장에 두 팀을 배치하고, 우리 팀의 수비 진영을 확보하고 싶다.
- 이러한 경계선(boundary)를 만들어주는 AI expert를 제작해보자.
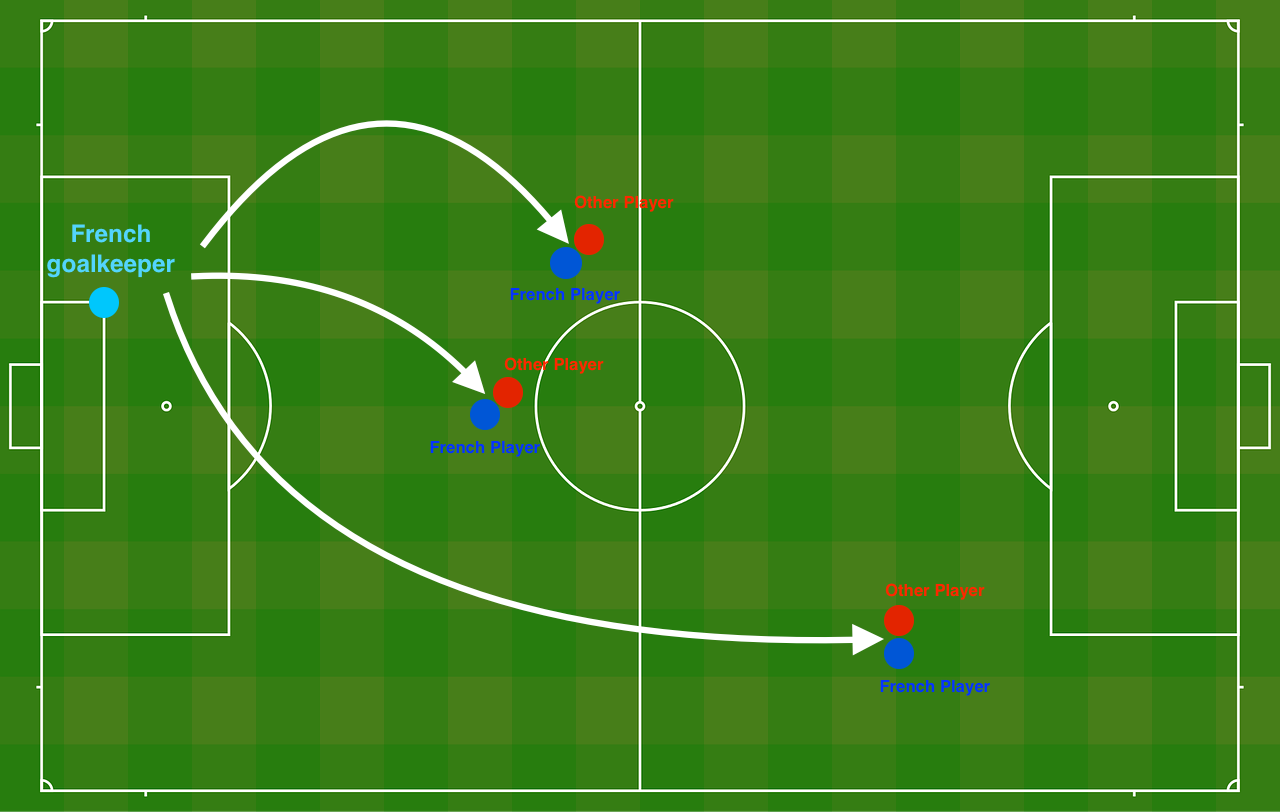
-
Non-Regularized model
def model(X, Y, learning_rate = 0.3, num_iterations = 30000, print_cost = True, lambd = 0, keep_prob = 1):
"""
Implements a three-layer neural network: LINEAR->RELU->LINEAR->RELU->LINEAR->SIGMOID.
Arguments:
X -- input data, of shape (input size, number of examples)
Y -- true "label" vector (1 for blue dot / 0 for red dot), of shape (output size, number of examples)
learning_rate -- learning rate of the optimization
num_iterations -- number of iterations of the optimization loop
print_cost -- If True, print the cost every 10000 iterations
lambd -- regularization hyperparameter, scalar
keep_prob - probability of keeping a neuron active during drop-out, scalar.
Returns:
parameters -- parameters learned by the model. They can then be used to predict.
"""
grads = {}
costs = [] # to keep track of the cost
m = X.shape[1] # number of examples
layers_dims = [X.shape[0], 20, 3, 1]
# Initialize parameters dictionary.
parameters = initialize_parameters(layers_dims)
# Loop (gradient descent)
for i in range(0, num_iterations):
# Forward propagation: LINEAR -> RELU -> LINEAR -> RELU -> LINEAR -> SIGMOID.
if keep_prob == 1:
a3, cache = forward_propagation(X, parameters)
elif keep_prob < 1:
a3, cache = forward_propagation_with_dropout(X, parameters, keep_prob)
# Cost function
if lambd == 0:
cost = compute_cost(a3, Y)
else:
cost = compute_cost_with_regularization(a3, Y, parameters, lambd)
# Backward propagation.
assert (lambd == 0 or keep_prob == 1) # it is possible to use both L2 regularization and dropout,
# but this assignment will only explore one at a time
if lambd == 0 and keep_prob == 1:
grads = backward_propagation(X, Y, cache)
elif lambd != 0: # only regularization
grads = backward_propagation_with_regularization(X, Y, cache, lambd)
elif keep_prob < 1: # only dropout
grads = backward_propagation_with_dropout(X, Y, cache, keep_prob)
# Update parameters.
parameters = update_parameters(parameters, grads, learning_rate)
# Print the loss every 10000 iterations
if print_cost and i % 10000 == 0:
print("Cost after iteration {}: {}".format(i, cost))
if print_cost and i % 1000 == 0:
costs.append(cost)
# plot the cost
plt.plot(costs)
plt.ylabel('cost')
plt.xlabel('iterations (x1,000)')
plt.title("Learning rate =" + str(learning_rate))
plt.show()
return parameters-
Training 결과 안정적인 loss 커브는 달성했으나 boundary 양상은 다소 튀는 부분이 생겼다.
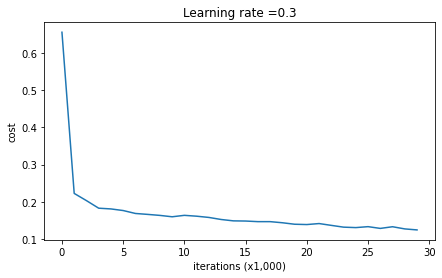
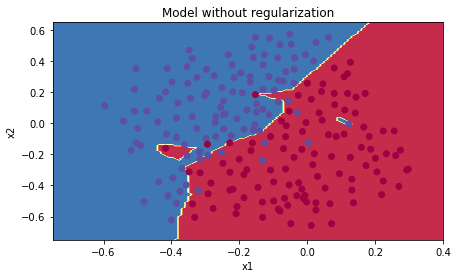
-
Cost function에 L2 regularization term을 추가해보자.
def compute_cost_with_regularization(A3, Y, parameters, lambd):
"""
Implement the cost function with L2 regularization. See formula (2) above.
Arguments:
A3 -- post-activation, output of forward propagation, of shape (output size, number of examples)
Y -- "true" labels vector, of shape (output size, number of examples)
parameters -- python dictionary containing parameters of the model
Returns:
cost - value of the regularized loss function (formula (2))
"""
m = Y.shape[1]
W1 = parameters["W1"]
W2 = parameters["W2"]
W3 = parameters["W3"]
cross_entropy_cost = compute_cost(A3, Y) # This gives you the cross-entropy part of the cost
L2_regularization_cost = lambd/(2*m) * (np.sum(np.square(W1)) + np.sum(np.square(W2)) + np.sum(np.square(W3)))
cost = cross_entropy_cost + L2_regularization_cost
return cost-
Backward propagation with regularization
def backward_propagation_with_regularization(X, Y, cache, lambd):
"""
Implements the backward propagation of our baseline model to which we added an L2 regularization.
Arguments:
X -- input dataset, of shape (input size, number of examples)
Y -- "true" labels vector, of shape (output size, number of examples)
cache -- cache output from forward_propagation()
lambd -- regularization hyperparameter, scalar
Returns:
gradients -- A dictionary with the gradients with respect to each parameter, activation and pre-activation variables
"""
m = X.shape[1]
(Z1, A1, W1, b1, Z2, A2, W2, b2, Z3, A3, W3, b3) = cache
dZ3 = A3 - Y
dW3 = 1./m * np.dot(dZ3, A2.T) + lambd/m * (W3)
db3 = 1./m * np.sum(dZ3, axis=1, keepdims=True)
dA2 = np.dot(W3.T, dZ3)
dZ2 = np.multiply(dA2, np.int64(A2 > 0))
dW2 = 1./m * np.dot(dZ2, A1.T) + lambd/m * (W2)
db2 = 1./m * np.sum(dZ2, axis=1, keepdims=True)
dA1 = np.dot(W2.T, dZ2)
dZ1 = np.multiply(dA1, np.int64(A1 > 0))
dW1 = 1./m * np.dot(dZ1, X.T) + lambd/m * (W1)
db1 = 1./m * np.sum(dZ1, axis=1, keepdims=True)
gradients = {"dZ3": dZ3, "dW3": dW3, "db3": db3,"dA2": dA2,
"dZ2": dZ2, "dW2": dW2, "db2": db2, "dA1": dA1,
"dZ1": dZ1, "dW1": dW1, "db1": db1}
return gradients-
Regularization이 들어가야 학습이 더욱 안정적으로 일어나며 boundary 결과 또한 매끄럽다.

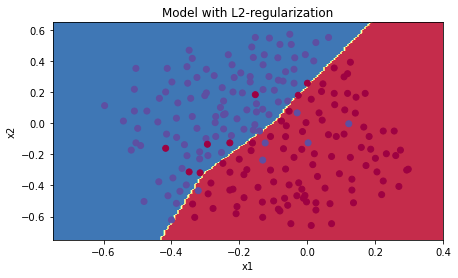
-
Dropout이 포함된 코드를 살펴보자.
-
D = (D < keep_prob).astype(int)로 masking을 설정한다.keep_prob을 0.8로 주면 20%의 node가 꺼지게 만든다는 것을 의미하며, 이를 기존 에 곱해 로 20% node를 masking한다.
-
-
Forward propagation with Dropout
def forward_propagation_with_dropout(X, parameters, keep_prob = 0.5):
"""
Implements the forward propagation: LINEAR -> RELU + DROPOUT -> LINEAR -> RELU + DROPOUT -> LINEAR -> SIGMOID.
Arguments:
X -- input dataset, of shape (2, number of examples)
parameters -- python dictionary containing your parameters "W1", "b1", "W2", "b2", "W3", "b3":
W1 -- weight matrix of shape (20, 2)
b1 -- bias vector of shape (20, 1)
W2 -- weight matrix of shape (3, 20)
b2 -- bias vector of shape (3, 1)
W3 -- weight matrix of shape (1, 3)
b3 -- bias vector of shape (1, 1)
keep_prob - probability of keeping a neuron active during drop-out, scalar
Returns:
A3 -- last activation value, output of the forward propagation, of shape (1,1)
cache -- tuple, information stored for computing the backward propagation
"""
np.random.seed(1)
# retrieve parameters
W1 = parameters["W1"]
b1 = parameters["b1"]
W2 = parameters["W2"]
b2 = parameters["b2"]
W3 = parameters["W3"]
b3 = parameters["b3"]
# LINEAR -> RELU -> LINEAR -> RELU -> LINEAR -> SIGMOID
Z1 = np.dot(W1, X) + b1
A1 = relu(Z1)
D1 = np.random.rand(A1.shape[0], A1.shape[1]) # rand : 0~1 균일 분포, randn : 0~1 가우시안 정규 분포(대체로 0 주변)
D1 = (D1 < keep_prob).astype(int) # masked
A1 = A1 * D1
A1 /= keep_prob
Z2 = np.dot(W2, A1) + b2
A2 = relu(Z2)
D2 = np.random.rand(A2.shape[0], A2.shape[1]) # rand : 0~1 균일 분포, randn : 0~1 가우시안 정규 분포
D2 = (D2 < keep_prob).astype(int) # masked
A2 = A2 * D2
A2 /= keep_prob
Z3 = np.dot(W3, A2) + b3
A3 = sigmoid(Z3)
cache = (Z1, D1, A1, W1, b1, Z2, D2, A2, W2, b2, Z3, A3, W3, b3)
return A3, cache- Backward propagation with Dropout
def backward_propagation_with_dropout(X, Y, cache, keep_prob):
"""
Implements the backward propagation of our baseline model to which we added dropout.
Arguments:
X -- input dataset, of shape (2, number of examples)
Y -- "true" labels vector, of shape (output size, number of examples)
cache -- cache output from forward_propagation_with_dropout()
keep_prob - probability of keeping a neuron active during drop-out, scalar
Returns:
gradients -- A dictionary with the gradients with respect to each parameter, activation and pre-activation variables
"""
m = X.shape[1]
(Z1, D1, A1, W1, b1, Z2, D2, A2, W2, b2, Z3, A3, W3, b3) = cache
dZ3 = A3 - Y
dW3 = 1./m * np.dot(dZ3, A2.T)
db3 = 1./m * np.sum(dZ3, axis=1, keepdims=True)
dA2 = np.dot(W3.T, dZ3)
dA2 = D2 * dA2
dA2 /= keep_prob
dZ2 = np.multiply(dA2, np.int64(A2 > 0))
dW2 = 1./m * np.dot(dZ2, A1.T)
db2 = 1./m * np.sum(dZ2, axis=1, keepdims=True)
dA1 = np.dot(W2.T, dZ2)
dA1 = dA1 * D1
dA1 /= keep_prob
dZ1 = np.multiply(dA1, np.int64(A1 > 0))
dW1 = 1./m * np.dot(dZ1, X.T)
db1 = 1./m * np.sum(dZ1, axis=1, keepdims=True)
gradients = {"dZ3": dZ3, "dW3": dW3, "db3": db3,"dA2": dA2,
"dZ2": dZ2, "dW2": dW2, "db2": db2, "dA1": dA1,
"dZ1": dZ1, "dW1": dW1, "db1": db1}
return gradientsGradient Checking
-
Gradient를 수식적으로 표현하면 다음과 같다.
# GRADED FUNCTION: gradient_check
def gradient_check(x, theta, epsilon=1e-7, print_msg=False):
"""
Implement the gradient checking presented in Figure 1.
Arguments:
x -- a float input
theta -- our parameter, a float as well
epsilon -- tiny shift to the input to compute approximated gradient with formula(1)
Returns:
difference -- difference (2) between the approximated gradient and the backward propagation gradient. Float output
"""
# Compute gradapprox using right side of formula (1). epsilon is small enough, you don't need to worry about the limit.
theta_plus = theta + epsilon
theta_minus = theta - epsilon
J_plus = forward_propagation(x, theta_plus)
J_minus = forward_propagation(x, theta_minus)
gradapprox = (J_plus - J_minus) / (2 * epsilon) # () is necessary!!
# Check if gradapprox is close enough to the output of backward_propagation()
grad = backward_propagation(x, theta) # real grad!
numerator = np.linalg.norm(grad - gradapprox) # None : frobenius norm
denominator = np.linalg.norm(grad) + np.linalg.norm(gradapprox) # 'fro' is okay
difference = numerator / denominator
if print_msg:
if difference > 2e-7:
print ("\033[93m" + "There is a mistake in the backward propagation! difference = " + str(difference) + "\033[0m")
else:
print ("\033[92m" + "Your backward propagation works perfectly fine! difference = " + str(difference) + "\033[0m")
return difference- N-dimentional Gradient checking은 for loop이 포함된다.
# GRADED FUNCTION: gradient_check_n
def gradient_check_n(parameters, gradients, X, Y, epsilon=1e-7, print_msg=False):
"""
Checks if backward_propagation_n computes correctly the gradient of the cost output by forward_propagation_n
Arguments:
parameters -- python dictionary containing your parameters "W1", "b1", "W2", "b2", "W3", "b3"
grad -- output of backward_propagation_n, contains gradients of the cost with respect to the parameters
X -- input datapoint, of shape (input size, number of examples)
Y -- true "label"
epsilon -- tiny shift to the input to compute approximated gradient with formula(1)
Returns:
difference -- difference (2) between the approximated gradient and the backward propagation gradient
"""
# Set-up variables
parameters_values, _ = dictionary_to_vector(parameters)
grad = gradients_to_vector(gradients)
num_parameters = parameters_values.shape[0]
J_plus = np.zeros((num_parameters, 1))
J_minus = np.zeros((num_parameters, 1))
gradapprox = np.zeros((num_parameters, 1))
# Compute gradapprox
for i in range(num_parameters):
# Compute J_plus[i]. Inputs: "parameters_values, epsilon". Output = "J_plus[i]".
theta_plus = np.copy(parameters_values)
theta_plus[i] += epsilon
J_plus[i], _ = forward_propagation_n(X, Y, vector_to_dictionary(theta_plus))
# Compute J_minus[i]. Inputs: "parameters_values, epsilon". Output = "J_minus[i]".
theta_minus = np.copy(parameters_values)
theta_minus[i] -= epsilon
J_minus[i], _ = forward_propagation_n(X, Y, vector_to_dictionary(theta_minus))
# Compute gradapprox[i]
gradapprox[i] = (J_plus[i] - J_minus[i]) / (2 * epsilon)
# Compare gradapprox to backward propagation gradients by computing difference.
numerator = np.linalg.norm(grad-gradapprox)
denominator = np.linalg.norm(grad) + np.linalg.norm(gradapprox)
difference = numerator / denominator
if print_msg:
if difference > 2e-7:
print ("\033[93m" + "There is a mistake in the backward propagation! difference = " + str(difference) + "\033[0m")
else:
print ("\033[92m" + "Your backward propagation works perfectly fine! difference = " + str(difference) + "\033[0m")
return difference