4.1 누락된 데이터 다루기
4.1.1 테이블 형태 데이터에서 누락된 값 식별
☑️CSV로부터 간단한 예제 데이터셋 만들기☑️
- read_csv 함수 : CSV 포맷의 데이터를 판다스 DataFrame으로 읽어들임
- 두 개의 누락된 값은 NaN으로 바꿈
- StringIO 함수 : 하드 디스크에 있는 일반 CSV 파일처럼 csv_data에 저장된 문자열을 읽어들임
import pandas as pd
from io import StringIO
import sys
csv_data = \
'''A,B,C,D
1.0,2.0,3.0,4.0
5.0,6.0,,8.0
10.0,11.0,12.0,'''
# 파이썬 2.7을 사용하는 경우
# 다음과 같이 문자열을 유니코드로 변환해야 합니다:
#if (sys.version_info < (3, 0)):
# csv_data = unicode(csv_data)
df = pd.read_csv(StringIO(csv_data))
df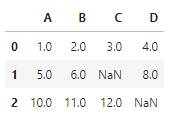
4.1.2 누락된 값이 있는 훈련 샘플이나 특성 제외
- dropna 메서드 : 누락된 값이 있는 행을 삭제
# 누락된 값이 있는 행을 삭제합니다
df.dropna(axis=0)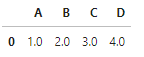
- axis=1 : NaN이 하나라도 있는 열을 삭제
# 누락된 값이 있는 열을 삭제합니다
df.dropna(axis=1)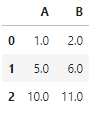
- how='all' : 모든 열이 NaN일 때만 행을 삭제
# 모든 열이 NaN인 행을 삭제합니다
# (여기서는 모든 값이 NaN인 행이 없기 때문에 전체 배열이 반환됨)
df.dropna(how='all') 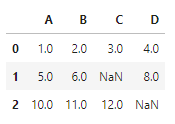
- thresh=4 : NaN이 아닌 값이 네 개보다 작은 행을 삭제
# NaN 값이 네 개보다 작은 행을 삭제합니다
df.dropna(thresh=4)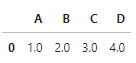
- subset=['C'] : 특정 열에 NaN이 있는 행만 삭제(여기서는 'C'열)
# 특정 열에 NaN이 있는 행만 삭제합니다(여기서는 'C'열)
df.dropna(subset=['C'])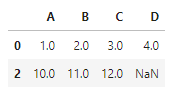
➡️ 단점 : 너무 많은 데이터를 제거하면 안정된 분석 불가능, 중요한 정보를 잃을 위험
4.1.3 누락된 값 대체
보간 기법 : 데이터셋에 있는 다른 훈련 샘플로부터 누락된 값을 추정
📍종류📍
1) 평균으로 대체 : 각 특성 열의 전체 평균으로 누락된 값을 바꿈
- 사이킷런의 SimpleImputer 클래스 사용하면 처리 가능
- strategy='median' : 데이터를 순서대로 나열했을 때 중간에 위치한 값으로 누락된 값을 대체
- strategy='most_frequent' : 가장 많이 나타난 값으로 누락된 값을 대체
- strategy='mean' : 전체 평균으로 누락된 값을 대체
# 행의 평균으로 누락된 값 대체하기
from sklearn.impute import SimpleImputer
import numpy as np
imr = SimpleImputer(missing_values=np.nan, strategy='mean')
imr = imr.fit(df.values)
imputed_data = imr.transform(df.values)
imputed_data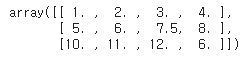
-
판다스의 fillna 메서드에 매개변수로 누락된 값을 채울 방법 전달
-
df.mean() : 평균값으로 누락된 값을 대체
df.fillna(df.mean())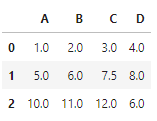
-
method='bfill' : 누락된 값을 다음 행의 값으로 대체
df.fillna(method='bfill') # method='backfill'와 같습니다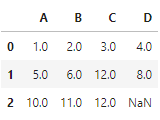
-
method='ffill' : 누락된 값을 이전 행의 값으로 대체
df.fillna(method='ffill') # method='pad'와 같습니다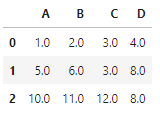
-
axis=1 : 행이 아니라 열을 사용
# 이전 열의 값으로 누락된 값을 대체 df.fillna(method='ffill', axis=1)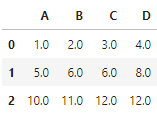
-
4.1.4 사이킷런 추정기 API 익히기
☑️사이킷런 변환기의 훈련과 변환 과정☑️
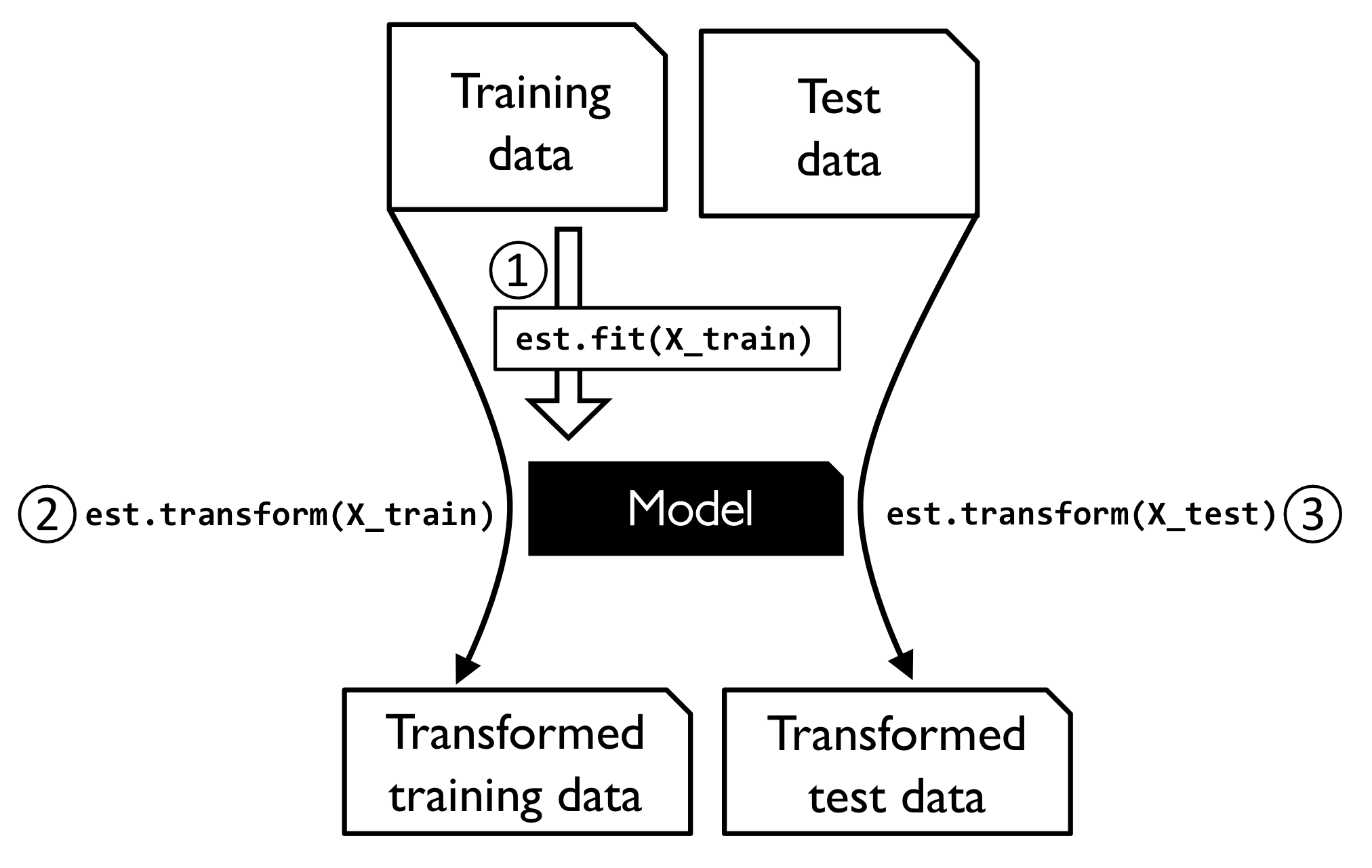
☑️사이킷런 추정기의 훈련과 예측 과정☑️
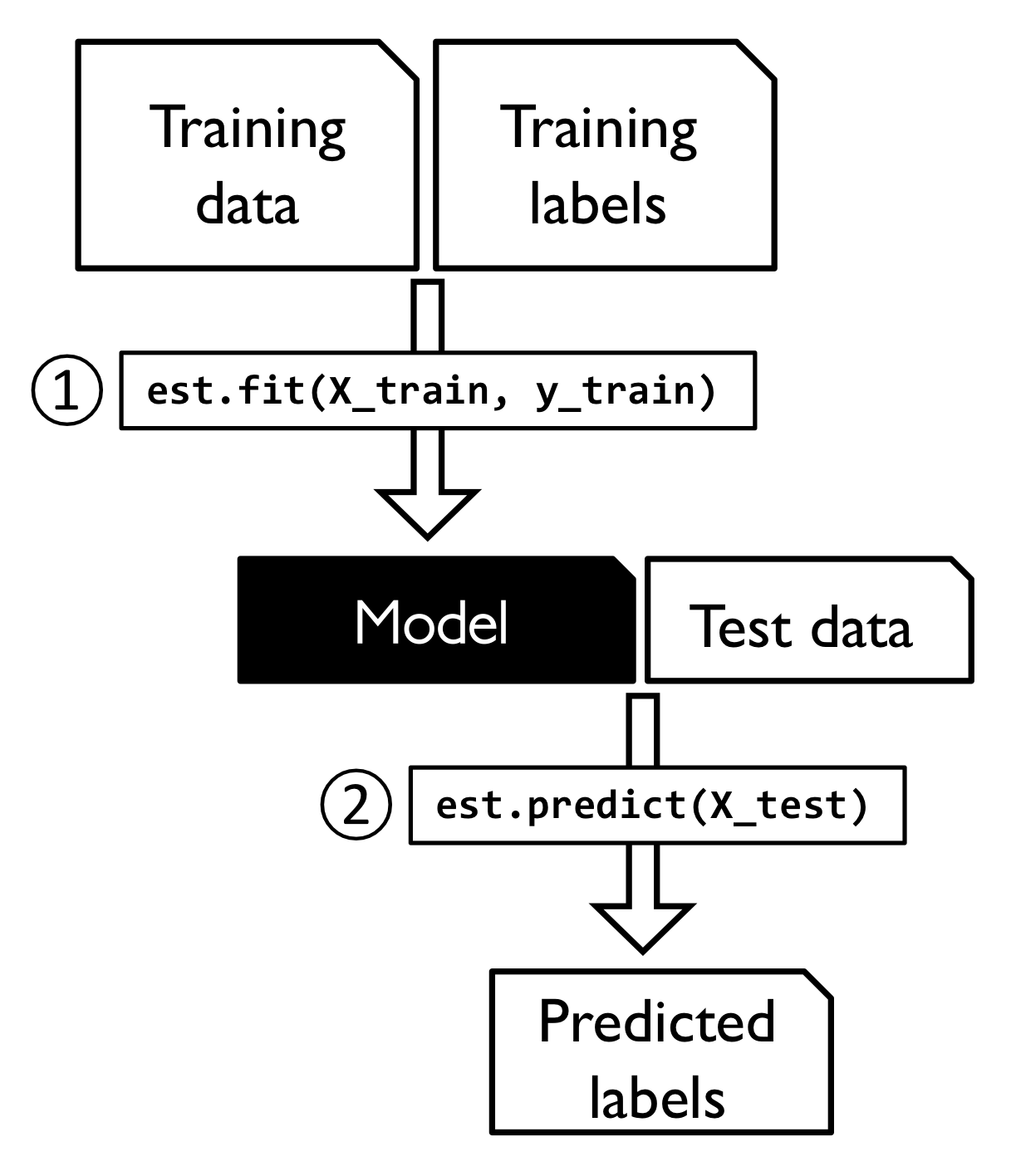
4.2 범주형 데이터 다루기
범주형 데이터 - 순서가 있는 것과 없는 것
- 순서가 있는 특성 : 차례대로 놓을 수 있는 범주형 특성
- 예 : 티셔츠 사이즈 XL > L > M
- 순서가 없는 특성 : 차례를 부여할 수 없는 범주형 특성
- 예 : 티셔츠 컬러 (순서가 ❌)
4.2.1 판다스를 사용한 범주형 데이터 인코딩
☑️범주형 데이터 인코딩☑️
import pandas as pd
df = pd.DataFrame([['green', 'M', 10.1, 'class2'],
['red', 'L', 13.5, 'class1'],
['blue', 'XL', 15.3, 'class2']])
df.columns = ['color', 'size', 'price', 'classlabel']
df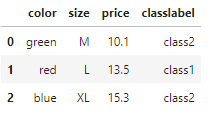
➡️ color(순서 ❌), size(순서 ⭕), price(수치형 특성), 마지막 열은 클래스 레이블
4.2.2 순서가 있는 특성 매핑
☑️범주형의 문자열 값을 정수로 바꿔주는 매핑 함수 만들기☑️
size_mapping = {'XL': 3,
'L': 2,
'M': 1}
df['size'] = df['size'].map(size_mapping)
df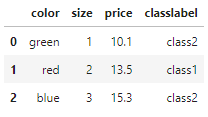
☑️정수 값을 다시 문자열 표현으로 바꾸는 매핑 딕셔너리 정의☑️
inv_size_mapping = {v: k for k, v in size_mapping.items()}
df['size'].map(inv_size_mapping)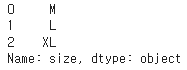
4.2.3 클래스 레이블 인코딩
☑️클래스 레이블을 정수로 바꾸기 위해 매핑 딕셔너리 생성☑️
- enumerate : 반복 가능한 객체를 입력으로 받아 인덱스와 값의 튜플을 차례대로 반환하는 파이썬 내장 함수
import numpy as np
# 클래스 레이블을 문자열에서 정수로 바꾸기 위해
# 매핑 딕셔너리를 만듭니다
class_mapping = {label: idx for idx, label in enumerate(np.unique(df['classlabel']))}
class_mapping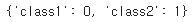
☑️매핑 딕셔너리로 클래스 레이블을 정수로 변환☑️
# 클래스 레이블을 문자열에서 정수로 바꿉니다
df['classlabel'] = df['classlabel'].map(class_mapping)
df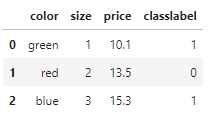
☑️클래스 레이블을 원본 문자열로 매핑☑️
# 클래스 레이블을 거꾸로 매핑합니다
inv_class_mapping = {v: k for k, v in class_mapping.items()}
df['classlabel'] = df['classlabel'].map(inv_class_mapping)
df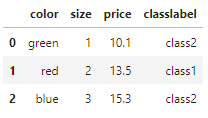
☑️사이킷런 LabelEncoder 클래스로 레이블 인코딩☑️
- fit_transform : fit 메서드와 transform 메서드를 합쳐 놓은 단축 메서드
from sklearn.preprocessing import LabelEncoder
# 사이킷런의 LabelEncoder을 사용한 레이블 인코딩
class_le = LabelEncoder()
y = class_le.fit_transform(df['classlabel'].values)
y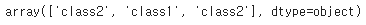
☑️클래스 레이블을 원본 문자열로 매핑☑️
- inverse_transform : 정수 클래스 레이블을 원본 문자열 형태로 되돌림
# 거꾸로 매핑
class_le.inverse_transform(y)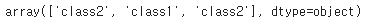
4.2.4 순서가 없는 특성에 원-핫 인코딩 적용
☑️순서가 없는 특성 문자열 레이블 인코딩☑️
X = df[['color', 'size', 'price']].values
color_le = LabelEncoder()
X[:, 0] = color_le.fit_transform(X[:, 0])
X
원-핫 인코딩 기법 : 순서 없는 특성에 들어 있는 고유한 값마다 새로운 더미 특성을 만드는 것
☑️사이킷런으로 원-핫 인코딩 수행☑️
- One HotEncoder : 원-핫 인코딩을 수행해줌
from sklearn.preprocessing import OneHotEncoder
X = df[['color', 'size', 'price']].values
color_ohe = OneHotEncoder()
color_ohe.fit_transform(X[:, 0].reshape(-1, 1)).toarray()
☑️ColumnTransformer를 사용하여 변환☑️
- ColumnTransformer : 여러 개의 특성이 있는 배열에서 특정 열만 변환
- passthrough : 변경하지 않는 열에 지정하면 변환하지 않음
- dtype=np.int : 정수로 원-핫 인코딩
from sklearn.compose import ColumnTransformer
X = df[['color', 'size', 'price']].values
c_transf = ColumnTransformer([ ('onehot', OneHotEncoder(dtype=np.int), [0]),
('nothing', 'passthrough', [1, 2])])
c_transf.fit_transform(X)
☑️판다스로 원-핫 인코딩 더미 변수 만들기☑️
- get_dummies 메서드 : 문자열 열만 변환하고 나머지 열은 그대로 둠
# 원-핫 인코딩 via 판다스
pd.get_dummies(df[['price', 'color', 'size']])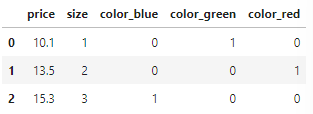
- drop_first=True : 첫 번째 열을 삭제
# get_dummies에서 다중 공선성 문제 처리
pd.get_dummies(df[['price', 'color', 'size']], drop_first=True)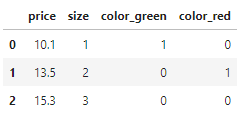
- drop='first' : 인코딩된 특성 중 첫 번째 열을 삭제
- categories='auto' : 중복된 열을 삭제
# OneHotEncoder에서 다중 공선성 문제 처리 (중복된 열 삭제)
color_ohe = OneHotEncoder(categories='auto', drop='first')
c_transf = ColumnTransformer([ ('onehot', color_ohe, [0]),
('nothing', 'passthrough', [1, 2])])
c_transf.fit_transform(X)
4.3 데이터셋을 훈련 데이터셋과 테스트 데이터셋으로 나누기
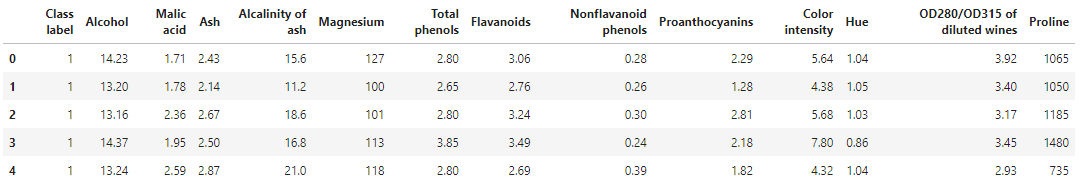
Wine 데이터셋 - 178개의 와인 샘플과 여러 가지 화학 성분을 나타내는 13개의 특성으로 구성
☑️Wine 데이터셋 읽어들이기☑️
df_wine = pd.read_csv('https://archive.ics.uci.edu/'
'ml/machine-learning-databases/wine/wine.data',
header=None)
# UCI 머신러닝 저장소의 Wine 데이터셋에 접근되지 않을 때
# 다음 코드의 주석을 제거하고 로컬 경로에서 데이터셋을 읽으세요:
# df_wine = pd.read_csv('wine.data', header=None)
df_wine.columns = ['Class label', 'Alcohol', 'Malic acid', 'Ash',
'Alcalinity of ash', 'Magnesium', 'Total phenols',
'Flavanoids', 'Nonflavanoid phenols', 'Proanthocyanins',
'Color intensity', 'Hue', 'OD280/OD315 of diluted wines',
'Proline']
print('Class labels', np.unique(df_wine['Class label']))
df_wine.head()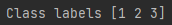
☑️데이터셋을 훈련 데이터셋과 테스트 데이터셋으로 나누기☑️
- 변수 X : 인덱스 1에서 인덱스 13까지 넘파이 배열로 변환해서 할당
- 변수 y : 첫 번째 열의 클래스 레이블
- train_test_split 함수 : X와 y를 랜덤하게 훈련 데이터셋과 테스트 데이터셋으로 분할
- test_size=0.3 : 와인 샘플의 30%가 X_test와 y_test에 할당 (나머지 샘플 70%는 X_train과 y_train에 각각 할당)
- stratify=y : 훈련 데이터셋과 테스트 데이터셋이 있는 클래스 비율이 원본 데이터셋과 동일하게 유지됨
from sklearn.model_selection import train_test_split
X, y = df_wine.iloc[:, 1:].values, df_wine.iloc[:, 0].values
X_train, X_test, y_train, y_test =\
train_test_split(X, y,
test_size=0.3,
random_state=0,
stratify=y)4.4 특성 스케일 맞추기
정규화 : 특성의 스케일을 [0, 1] 범위에 맞추는 것
-
최소-최대 스케일 변환 : 각 특성의 열마다 최소-최대 스케일 변환을 적용하여 새로운 값을 계산 (정해진 범위의 값이 필요할 때 유용하게 사용)
☑️ 사이킷런으로 최소-최대 스케일 변환 수행☑️
from sklearn.preprocessing import MinMaxScaler
mms = MinMaxScaler()
X_train_norm = mms.fit_transform(X_train)
X_test_norm = mms.transform(X_test)표준화 : 특성의 평균을 0에 맞추고 표준 편차를 1로 만들어 정규 분포와 같은 특징을 가지도록 만듦 -> 가중치를 더 쉽게 학습할 수 있도록 만듦
☑️사이킷런으로 표준화 수행☑️
- StandardScaler : 표준화를 위한 클래스
from sklearn.preprocessing import StandardScaler
stdsc = StandardScaler()
X_train_std = stdsc.fit_transform(X_train)
X_test_std = stdsc.transform(X_test)- RobustScaler : 특성 열마다 독립적으로 작용하며 중간 값을 뺀 다음 데이터셋의 1사분위수와 3사분위수(즉, 25백분위수와 75백분위수)를 사용해서 데이터셋의 스케일을 조정
4.5 유용한 특성 선택
4.5.1 모델 복잡도 제한을 위한 L1 규제와 L2 규제
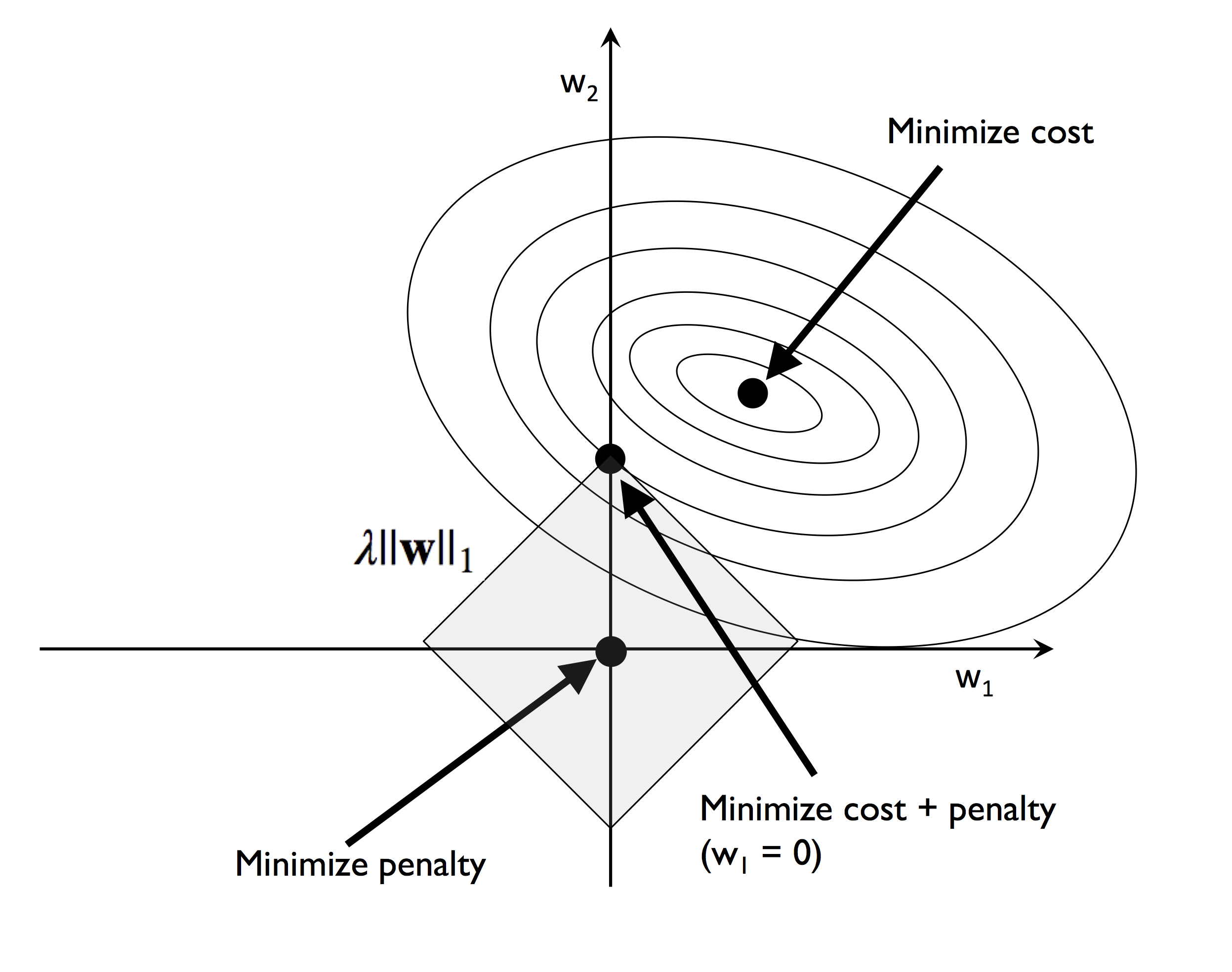
L1 규제 : 가중치 제곱을 가중치 절댓값으로 바꾼 것 -> 대부분의 특성 가중치가 0이 됨
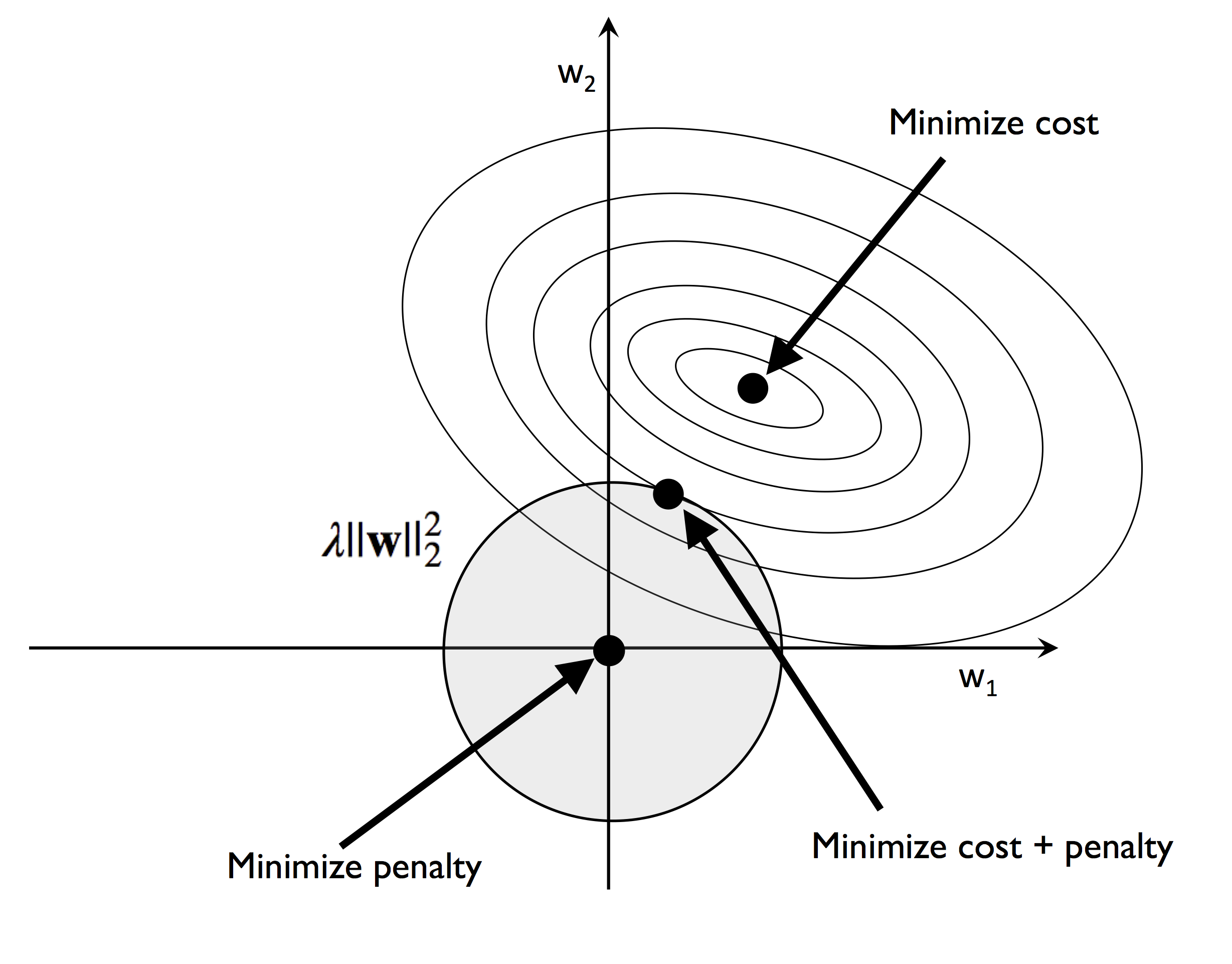
4.5.2 L2 규제의 기하학적 해석
L2 규제 : 비용 함수에 페널티 항을 추가 -> 가중치 값을 아주 작게 만드는 효과
4.5.3 L1 규제를 사용한 희소성
☑️사이킷런으로 L1 규제를 지원하는 모델 만들기☑️
- penalty='l1' : L1 규제를 지원하게 만듦
from sklearn.linear_model import LogisticRegression
LogisticRegression(penalty='l1', solver='liblinear')☑️표준화 전처리된 Wine 데이터에 L1 규제가 있는 로지스틱 회귀 적용☑️
from sklearn.linear_model import LogisticRegression
lr = LogisticRegression(penalty='l1', C=1.0, solver='liblinear', random_state=1)
# C=1.0이 기본입니다.
# 규제 효과를 높이거나 낮추려면 C 값을 증가시키거나 감소시킵니다.
lr.fit(X_train_std, y_train)
print('훈련 정확도:', lr.score(X_train_std, y_train))
print('테스트 정확도:', lr.score(X_test_std, y_test))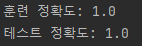
➡️ 훈련과 테스트 정확도(둘 다 100%)를 보면 모델이 두 데이터셋에 완벽하게 작동함을 알 수 있음
☑️규제 강도를 달리하여 특성의 가중치 변화를 그래프로 그려보기☑️
import matplotlib.pyplot as plt
fig = plt.figure()
ax = plt.subplot(111)
colors = ['blue', 'green', 'red', 'cyan',
'magenta', 'yellow', 'black',
'pink', 'lightgreen', 'lightblue',
'gray', 'indigo', 'orange']
weights, params = [], []
for c in np.arange(-4., 6.):
lr = LogisticRegression(penalty='l1', C=10.**c, solver='liblinear',
multi_class='ovr', random_state=0)
lr.fit(X_train_std, y_train)
weights.append(lr.coef_[1])
params.append(10**c)
weights = np.array(weights)
for column, color in zip(range(weights.shape[1]), colors):
plt.plot(params, weights[:, column],
label=df_wine.columns[column + 1],
color=color)
plt.axhline(0, color='black', linestyle='--', linewidth=3)
plt.xlim([10**(-5), 10**5])
plt.ylabel('weight coefficient')
plt.xlabel('C')
plt.xscale('log')
plt.legend(loc='upper left')
ax.legend(loc='upper center',
bbox_to_anchor=(1.38, 1.03),
ncol=1, fancybox=True)
plt.show()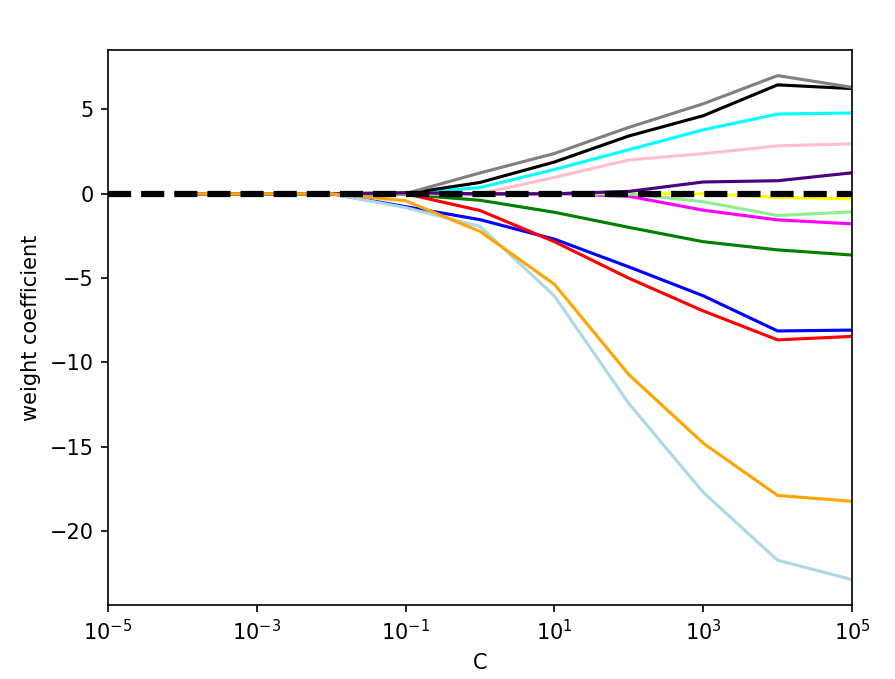
➡️강한 규제 파라미터(C < 0.1)로 모델을 제약하면 모든 가중치가 0이 됨
4.5.4 순차 특성 선택 알고리즘
차원 축소 - 모델 복잡도를 줄이고, 과대적합을 피함 -> 규제가 없는 모델에서 특히 유용
📍주요 카테고리📍
1) 특성 선택 : 원본 특성에서 일부를 선택
- 순차 특성 선택 : 탐욕적 탐색 알고리즘으로 초기 d 차원의 특성 공간을 k < d인 k 차원의 특성 부분 공간으로 축소
- 순차 후진 선택(SBS) : 계산 효율성을 향상하기 위해 모델 성능을 가능한 적게 희생하면서 초기 특성의 부분 공간으로 차원을 축소
- 단계
- 알고리즘을 k=d로 초기화. d는 전체 특성 공간의 차원
- 조건을 최대화하는 특성을 결정
- 특성 집합에서 조건을 최대화하는 특성을 제거
- k가 목표하는 특성 개수가 되면 종료. 아니면 단계 2로 돌아가기 - SBS 파이썬으로 직접 구현
- 단계
- 순차 후진 선택(SBS) : 계산 효율성을 향상하기 위해 모델 성능을 가능한 적게 희생하면서 초기 특성의 부분 공간으로 차원을 축소
from sklearn.base import clone
from itertools import combinations
import numpy as np
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
class SBS():
def __init__(self, estimator, k_features, scoring=accuracy_score,
test_size=0.25, random_state=1):
self.scoring = scoring
self.estimator = clone(estimator)
self.k_features = k_features
self.test_size = test_size
self.random_state = random_state
def fit(self, X, y):
X_train, X_test, y_train, y_test = \
train_test_split(X, y, test_size=self.test_size,
random_state=self.random_state)
dim = X_train.shape[1]
self.indices_ = tuple(range(dim))
self.subsets_ = [self.indices_]
score = self._calc_score(X_train, y_train,
X_test, y_test, self.indices_)
self.scores_ = [score]
while dim > self.k_features:
scores = []
subsets = []
for p in combinations(self.indices_, r=dim - 1):
score = self._calc_score(X_train, y_train,
X_test, y_test, p)
scores.append(score)
subsets.append(p)
best = np.argmax(scores)
self.indices_ = subsets[best]
self.subsets_.append(self.indices_)
dim -= 1
self.scores_.append(scores[best])
self.k_score_ = self.scores_[-1]
return self
def transform(self, X):
return X[:, self.indices_]
def _calc_score(self, X_train, y_train, X_test, y_test, indices):
self.estimator.fit(X_train[:, indices], y_train)
y_pred = self.estimator.predict(X_test[:, indices])
score = self.scoring(y_test, y_pred)
return score ☑️사이킷런의 KNN 분류기를 사용하여 SBS 구현 동작 테스트☑️import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors=5)
# 특성을 선택합니다
sbs = SBS(knn, k_features=1)
sbs.fit(X_train_std, y_train)
# 선택한 특성의 성능을 출력합니다
k_feat = [len(k) for k in sbs.subsets_]
plt.plot(k_feat, sbs.scores_, marker='o')
plt.ylim([0.7, 1.02])
plt.ylabel('Accuracy')
plt.xlabel('Number of features')
plt.grid()
plt.tight_layout()
plt.show() ☑️검증 데이터셋에서 계산한 KNN 분류기의 정확도 그리기☑️import matplotlib.pyplot as plt
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors=5)
# 특성을 선택합니다
sbs = SBS(knn, k_features=1)
sbs.fit(X_train_std, y_train)
# 선택한 특성의 성능을 출력합니다
k_feat = [len(k) for k in sbs.subsets_]
plt.plot(k_feat, sbs.scores_, marker='o')
plt.ylim([0.7, 1.02])
plt.ylabel('Accuracy')
plt.xlabel('Number of features')
plt.grid()
plt.tight_layout()
plt.show()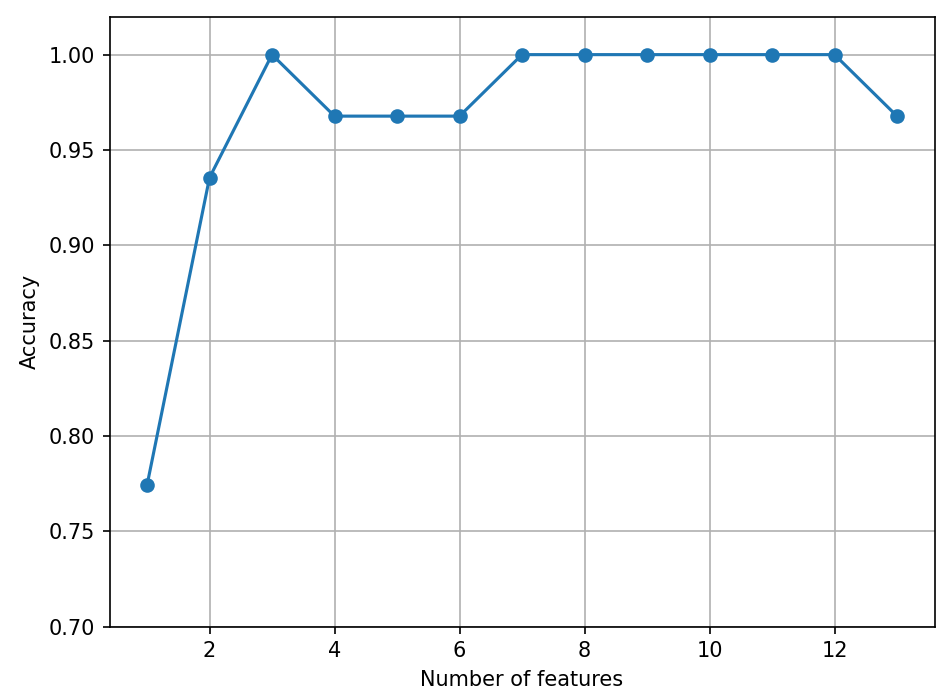
➡️ 특성 개수를 줄여서 KNN 모델의 성능이 증가하지는 ❌ but, 데이터셋 크기를 줄였고, 더 간단한 모델을 얻었고, 해석하기도 쉬워짐
2) 특성 추출 : 일련의 특성에서 얻은 정보로 새로운 특성을 만듦
4.6 랜덤 포레스트의 특성 중요도 사용
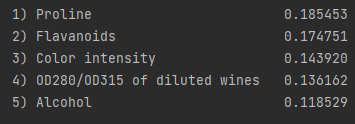
☑️ 랜덤 포레스트 모델의 특성 중요도 출력하기☑️
from sklearn.ensemble import RandomForestClassifier
feat_labels = df_wine.columns[1:]
forest = RandomForestClassifier(n_estimators=500,
random_state=1)
forest.fit(X_train, y_train)
importances = forest.feature_importances_
indices = np.argsort(importances)[::-1]
for f in range(X_train.shape[1]):
print("%2d) %-*s %f" % (f + 1, 30,
feat_labels[indices[f]],
importances[indices[f]]))
plt.title('Feature Importance')
plt.bar(range(X_train.shape[1]),
importances[indices],
align='center')
plt.xticks(range(X_train.shape[1]),
feat_labels[indices], rotation=90)
plt.xlim([-1, X_train.shape[1]])
plt.tight_layout()
plt.show()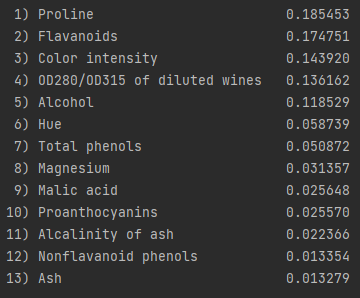
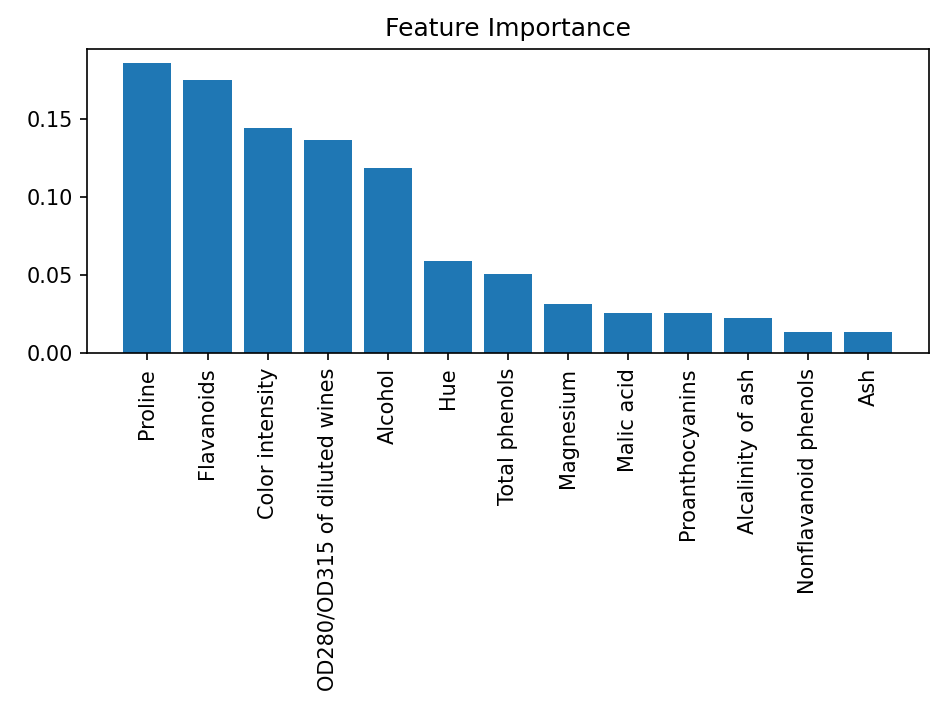
➡️ Wine 데이터셋 특성의 상대적인 중요도에 따른 순위를 그래프로 그림
➡️ 특성 중요도는 합이 1이 되도록 정규화된 값
☑️사이킷런으로 특성 중요도 사용☑️
- SelectFromModel : 모델 훈련이 끝난 후 사용자가 지정한 임계 값을 기반으로 특성을 선택
from sklearn.feature_selection import SelectFromModel
sfm = SelectFromModel(forest, threshold=0.1, prefit=True)
X_selected = sfm.transform(X_train)
print('이 임계 조건을 만족하는 샘플의 수:',
X_selected.shape[1])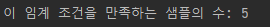
for f in range(X_selected.shape[1]):
print("%2d) %-*s %f" % (f + 1, 30,
feat_labels[indices[f]],
importances[indices[f]]))