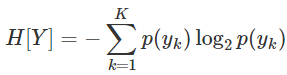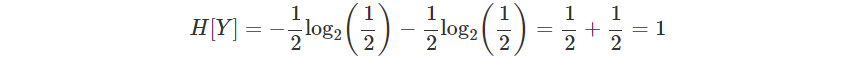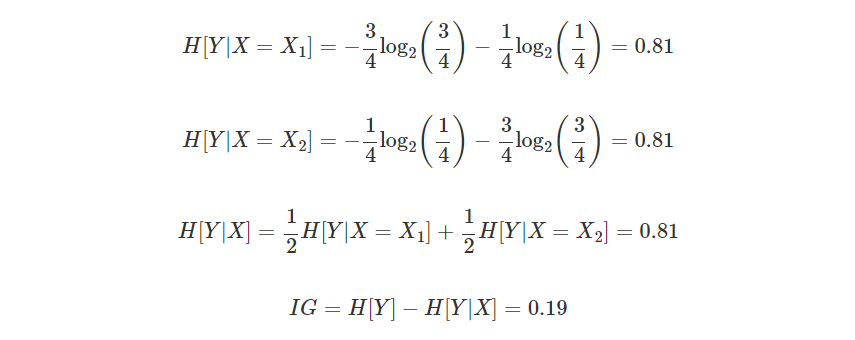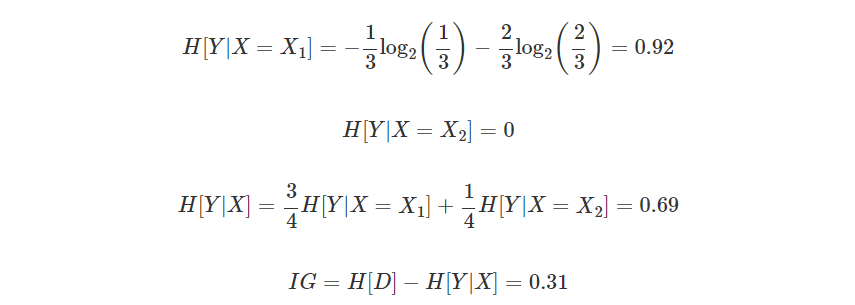1. 로지스틱 회귀분석(Logistic Regression
1. 회귀분석 예제
보스턴 집값 예측
이제 제대로 된 회귀분석에 앞서 회귀분석이 어떠한 것을 알아내는 것인지를 알기 위해 보스턴 집값 데이터를 가지고 데이터간의 상관관계를 보도록 하겠다.
from sklearn.datasets import load_boston
boston = load_boston()
- 알겠지만 여기에서 가져오는 데이터는 행렬값으로 나오지 데이터 프레임이 아니다.
- 그래서 데이터 프레임으로 바꾸어주는 과정이 필요하다.
- 그렇다고 바로 pd.DataFrame 해버리면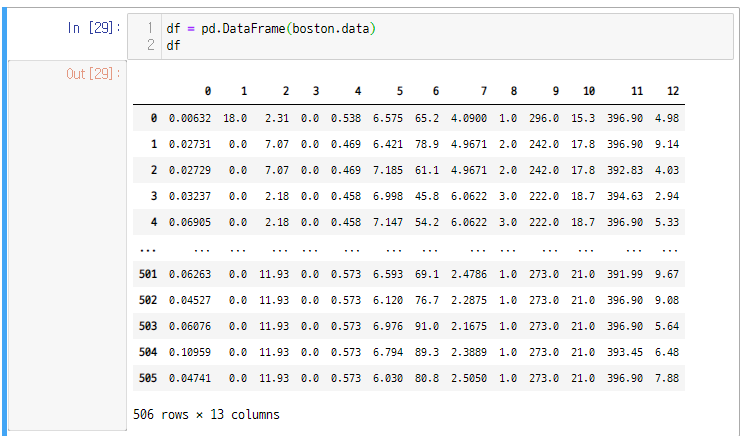
이렇게 이상하게 된다.
- 그래서 load_boston으로 받은 데이터는 x와 y로 나누어서 concat 해주는 과정이 필요하다.
- 참고로 load_boston 데이터 파일을 열어보면 data,target,feature_name의 구성을 가진다.
- 우선 feature_name은 보스턴 데이터를 그대로 사용하고
- target값은 MEDV라는 열을 새로 만든다.
dfx = pd.DataFrame(boston.data,columns=boston.feature_names)
dfy = pd.DataFrame(boston.target,columns = ["MEDV"])
- pandas의 concat을 하되 열단위로 붙여버린다.
df = pd.concat([dfx,dfy],axis=1)
df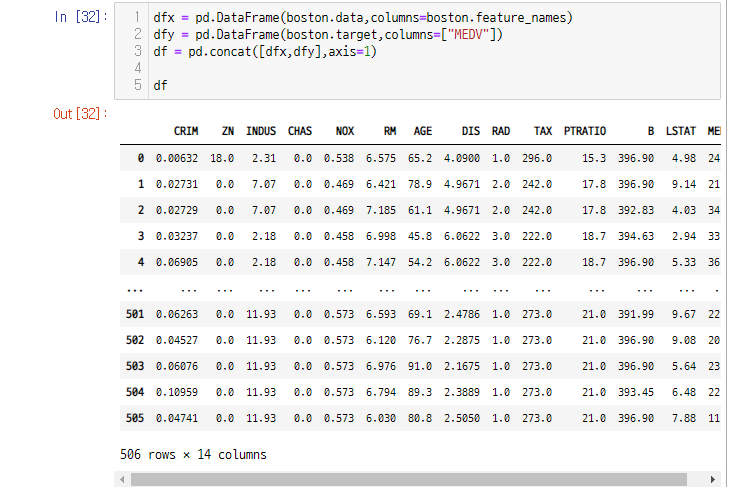
하면 이렇게 예쁜 모양의 데이터 프레임이 생성된다.
이것의 상관 관계를 알아보기 위해 스캐터 플롯을 활용하여 살펴보면
sns.pairplot(df[["MEDV","RM","AGE","CHAS"]])
plt.show()
와 같이 표현되고 이를 통해 방의 수가 증가하면 가격(MEDV)도 증가한다는 것을 볼 수 있다.
이렇게 독립변수끼리 상관관계를 가지는 것을 다중공선성(multicolinearity)라고 부른다.
- 위에서 구지 concat을 사용하기 싫다면 아래와 같이 코드를 짜도 된다.
df = pd.DataFrame(boston.value,columns=boston.feature_names)
df["MEDV"] = pd.DataFrame(boston.target)2. 가상 데이터 예측
이제 본격적으로 회귀분석을 해보도록 하자
회귀분석은 다시금 이야기 하지만 x와 y 간에 라벨링 되어 있는 데이터셋을 가지고 모델을 만들어 다른 독립변수를 그 모델에 적용하는 과정을 의미한다.
make_regression() 함수가 sklearn 에서 제공하는 함수로 이를 통해 회귀분석을 만들어준다.
x,y,w = make_regression(n_samples,n_features,n_informative,effective_rank,
bias,noise,coef=True,random_state)위와 같이 함수에 매개변수를 전달하여 주어서 x,y,w를 구할 수 있는데
n_samples는 데이터셋의 갯수로 숫자를 넣어주면 된다.
n_features는 독립변수의 수. 즉, x변수 종류의 갯수
n_informative는 변수가 서로 독립일때 어떤것만 y와 종속이지 알려주는 인자이다.
effective_rank는 변수가 서로 독립일때 어떤 것이 y와 독립인지 알려주는 인자이다.
bias는 y절편의 값
noise는 종속변수에 더해주는 잡음 e의 표준편차(실재 데이터에서 얼마나 떨어졌는지)
coef는 선형 모형의 계수도 출력인데 항상 True로 설정해두자 - 아래쪽 w이다.
random_state는 난수 발생용 변수
이때 출력은
x : 독립변수의 표본 데이터 행렬 X
y : 종속변수의 표본 데이터 벡터 y
w : 선형 모형의 계수 벡터 w, 입력 인수 coef가 True인 경우에만 출력이 된다.
최종적으로 선형회귀 분석이기 때문에 선형식이 만들어지고
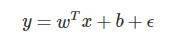
위와 같은 형태의 식이 나오게 된다.
w^T가 출력값으로 나온 것이고
b는 bias값
e는 난수이다.
실재 내부를 한번 구현해 보면
def make_regression2(n_samples,bias,noise,random_state=0):
np.random.seed(random_state)
x = np.random.normal(size=n_samples)
w = 100 * np.random.normal(size=1)[0]
y0 = w*x + bias
e = np.random.normal(scale=noise,size=n_samples)
y = y0 + e
return x,y,w위와 같은 형태를 뛸꺼 같다.
w는 랜덤으로 값을 설정해 두었지만 이는 실재 함수에서는 다르게 사용 될 것이다.
이렇게 만든것을 plt.scatter(x,y,s=100) 함수를 이용해 출력해 보면
(scatter 함수는 x,y 그리고 원의 크기를 넣는 함수이다.)

와 같이 나온다.
반대로 함수의 w값을 10정도로 바꾸면
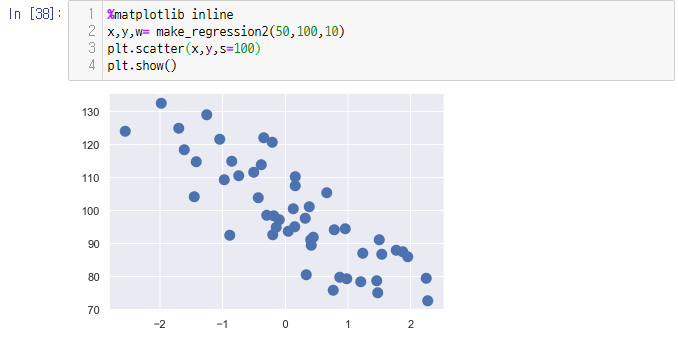
이런게 나온다. 보면 알겠지만 상관관계가 크게 바뀐것을 확인할 수 있다.
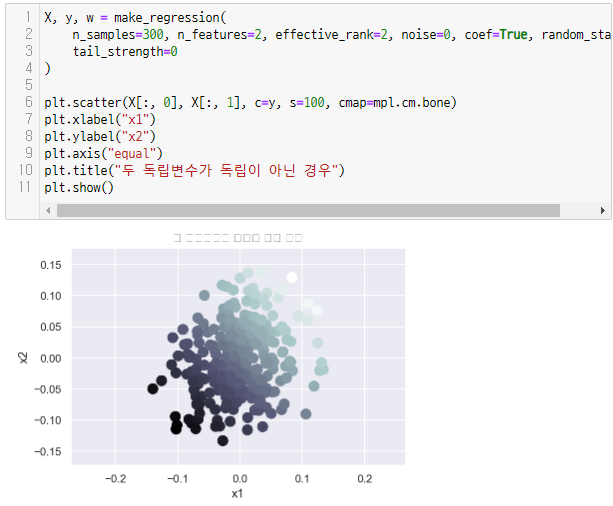
또한 n_informative나 effective_rank중 하나를 사용하여서 y와의 독립 혹은 종속 관계를 표현할 수도 있다.
X, y, w = make_regression(
n_samples=300, n_features=1, effective_rank=1, noise=0, coef=True, random_state=0,
tail_strength=0
)
plt.scatter(X[:, 0], X[:, 1], c=y, s=100, cmap=mpl.cm.bone)
plt.xlabel("x1")
plt.ylabel("x2")
plt.axis("equal")
plt.title("두 독립변수가 독립이 아닌 경우")
plt.show()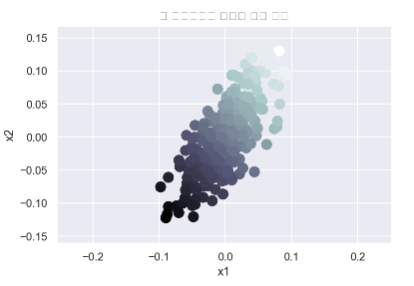
2. 의사결정나무(Decision Tree)
회귀분석 및 카테고리에도 사용 가능 그래서 CART(Classification And Regression Tree)라고 부른다.
의사결정나무를 이용한 분류법은 다음과 같다.
1. 독립변수 딱 하나를 골라 그 독립변수를 기준값(threshold)로 정한다. (분류규칙)
2. 분류규칙을 기준값으로 하여 이보다 보다 작고 크고를 가지고 데이터를 나눈다.(부모노드 - 자식노드 1,2)
3. 이를 자식노드에서도 계속 반복한다. 단, 자식 노드에 한가지 클래스의 데이터만 존재한다면 더 이상 자식 노드를 나누지 않고 중지한다.
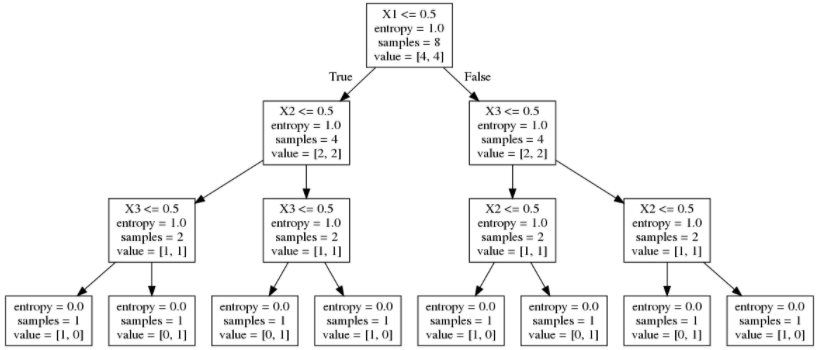
여기에서 너무 깊어지면 문제가 되기 때문에 이를 제한 할 수도 있다.
이렇게 만드는 것을 학습을 시킨다고 이야기 한다. 그리고 이렇게 만든 모델을 가지고 Xtest 데이터를 삽입하여서 확인해 본다.
의사결정나무를 사용한 분류예측
분류규칙을 정하는 방법
분류규칙을 정하는 방법은 부모 노드와 자식 노드 간의 엔트로피를 가장 낮게 만드는 최상의 독립 변수와 기준값을 찾는 것이다. 이러한 기준을 정량화한 것이 정보획득량(information gain)이다. 이에 대해서는 아래 정복획득량에서 자세하게 설명한다.
엔트로피란? 확률분포가 존재할때(H[Y]) 이 확률이 얼마나 골고루 분포하였느지 혹은 얼마나 한쪽에 집중되어 있는지를 숫자로 나타낸 거을 엔트로피라고 부른다. : 이때 퍼져있을 수록 엔트로피는 증가한거고 몰려있을 수록 엔트로피가 감소한 것으로 본다.
이떄 엔트로피는 0에서 1까지의 값으로 존재한다.
정보획득량
정복획득량을 구하기 위해서는 다음의 공식을 사용한다.
IG[Y,X]=H[Y]−H[Y|X]
정보획득량은 X라는 조건에 의해 확률 변수 Y의 엔트로피가 얼마나 감소하였는가를 나타낸다. 그렇기 때문에 원래의 Y의 엔트로피에서 X가 등장하였을 때의 Y의 조건부 엔트로피를 뺀 값으로 정의한다.
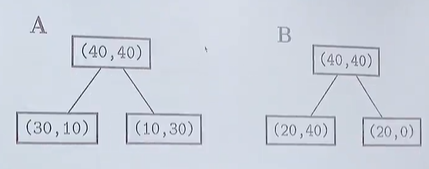
예를 들어서 위와 같이 다른 기준을 사용하는 A,B가 있다고 가정하자. 이때 어디가 엔트로피가 더 클까?
위의 엔트로피를 직접 계산해 보자
A에 대해서
B에 대해서
계산결과 B가 더 많이 엔트로피가 줄은 것을 확인할 수 있다.
이러한 엔트로피 계산을 모든 변수에 대해서 값을 나누어서 진행하여 본다.
Scikit-Learn의 의사결정나무 클래스(사용법)
sklearn에는 DecisiponTree가 존재하고 있다.
from sklearn.datasets import load_iris
data = load_iris()
y = data.target
X = data.data[:, 2:]
feature_names = data.feature_names[2:]
from sklearn.tree import DecisionTreeClassifier
tree1 = DecisionTreeClassifier(criterion='entropy', max_depth=1, random_state=0).fit(X, y)- criterion에는 entropy를 직접넣어주어야 한다. 원래 default는 gini impurity로 되어 있다. gini impurity는 엔트로피와 비슷하기는 하나 로그를 사용하지 않기 때문에 계산량이 적어서 보통은 gini impurity을 사용한다.
- max_depth : 깊이를 조절
- random_state : 사실 모든 변수에 들어간 값 전체에 대해 엔트로피를 구하지는 않는다. 너무 많아서;; 그래서 랜덤하게 잘라서 하기 때문에 random_state가 인자로 들어가 있다.
import io
import pydot
from IPython.core.display import Image
from sklearn.tree import export_graphviz
def draw_decision_tree(model):
dot_buf = io.StringIO()
export_graphviz(model, out_file=dot_buf, feature_names=feature_names)
graph = pydot.graph_from_dot_data(dot_buf.getvalue())[0]
image = graph.create_png()
return Image(image)
def plot_decision_regions(X, y, model, title):
resolution = 0.01
markers = ('s', '^', 'o')
colors = ('red', 'blue', 'lightgreen')
cmap = mpl.colors.ListedColormap(colors)
x1_min, x1_max = X[:, 0].min() - 1, X[:, 0].max() + 1
x2_min, x2_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
Z = model.predict(
np.array([xx1.ravel(), xx2.ravel()]).T).reshape(xx1.shape)
plt.contour(xx1, xx2, Z, cmap=mpl.colors.ListedColormap(['k']))
plt.contourf(xx1, xx2, Z, alpha=0.4, cmap=cmap)
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=X[y == cl, 0], y=X[y == cl, 1], alpha=0.8,
c=[cmap(idx)], marker=markers[idx], s=80, label=cl)
plt.xlabel(data.feature_names[2])
plt.ylabel(data.feature_names[3])
plt.legend(loc='upper left')
plt.title(title)
return Z- from sklearn.tree import export_graphviz : export_graphviz라는 프로그램을 이용하여 이를 출력한다. export_graphviz는 다이어그램을 그리는 프로그램으로 DOT이라는 프로그래밍 언어를 사용하여서 다이어그램을 그리어 준다.
- import pydot : export_graphviz를 위해 import pydot를 사용한다. 이 것은 decision tree에 있는 룰을 dot 언어로 바꾸어 주는 패키지이다. 그렇기 때문에 export_graphviz와 함께 사용되어야 한다.
이제 직접 의사결정나무를 그리어 보자
draw_decision_tree(tree1)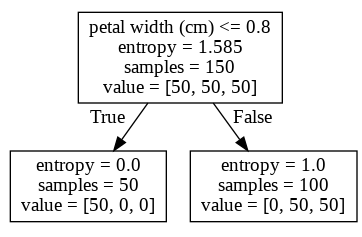
이렇게 만든 의사결정나무를 실재 그래프로 표현해 보자
%matplotlib inline
plot_decision_regions(X, y, tree1, "Depth 1")
plt.show()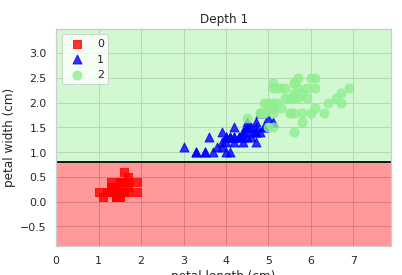
이렇게 만든 트리의 깊이를 더 높이어 보자
tree2 = DecisionTreeClassifier(
criterion='entropy', max_depth=2, random_state=0).fit(X, y)
draw_decision_tree(tree2)
plot_decision_regions(X, y, tree2, "Depth 2")
plt.show()
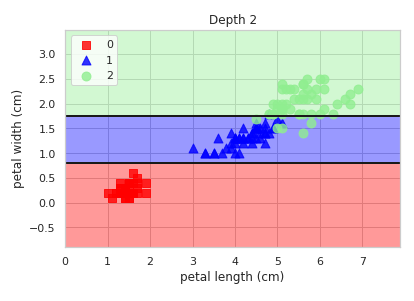
더더더 깊이를 깊게 가보자
tree3 = DecisionTreeClassifier(
criterion='entropy', max_depth=3, random_state=0).fit(X, y)
draw_decision_tree(tree3)
plot_decision_regions(X, y, tree3, "Depth 3")
plt.show()
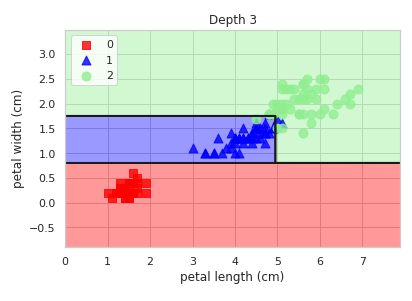
그런데 어라 이번에는 뭔가 이상하다. 그래프가 3번 깊이까지 들어갔고 4.85로 나누어졌을 텐데 어째서인지 그래프 상에는 그것이 표현되어 있지 않다. 이것을 이해하기 위해 가장 마지막 자식 노드를 다시금 봐보자. 두 자식노드에서 과반수를 차지하고 있는 것은 2번 클래스이다. 그렇기 때문에 나중에 Xtest값을 어떻게 넣든 2번 클래스로 떨어질 것이 확실하다. 그래서 그래프 상에 이를 구지 나누어 놓지 않은 오히려 나누지 못한 것이라고 보는 것이 정확하다.
더더더더 깊이를 깊게 가보자
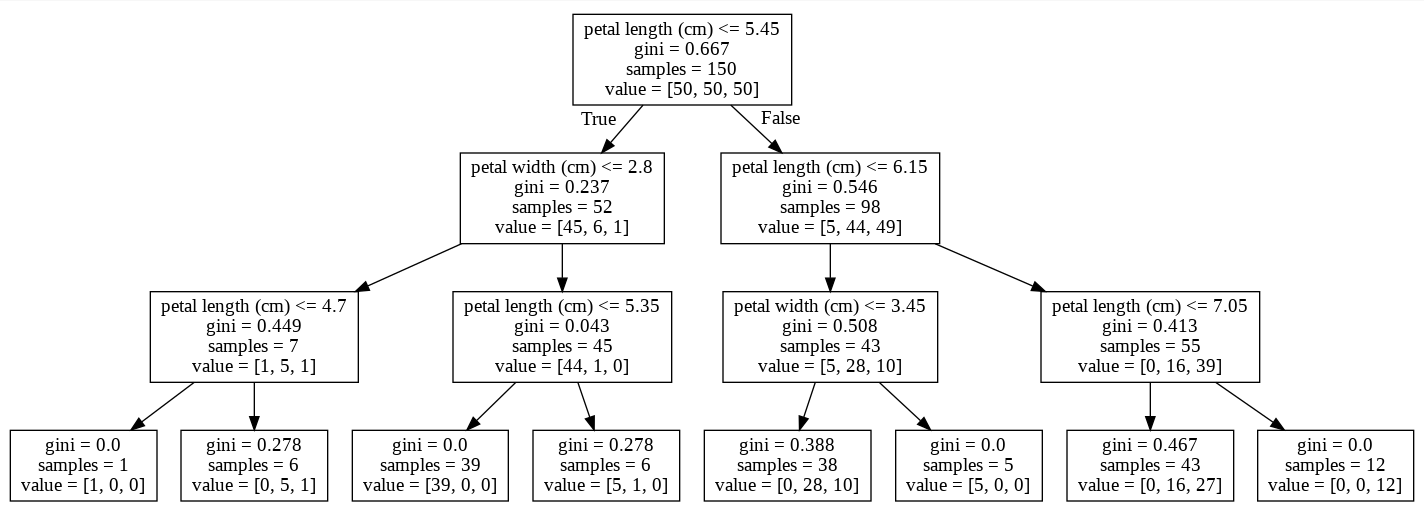
tree4 = DecisionTreeClassifier(
criterion='entropy', max_depth=4, random_state=0).fit(X, y)
draw_decision_tree(tree4)
plot_decision_regions(X, y, tree4, "Depth 4")
plt.show()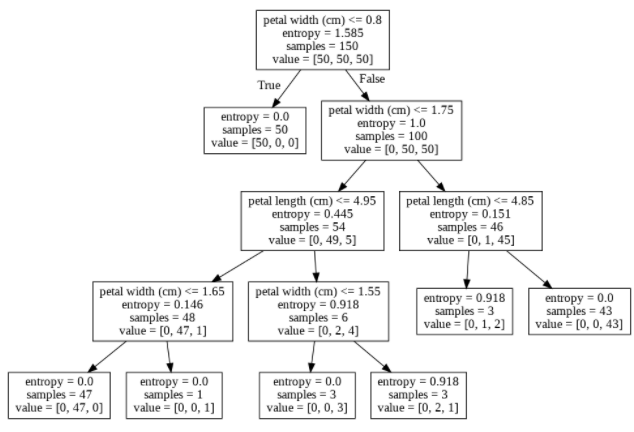
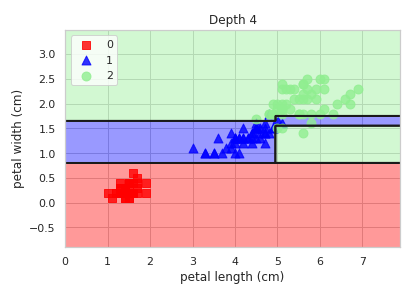
이번에는 decision_tree가 나온 결과가 이상하다. 가장 오른쪽에서 두번째 자식노드와 같은 경우 [0, 1, 2] 인데도 분해가 되지 않았다. 이것은 1번 클래스가 1개이기 때문에 생기는 결과이다. 더 이상 잘개 분해할 수 없는 요소가 존재하기 때문에 더 이상 진행되지 않은 것이다.
연습 문제 1
- 붓꽃 분류 문제에서 꽃받침의 길이와 폭(sepal length, sepal width)을 사용하여 max_depth=3인 의사결정나무 모형을 만들고 정확도(accuracy)를 계산하라.
- K=5 인 교차 검증을 통해 테스트 성능 평균을 측정하라.
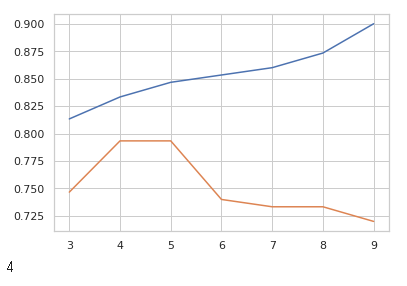
- max_depth 인수를 바꾸어 가면서 테스트 성능 평균을 구하여 cross validation curve를 그리고 가장 테스트 성능 평균이 좋은 max_depth 인수를 찾아라.
## 1번
from sklearn.datasets import load_iris
iris = load_iris()
x1 = iris.data[:,:2]
y1=iris.target
from sklearn.tree import DecisionTreeClassifier
model1 = DecisionTreeClassifier(max_depth=3).fit(x1,y1)
y1_pred = model1.predict(x1)
from sklearn.metrics import accuracy_score
accuracy_score(y1,y1_pred)

## 2번
from sklearn.model_selection import cross_val_score
cross_val_score(model1,x1,y1,scoring="accuracy",cv=5).mean()
## 3번
acc_test=[]
cross_test=[]
best_depth = 1
pre_cross=0
for max_depth in range(3,10):
model1 = DecisionTreeClassifier(max_depth=max_depth).fit(x1,y1)
acc_test.append(accuracy_score(y1,model1.predict(x1)))
cross_test.append(cross_val_score(model1,x1,y1,scoring="accuracy",cv=5).mean())
if(cross_test[max_depth-3]>pre_cross):
pre_cross=cross_test[max_depth-3]
best_depth=max_depth
plt.plot(np.arange(3,10),acc_test)
plt.plot(np.arange(3,10),cross_test)
plt.show()
print(best_depth)