마스크 배열이란
유효하지 않은 데이터가 많을 경우 데이터셋이 불완전할 수 있다.
예를 들면, 센서가 데이터를 기록하지 못하거나 유효하지 않은 값을 기록하는 등이 있다.
numpy.ma 모듈은 마스크 배열을 도입하여 이런 문제를 해결할 편리한 방법을 제시한다.
마스크 배열은 표준 numpy.ndarray 와 마스크의 조합이다.
마스크는 nomask 이며 연관된 배열의 값이 유효하지 않음을 나타내거나 유효한지 여부를 판별하는 부울 배열이다.
마스크 요소가 False 이면 연관된 배열의 해당 요소가 유효하며 마스크 해제된 것으로 간주된다.
True 면 관련 배열의 요소가 마스킹(유효하지 않음)된 것으로 간주된다.
MaskedArray 는 다음의 조합으로 생각할 수 있다.
- 모든 모양 도는 데이터 유형의 일반
numpy.ndarray로서의 데이터 - 데이터와 모양이 같은 부울 마스크
fill_value를 표준numpy.ndarray를 반환하기 위한 잘못된 항목을 대체할 수 있는 값
언제 유용할까?
마스킹된 배열이 유효하지 않은 항목을 제거하는 것보다 더 유용한 경우가 있다.
- 배열을 복사하지 않고 나중에 처리하기 위해 마스크 한 값을 보존하려는 경우
- 각 고유한 마스크의 여러 배열을 처리해야 하는 경우. 이때 버그를 방지하고 코드가 더 간결해진다.
- 누락되거나 유효하지 않은 값에 대한 다른 플래그가 있고 원래 데이터 세스에서 대체하지 않고 보존하고 싶지만 계산에서만 제외하고 싶은 경우
- 누락된 값을 피하거나 제거할 수 없지만 작업에서
NaN값을 처리하고 싶지 않은 경우
nunpy.ma 모듈에는 대부분의 NumPy 범용 함수의 특정 구현이 함께 제공되어 마스킹된 배열도 좋은 방법이다.
즉, 마스킹된 데이터에 빠른 벡터화된 함수와 연산을 계속 적용할 수 있다.
아래에 실제로 어떻게 작용되는지 살펴보자.
마스킹된 배열을 사용한 코로나 데이터 조회
코로나에 대한 데이터와 데이터 세트는 Kaggle 로부터 who_covid_19_sit_rep_time_series.csv 파일을 다운로드 받을 수 있다.
import numpy as np
import os
filepath = os.getcwd()
filename = os.path.join(filepath, "who_covid_19_sit_rep_time_series.csv")데이터 파일은 서로 다른 타입의 데이터를 포함하며 다음과 같이 조직화되어 있다.
- 첫 번째 행은 아래 행에 있는 각 열의 데이터를 설명하는 헤더 라인이며 네 번째 열에서 시작하는 헤더는 날짜이다.
- 두 번째부터 일곱 번째 행에 검토할 데이터와 다른 유형의 요약 데이터가 포함되어 있으니 작업할 데이터에 해당 데이터를 제외해야 한다.
- 작업하려는 숫자 데이터는 4열, 8행에서 시작하여 가장 오른쪽 열과 가장 아래 행까지 확장된다.
14 동안의 파일 내의 데이터를 살펴보자. csv 파일의 데이터를 수집하기 위해 numpy.genfromtxt 함수를 사용하여 위치 데이터를 포함하는 처음 세 열 대신 실제 숫자가 있는 열만 선택한다.
또한 파일의 처음 7개 행에는 관심이 없는 다른 데이터가 포함되어 있으니 건너 뛰어 준다.
# skip_header 와 usecols 로 데이터 파일을 각 변수에 추가한다.
# 첫 번째 행에서 3-7 열의 날짜만 읽는다.
dates = np.genfromtxt(filename, dtype=np.unicode_, delimiter=",",
max_rows=1, usecols=range(3, 17),
encoding="utf-8-sig")
# 처음 두 컬럼으로 부터 위치 이름을 읽는다.
# 처음 7 행은 생략한다.
locations = np.genfromtxt(filename, dtype=np.unicode_, delimiter=",",
skip_header=7, usecols=(0, 1),
encoding="utf-8-sig")
# 처음 14일 동안의 숫자 데이터를 읽는다.
nbcases = np.genfromtxt(filename, dtype=np.int_, delimiter=",",
skip_header=7, usecols=range(3, 17),
encoding="utf-8-sig")numpy.genfromtxt 함수 호출에 포함된 데이터의 각 하위 집합에 대해 numpy.dtype 을 선택하자.
또한 인코딩 파일에 대해 encoding 인수에 utf-8-sig 를 사용한다.
데이터 탐색
우선, 전체 데이터 세트를 플로팅하고 어떻게 생겼는지 살펴보자.
읽을 수 있는 플롯을 얻기 위해 x-axis ticks 에 표시할 날짜 중 몇 개만 선택하자. 또한 plot 명령에서 nbcases.T 를 사용한다.
이는 파일의 각 행을 별도의 줄로 플로팅한다는 것을 의미하기 때문이다.
-- 선 스타일을 사용하여 점선을 플로팅하도록 선택한다.
import matplotlib.pyplot as plt
selected_dates = [0, 3, 11, 13]
plt.plot(dates, nbcases.T, '--')
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases")
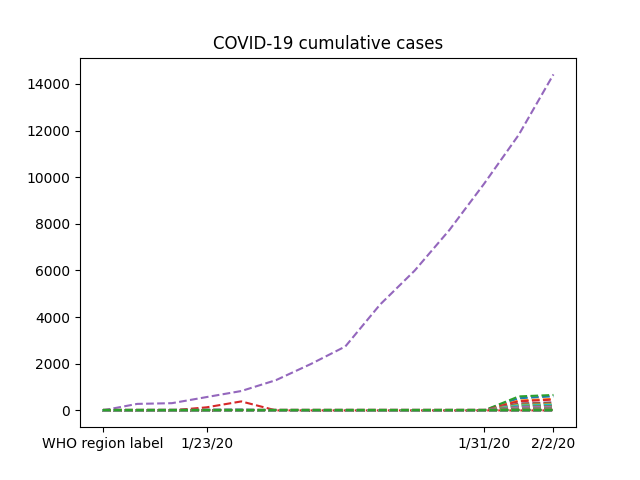
그래프는 1월 24일부터 2월 1일까지의 모양이다.
파일에서 추출한 배열을 보면 첫 번째 열에 지역이 포함, 두 번째 열에는 국가가 포함되어 있다.
하지만 첫 번째 열에 몇몇은 국가 데이터(중국의 지방 이름)를 포함하고 있다.
따라서 두 번째 항목이 중국의 모든 데이터를 단일 행으로 그룹화하는 것이 가장 합리적이다.
이를 위해 두 번째 항목이 중국에 해당하는 행만 배열에서 선택하여 numpy.sum 함수를 사용하여 선택한 모든 행을 합산한다. locations.csvnbcaseslocationsaxis=0
china_total = nbcases[locations[:, 1] == 'China'].sum(axis=0)
print(china_total)
array([ 247, 288, 556, 817, -22, -22, -15, -10, -9,
-7, -4, 11820, 14410, 17237])근데 위 출력된 china_total 을 살펴보면 음수가 존재한다.
누락된 데이터
데이터를 보면 누락된 기간이 있다.
print(nbcases)
array([[ 258, 270, 375, ..., 7153, 9074, 11177],
[ 14, 17, 26, ..., 520, 604, 683],
[ -1, 1, 1, ..., 422, 493, 566],
...,
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1],
[ -1, -1, -1, ..., -1, -1, -1]])모든 값은 원래 파일에서 누락된 데이터를 읽으려고 시도하는 numpy.genfromtxt 에서 비롯된다. 물론 누락된 데이터를 분석에 방해되지 않도록 건너 뛰고자 하는 것은 아니다.
이번엔 numpy.ma 모듈을 가져온 후 잘못된 값을 마스킹하는 새 배열을 만들어보자.
from numpy import ma
nbcases_ma = ma.masked_value(nbcases, -1)마스크된 배열은 다음과 같다.
print(nbcases_ma)
masked_array(
data=[[258, 270, 375, ..., 7153, 9074, 11177],
[14, 17, 26, ..., 520, 604, 683],
[--, 1, 1, ..., 422, 493, 566],
...,
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --],
[--, --, --, ..., --, --, --]],
mask=[[False, False, False, ..., False, False, False],
[False, False, False, ..., False, False, False],
[ True, False, False, ..., False, False, False],
...,
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True],
[ True, True, True, ..., True, True, True]],
fill_value=-1)보는 바와 같이 세 가지 속성을 갖가 있으며 잘못된 데이터에 해당하는 요소에 대한 값이 있는 것을 볼 수 있다.
누락된 데이터를 보다 자세히 볼 수 있도록 첫 번째 행(중국 후베이성)을 제외하는 데이터 모양을 살펴보자.
plt.plot(dates, nbcases_ma[1:].T, '--');
plt.xticks(selected_dates, dates[selected_dates]);
plt.title('COVID-19 cumulative cases");
이제 데이터가 가려졌으니 중국의 모든 사례를 요약해보자.
china_masked = nbcases_ma[locations[:, 1] == 'China'].sum(axis=0)
print(china_masked)
masked_array(data=[278, 309, 574, 835, 10, 10, 17, 22, 23, 25, 28, 11821,
14411, 17238],
mask=[False, False, False, False, False, False, False, False,
False, False, False, False, False, False],
fill_value=999999)마스크된 배열이므로 일반 NumPy 배열과 다른 구조이다.
이제 특성을 사용하여 데이터에 직접 액세스할 수 있다.
china_total = china_masked.data
print(china_total)
array([ 278, 309, 574, 835, 10, 10, 17, 22, 23,
25, 28, 11821, 14411, 17238])위 누적되는 값은 835 에서 10으로 내려가는 것을 볼 수 있다.
이는 중국의 지정되지 않은 지역에 유효한 데이터가 있음을 알 수 있다.
먼저, 중국 본토의 위치 지수를 확인한다.
china_mask = ((locations[:, 1] == 'China') &
(locations[:, 0] != 'Hong Kong') &
(locations[:, 0] != 'Taiwan') &
(locations[:, 0] != 'Macau') &
(locations[:, 0] != 'Unspecified*'))지금 부울 값의 배열인데 마스크된 배열에 대한 ma.nonzero 메소드를 사용하여 원하는 지수를 확인할 수 있다.
print(china_mask.nonzero())
(array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19, 20, 21, 22, 23, 25, 26, 27, 28, 29, 31, 33]),)이제 정확하게 중국 본토에 대해 합계를 구할 수 있다.
china_total = nbcases_ma[china_mask].sum(axis=0)
print(china_total)
masked_array(data=[278, 308, 440, 446, --, --, --, --, --, --, --, 11791,
14380, 17205],
mask=[False, False, False, False, True, True, True, True,
True, True, True, False, False, False],
fill_value=999999)이 정보로 데이터를 대체하고 중국 본토에 초점을 맞추어 새 그래프를 플롯할 수 있다.
plt.plot(dates, china_total.T, '--');
plt.xticks(selected_dates, dates[selected_dates]);
plt.title('COVID-19 cumulative cases')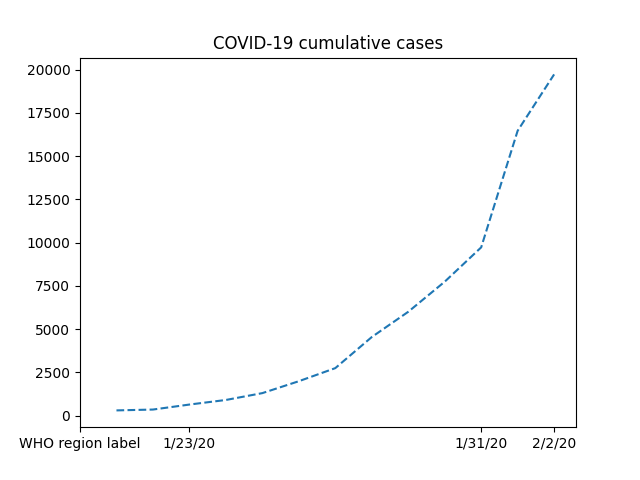
곡선의 진화를 잘못 표기하지 않고는 누락된 데이터를 나타낼 수 없다.
데이터 피팅
한 가지 가능성은 누락된 데이터를 보관하여 수를 측정하는 것이다.
mask 의 특성을 사용하여 마스크된 요소를 선택할 수 있는지 관찰해보자.
print(china_total.mask)
array([False, False, False, False, True, True, True, True, True,
True, True, False, False, False])
invalid = china_total[china_total.mask]
print(invalid)
masked_array(data=[--, --, --, --, --, --, --],
mask=[ True, True, True, True, True, True, True],
fill_value=999999,
dtype=int64)이 마스크의 논리적 부정을 사용하여 유효한 항목에 액세스할 수도 있다.
valid = china_total[~china_total.mask]
print(valid)
masked_array(data=[278, 308, 440, 446, 11791, 14380, 17205],
mask=[False, False, False, False, False, False, False],
fill_value=999999)이제 이 데이터에 대한 매우 간단한 근사치를 만들려면 잘못된 항목과 관련된 유효한 항목을 고려해야 한다. 먼저 데이터가 유효한 날짜를 선택해본다.
마스크된 배열의 마스크를 사용하여 날짜 배열을 인덱싱할 수 있다.
print(dates[~china_total.mask])
array(['1/21/20', '1/22/20', '1/23/20', '1/24/20', '2/1/20', '2/2/20',
'2/3/20'], dtype='<U7')마지막으로 numpy.polyfit 및 numpy.polyval 함수를 사용하여 가능한 한 데이터에 가장 적합한 입방 다각형을 만들 수 있다.
t = np.arange(len(china_total))
params = np.polyfit(t[~china_total.mask], valid, 3)
cubic_fit = np.polyval(params, t)
plt.plot(t, china_total);
plt.plot(t, cubic_fit, '--');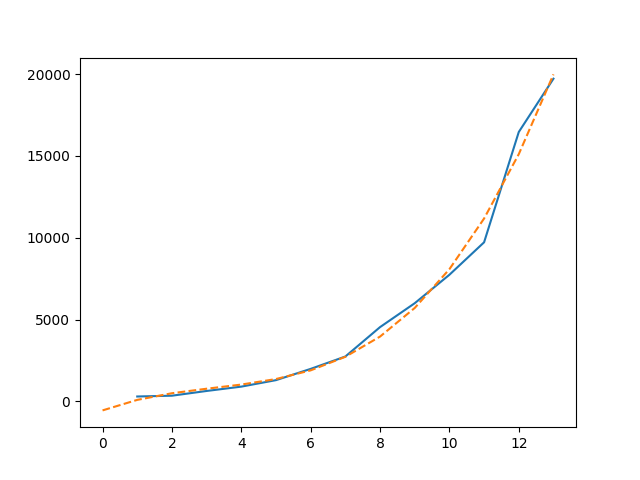
해당 플롯은 선이 서로 위에 얹어있어 제대로 볼 수 없으니 정교하게 해본다.
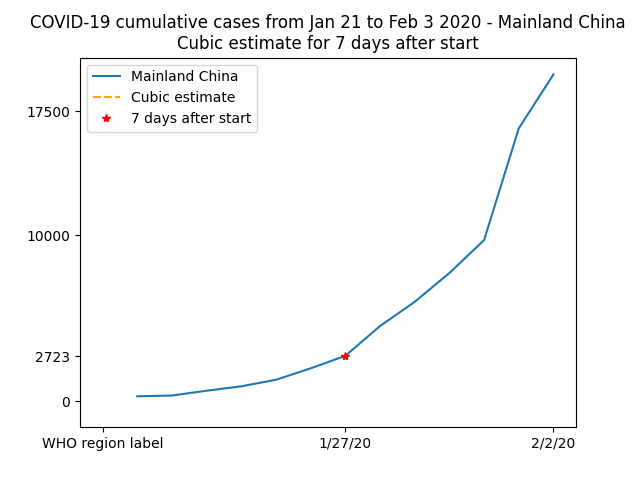
plt.plot(t, china_total, label='Mainland China');
plt.plot(t[china_total.mask], cubic_fit[china_total.mask], '--',
color='orange', label='Cubic estimate');
plt.plot(7, np.polyval(params, 7), 'r*', label='7 days after start');
plt.xticks([0, 7, 13], dates[[0, 7, 13]]);
plt.yticks([0, np.polyval(params, 7), 10000, 17500]);
plt.legend();
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China\n"
....: "Cubic estimate for 7 days after start");