1. 모델의 정의
- 폐암 수술 환자의 생존율 예측하기 (딥러닝 코드)
- 코드 작성해보기
# 텐서플로 라이브러리 안에 있는 케라스 API에서 필요한 함수 불러오기
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import numpy as np
! git clone https://github.com/taehojo/data.git
# 수술 환자 데이터 불러오기
Data_set = np.loadtxt("./data/ThoraricSurgery3.csv", delimiter=",")
X = Data_set[:, 0:16] # 환자의 진찰 기록
y = Data_set[:, 16] # 수술 1년후 사망/ 생존 여부
# 딥러닝 모델 구조 결정
model = Sequential()
model.add(Dense(30, input_dim = 16, activation = 'relu'))
model.add(Dense(1, activation = 'sigmoid'))
model.compile(loss = 'binary_crossentropy', optimizer = 'adam', metrics = ['accuracy'])
history = model.fit(X, y, epochs =5, batch_size = 16)
1-1. 라이브러리 불러오기
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import numpy as npSequential
딥러닝 모델을 하나의 순차적인 레이어(층)로 쌓아 올리는 가장 간단한 방식
모델의 레이어들을 차례대로 추가할 때 사용Dense
Fully connected layer(완전 연결층)를 의미
각 입력 노드가 모든 출력 노드와 연결되는 일반적인 신경망 레이어
1-2. 데이터불러오기
Data_set = np.loadtxt("./data/ThoraricSurgery3.csv", delimiter=",")np.loadtxt
CSV 파일을 넘파이 배열로 불러오는 함수
이 함수는 파일 내의 숫자를 불러와서 배열로 변환"./data/ThoraricSurgery3.csv"
이 파일은 환자의 수술 관련 데이터를 포함한 CSV 파일delimiter=","
CSV 파일에서 데이터를 구분하는 기준이 콤마(,)라는 것을 의미
1-3. 모델 설계
# 딥러닝 모델 구조 결정
model = Sequential()
model.add(Dense(30, input_dim = 16, activation = 'relu'))
model.add(Dense(1, activation = 'sigmoid'))model = Sequential()
순차적인 모델을 시작
모델을 하나씩 레이어를 추가해 쌓아 올릴 수 있도록 준비Dense(30): 첫 번째 레이어는 30개의 뉴런(노드)을 가짐
input_dim=16: 입력으로 16개의 특성(환자의 진찰 기록)을 받기
activation='relu': 활성화 함수로 ReLU(Rectified Linear Unit)를 사용
이는 뉴런이 활성화되는 조건을 결정하는 함수로, 음수를 0으로 만들고, 양수는 그대로 반환
- 이 코드에서는 입력 데이터 X가 16개의 특성(입력 노드)을 가지고 있다
- 첫 레이어의 뉴런 수는 입력 차원보다 크게 설정하는 것이 일반적이다
- 뉴런 수가 많을수록 모델은 더 복잡한 패턴을 학습할 수 있다
- 뉴런 개수를 너무 적게 설정하면 모델이 데이터를 충분히 학습하지 못할 수 있지만, 너무 많이 설정하면
과적합(overfitting)의 위험이 있다
Dense(1): 출력층은 뉴런 1개로, 이 모델이 두 가지 결과(사망/생존)를 예측하므로 출력층에 1개의 뉴런을 사용
activation='sigmoid': 이진 분류를 위해 출력값을 0과 1 사이로 압축하는 시그모이드 함수를 사용
0에 가까우면 '사망', 1에 가까우면 '생존'을 의미함
1-4. 모델 컴파일 및 학습
model.compile(loss = 'binary_crossentropy', optimizer = 'adam', metrics = ['accuracy'])
history = model.fit(X, y, epochs =5, batch_size = 16)-
loss='binary_crossentropy': 이진 분류 문제이므로, 모델이 학습할 때 사용할 손실 함수로 이진 크로스엔트로피를 사용
이는 예측값과 실제값 사이의 차이를 계산하는 방식
optimizer='adam': Adam 최적화 알고리즘을 사용
이 알고리즘은 경사하강법을 기반으로 하며, 학습 속도를 빠르게 하고 효율적으로 최적화
metrics=['accuracy']: 학습 동안 모델 성능을 평가할 때 정확도를 사용 -
model.fit(X, y): 입력 데이터 X와 출력(레이블) y를 사용해 모델을 학습
epochs=5: 데이터를 5번 반복해서 학습
한 에포크는 데이터셋 전체를 한 번 학습시키는 단위를 의미
batch_size=16: 16개의 데이터를 한 번에 학습시키며, 이를 배치(batch) 라고 부름
한 번의 배치가 끝나면 가중치가 업데이트
2. 데이터 다루기
- 머신러닝과 딥러닝에서 사용할 수 있게 잘 정제된 데이터 형식은 매우 중요하다
- 데이터 다루기 작업은 모든 머신러닝과 딥러닝 프로젝트의 첫 단추이자 중요한 작업이다
- 실습환경 : colab
2-1. 인디언 데이터 다루기
- 피마 인디언을 대상으로 당뇨병 여부를 측정한 데이터 다뤄보기
read_csv()함수로 csv파일을 불러와 df라는 이름의 데이터 프레임으로 저장head()함수를 사용하여 데이터의 내용을 간단히 확인하였음
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
df = pd.read_csv('/content/data/pima-indians-diabetes3.csv')
df.head()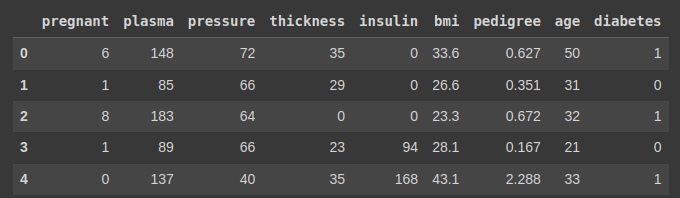
2-2. 데이터 살펴보기
2-2-1. 정상과 당뇨병 환자가 각각 몇 명인지 조사하기
df['칼럼명']: 데이터 프레임의 특정 칼럼을 불러오기value_counts(): 각 컬럼의 값이 몇 개씩 있는지 조사- 정상 : 0 , 당뇨병 : 1
df['diabetes'].value_counts()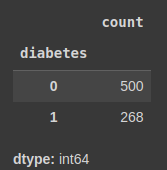
2-2-2. describe() : 정보별 특징을 더 자세히 조사하기
df.describe()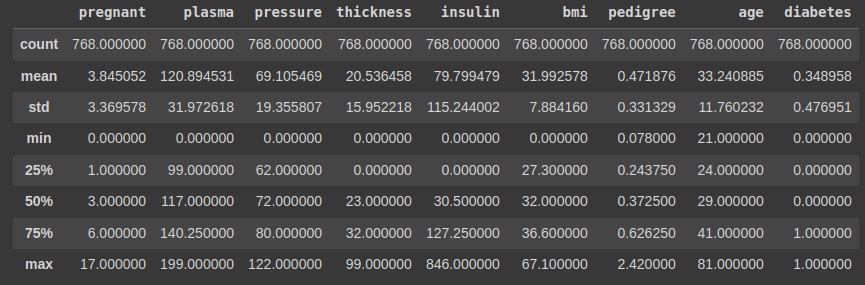
2-2-3. 데이터의 상관관계 살펴보기
colormap = plt.cm.gist_heat # 그래프의 색상 구성 정하기
plt.figure(figsize = (12, 12)) # 그래프의 크기 정하기
sns.heatmap(df.corr(), linewidths = 0.1, vmax = 0.5, cmap = colormap, linecolor = 'white', annot = True)
plt.show()heatmap(): seaborn 라이브러리에서 각 항목간의 상관관계를 나타내줌- 두 항목이 전혀 다른 패턴으로 변화하면 0
- 서로 비슷한 패턴으로 변할수록 1에 가까움
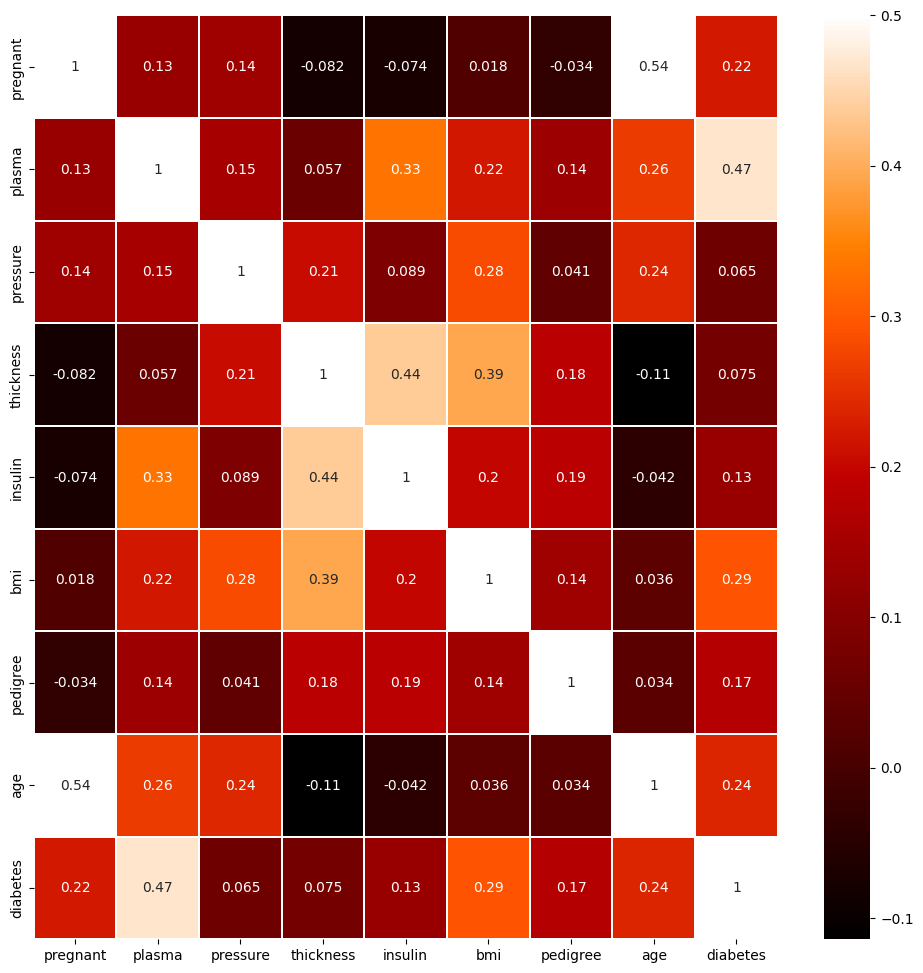
우리가 주의깊게 살펴볼 부분은 맨 밑의 diabetes 행이다
해당 행에서 숫자가 높을 수록 두 데이터 사이의 상관관계가 높다는 의미
2-3. 중요 데이터 추출하기
- 위의 heatmap에서
plasma (공복 혈당 농도)와BMI가 우리가 예측하고자 하는 diabetes와 상관관계가 높은 2가지 변수임 - 따라서
plasma와BMI가 예측모델을 만드는데 중요한 역할을 할 것이라는 생각을 해야함
두 항목에 대하여 당뇨의 발병 여부와 어떤 관계가 있는지 살펴보기
2-3-1. 히스토그램 사용
plasma 와 당뇨 사이의 관계
plt.hist(x = [df.plasma[df.diabetes == 0], df.plasma[df.diabetes == 1]],
bins = 30, histtype = 'barstacked', label = ['normal', 'diabetes'])
plt.legend()-
plasma 칼럼 중 diabetes 값이 0인 것과 1인 것을 구분하여 불러옴
-
bins: x 축을 몇 개의 막대로 쪼개어 보여줄 것인지 정하는 것
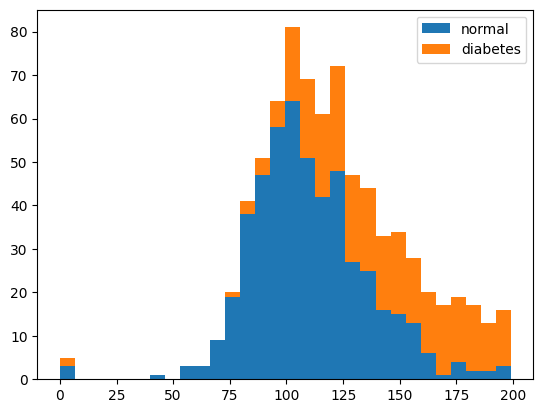
-
plasma수치가 높아질수록 당뇨인 경우가 많다
bmi 와 당뇨 사이의 관계
plt.hist(x = [df.bmi[df.diabetes == 0], df.bmi[df.diabetes == 1]],
bins = 30, histtype = 'barstacked', label = ['normal', 'diabetes'])
plt.legend()
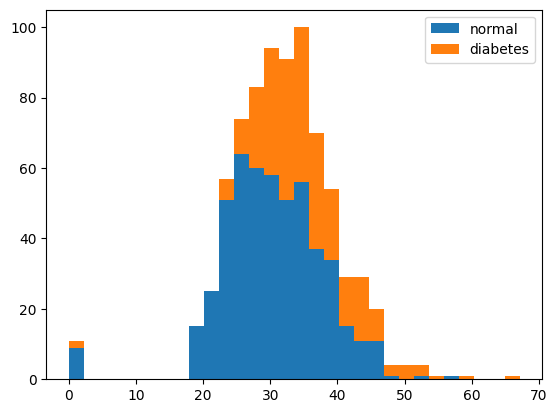
- 이 경우에도
bmi가 높아질 수록 당뇨의 발병률도 증가하는 것을 확인할 수 있다
- 이런식으로 결과에 미치는 영향이 큰 항목을 발견하는 것이 데이터 전처리 과정 중 하나임
- 이 외에도, NULL 값을 평균이나 최빈 값으로 채우거나 이상치를 제거하는 과정 등이 데이터 전처리 과정에 포함될 수 있다
2 - 4. 피마 인디언 당뇨병 예측 실행
pandas라이브러리를 사용하기에iloc[]함수를 사용하여 X와 y를 저장iloc[]: 데이터 프레임에서 원하는 범위만큼 가져와 저장
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import pandas as pd
df = pd.read_csv('/content/data/pima-indians-diabetes3.csv')
X = df.iloc[:, 0:8] # 8개의 특징 변수를 X로 저장
y = df.iloc[:, 8] # 당뇨병 여부를 y로 저장
model = Sequential()
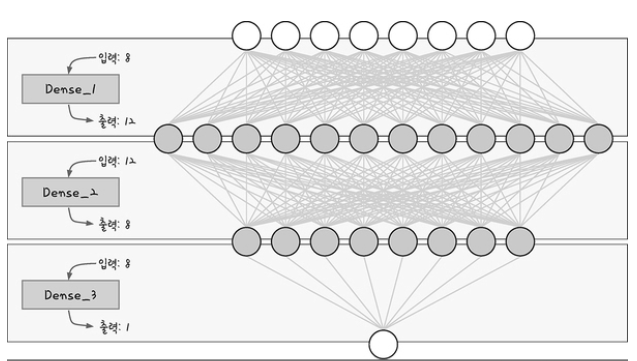
model.add(Dense(12, input_dim = 8, activation = 'relu', name = 'Dense_1'))
model.add(Dense(8, activation = 'relu', name = 'Dense_2'))
model.add(Dense(1, activation = 'sigmoid', name = 'Dense_3'))
model.summary()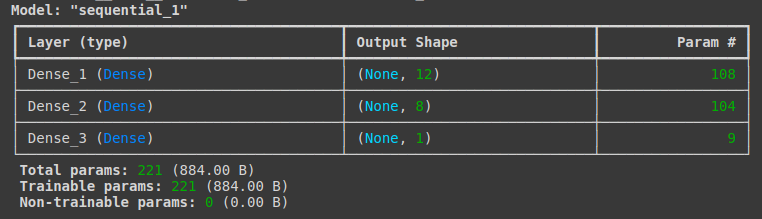
Layer: 층의 이름과 유형을 나타냄
각 층의 이름은 자동으로 정해지지만, name = '층 이름' 으로 이름 지정이 가능Output Shape: 각 층에 몇 개의 출력이 발생하는지 나타냄
( 행(샘플)의 수, 열(속성)의 수)를 의미함- 행(샘픙)의 수는 batch_size에 정한만큼 입력되므로 딥러닝 모델에서는 이를 특별히 세지 않는다 (None으로 나타남)
- 8개의 입력이 첫 번째 은닉층을 지나 12개가 되고, 두 번째 은닉층을 지나 8개가 되었다가 출력층에서는 1개의 출력을 만든다
Param: 파라미터의 수, 총 가중치와 바이어스 수의 합을 나타냄
- 첫 번째 층 : 입력 8개가 12개의 노드로 분산됨
8 x 12 (가중치) + 12 (바이어스) = 108- 두 번째 층 : 입력 12개가 8개의 노드로 분산됨
12 x 8 (가중치) + 8 (바이어스) = 104- 세 번째 층 : 입력 8개가 1개의 노드로 분산
8 x 1 (가중치) + 1 (바이어스) = 9
출처 : 모두의 딥러닝
- 학습 후 결과 출력
model.compile(loss = 'binary_crossentropy', optimizer = 'adam', metrics = ['accuracy'])
history = model.fit(X, y, epochs = 100, batch_size = 5)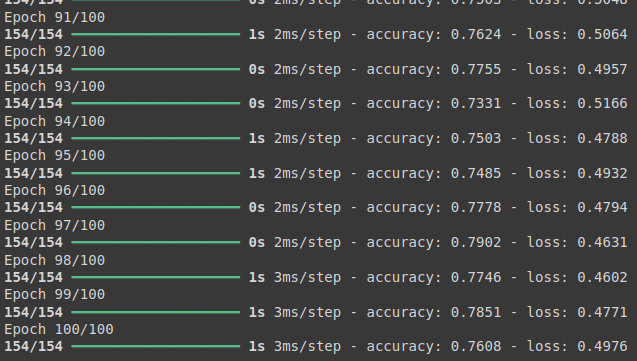
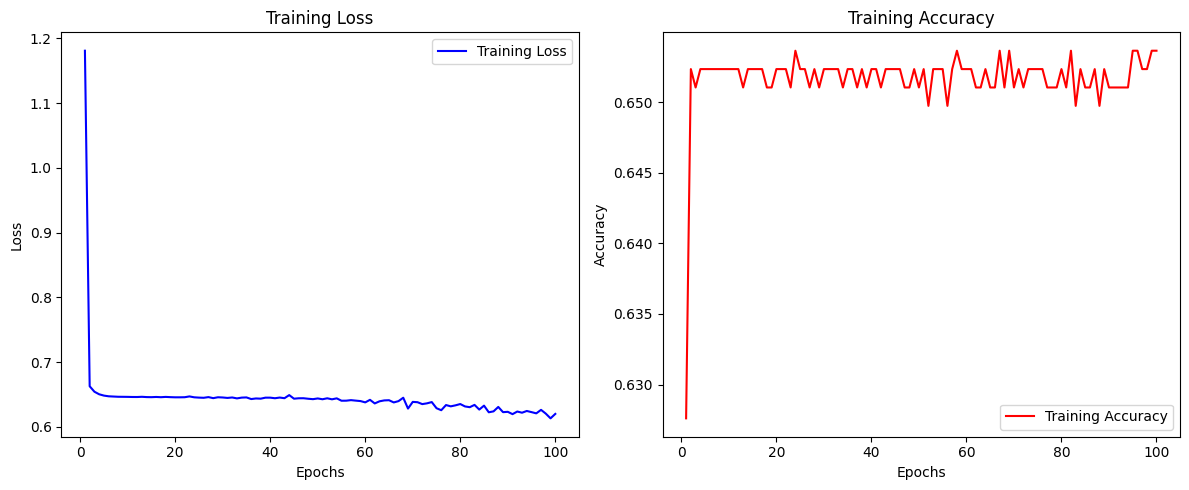
accuracy와loss에 대한 그래프 출력
import matplotlib.pyplot as plt
# 학습 결과 저장된 history 객체에서 손실값과 정확도 값 가져오기
loss = history.history['loss']
accuracy = history.history['accuracy']
epochs = range(1, len(loss) + 1)
# 손실 그래프 그리기
plt.figure(figsize=(12, 5))
plt.subplot(1, 2, 1)
plt.plot(epochs, loss, 'b', label='Training Loss')
plt.title('Training Loss')
plt.xlabel('Epochs')
plt.ylabel('Loss')
plt.legend()
# 정확도 그래프 그리기
plt.subplot(1, 2, 2)
plt.plot(epochs, accuracy, 'r', label='Training Accuracy')
plt.title('Training Accuracy')
plt.xlabel('Epochs')
plt.ylabel('Accuracy')
plt.legend()
# 그래프 출력
plt.tight_layout()
plt.show()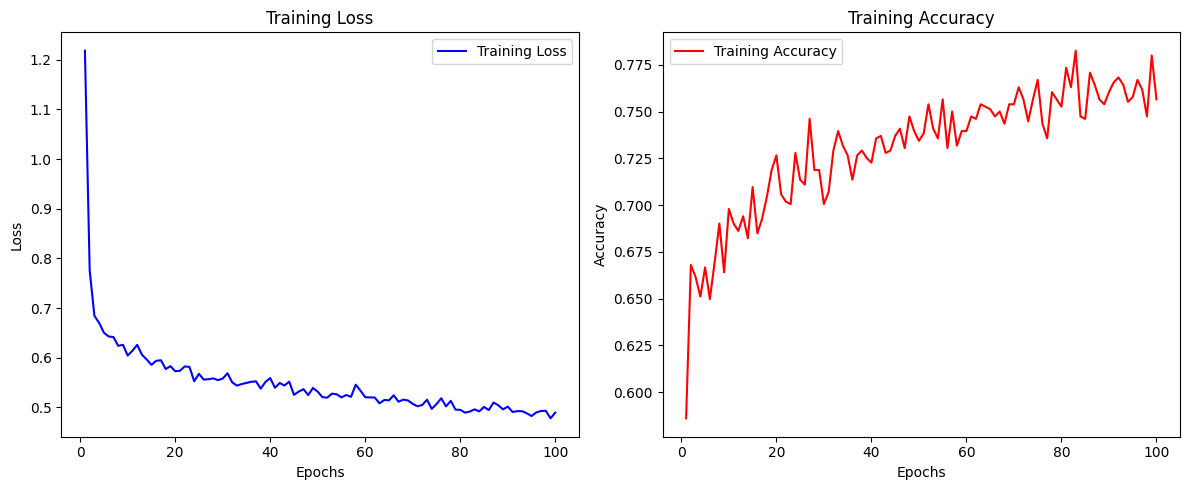
optimizer를 adam에서sgd(확률적 경사 하강법)으로 변경시켜 돌려보았음model.compile(loss = 'binary_crossentropy', optimizer = 'sgd', metrics = ['accuracy'])
- loss와 accuracy가 임계값에서 감소하거나 증가하지 않음 (수렴한다)