Support Vector Machine
-
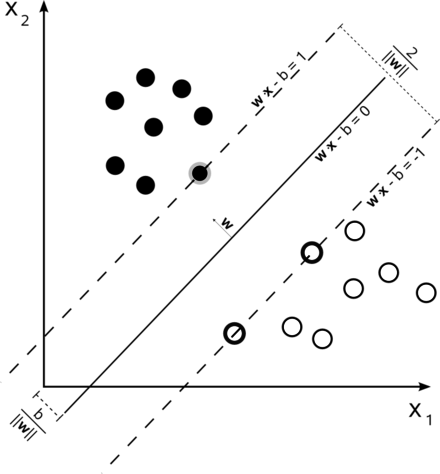
hyperplane과 가장 가까운support vector사이의 거리margin을 최대가 되도록 하는 알고리즘hyperplane: class 를 구분짓는 평면. N 차원일 경우 N-1 차원으로 표현되는 평면support vector:hyperplane과 가장 가까운 samplemargin:hyperpiane과support vector사이의 거리
-
classfication(분류), regression (회귀) and outliers detection (이상치 탐지) 에 활용된다.
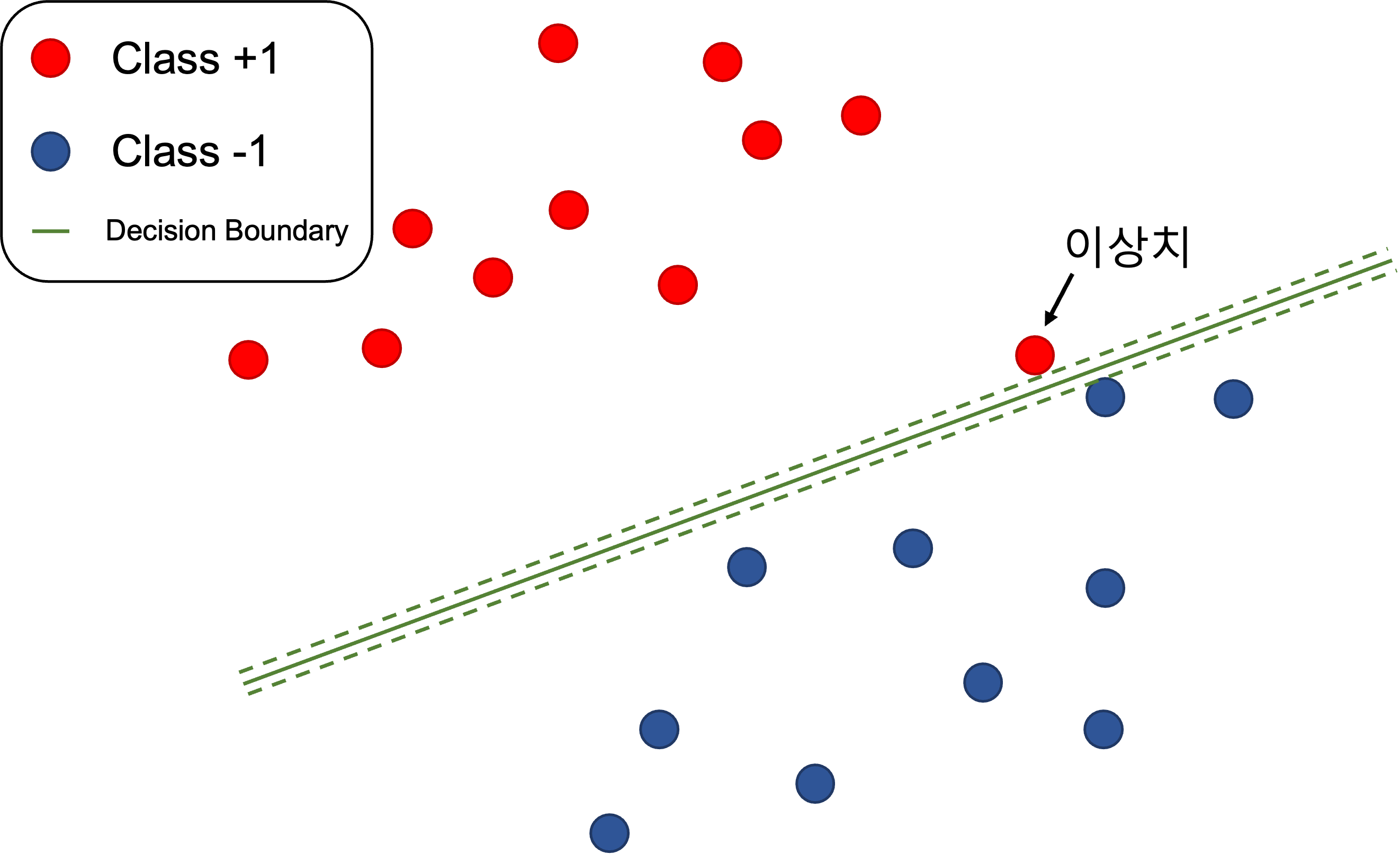
soft margin classification
- 현실에서는 noise, outlier (이상치) 로 선형 분류가 불가능하거나 마진이 작아지는 문제가 생길 수 있다.
- 이를 보정하기 위해
slack variableξ를 허용하여 보다 조건을 완화하고 여유 있게 최적화
- 이를 보정하기 위해
kernel trick
- 선형 분류가 불가능한 경우 (
non-linearly separable)kernel trick을 통해 비선형 분류로 활용한다.- 보다 고차원으로의 mapping function 을 통해 매핑,
hyperplane을 찾을 수 없던 모델에 대해hyperplane을 찾을 수 있도록 한다- 대표적인 커널 함수로
polynomial,gaussian,sigmoidkernel 등이 있다. - scikit-learn SVM 라이브러리에서는
linear,polynomial,rbf,sigmoid옵션으로 제공된다.
>>> linear_svc = svm.SVC(kernel='linear')
>>> linear_svc.kernel
'linear'
>>> rbf_svc = svm.SVC(kernel='rbf')
>>> rbf_svc.kernel
'rbf'Scikit-learn 으로 SVM training
- SVM training
from sklearn.svm import SVC
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from matplotlib.colors import ListedColormap
import matplotlib.pyplot as plt
import numpy as np
def train_svm():
"""
iris(붓꽃) 데이터셋으로 꽃받침 길이, 꽃잎 길이 2개 feature 로 분류하기
:return:
"""
iris_dataset = datasets.load_iris()
x = iris_dataset.data[:, [2, 3]] # 꽃 데이터셋 중 일부 feature 만 추출 (꽃받침, 꽃길이)
y = iris_dataset.target # 꽃 품종 label
# test_size : test data와 training data 비율. 0.3이면 test 30%, training 70%
# random_state: 데이터셋을 섞기 위해 사용되는 random seed. 고정일 경우 매번 동일하게 재현
# stratify : stratification (계층화). training, test 데이터 셋의 label 비율을 input 데이터셋과 동일하게 함
x_train, x_test, y_train, y_test = train_test_split(x, y, test_size=0.3, random_state=1, stratify=y)
print(f'class label : {np.unique(y)}')
# stratification 결과 체크
print(f'y original data set label count : {np.bincount(y)}')
print(f'y training data set label count : {np.bincount(y_train)}')
print(f'y test data set label count : {np.bincount(y_test)}')
# feature 표준화
sc = StandardScaler()
# traing data set 의 feature dimension 마다 평균과 표준 편차 계산
sc.fit(x_train)
# transform: 평균과 표준 편차로 data set 을 표준화
x_train_std = sc.transform(x_train)
x_test_std = sc.transform(x_test)
# training
svm = SVC(kernel='linear', C=1.0, random_state=1)
svm.fit(x_train_std, y_train)
# 시각화를 위해 training, test data set 결합
x_combined_std = np.vstack((x_train_std, x_test_std))
y_combined = np.hstack((y_train, y_test))
# 시각화
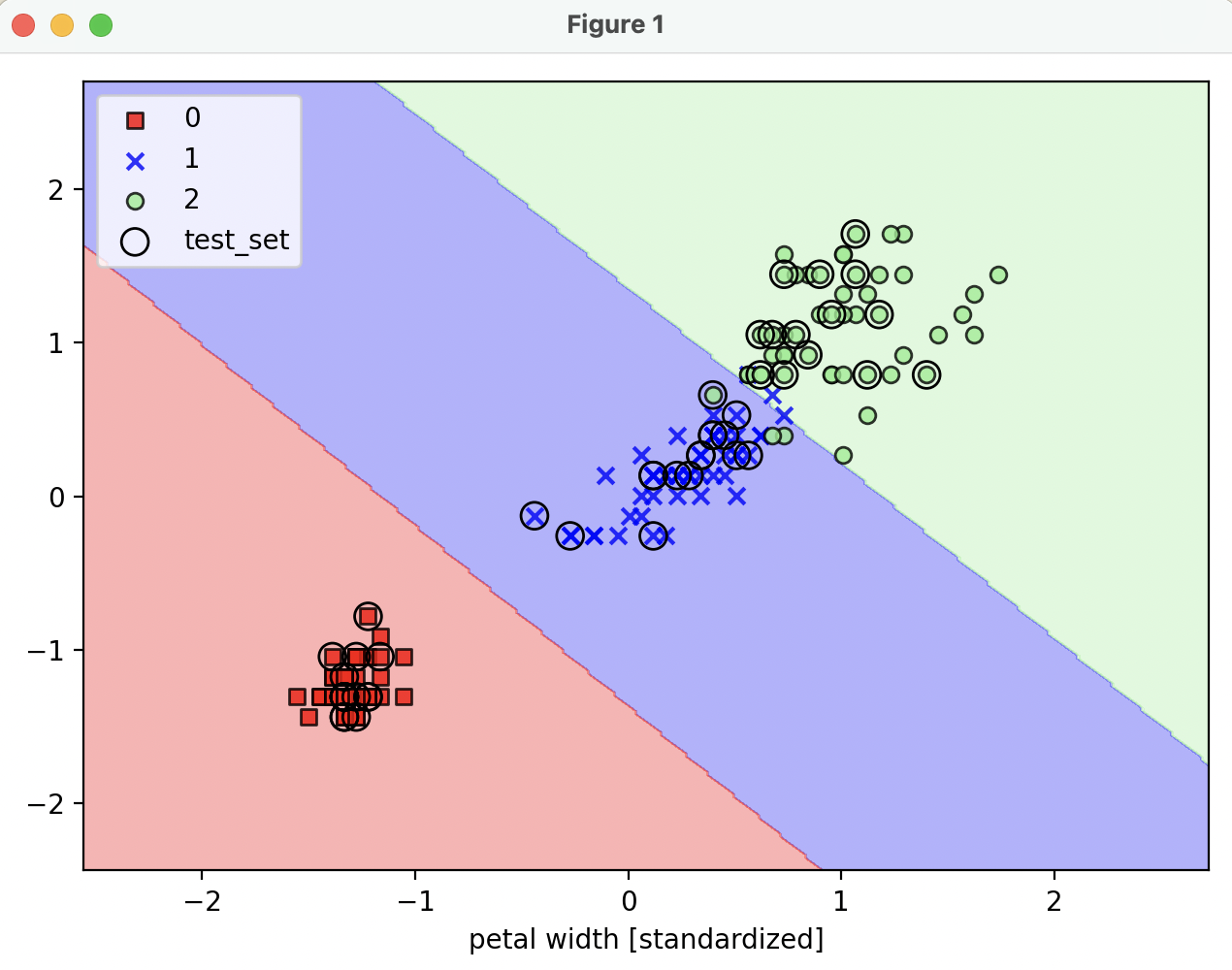
plot_decision_regions(x=x_combined_std, y=y_combined, classifier=svm, test_idx=range(105,150))
plt.xlabel('petal length [standardized]')
plt.xlabel('petal width [standardized]')
plt.legend(loc='upper left')
plt.tight_layout()
plt.show()- 시각화
def plot_decision_regions(x, y, classifier, test_idx=None, resolution=0.02):
"""
시각화
:param x: 붓꽃 데이터셋 feature (0번 열 : 꽃받침 길이, 1번 열 : 꽃잎 길이)
:param y: class label
:param classifier: 분류기
:param test_idx:
:param resolution:
:return:
"""
# 마커, 컬러맵 설정
markers = ('s', 'x', 'o', '^', 'v')
colors = ('red', 'blue', 'lightgreen', 'gray', 'cyan')
cmap = ListedColormap(colors[:len(np.unique(y))])
# decision boundary (결정 경계) 그리기
# feature
x1_min, x1_max = x[:, 0].min() - 1, x[:, 0].max() + 1 # feature column 0 -> 꽃받침 길이
x2_min, x2_max = x[:, 1].min() - 1, x[:, 1].max() + 1 # feature column 1 -> 꽃잎 길이
# meshgrid : 축에 해당하는 1차원 배열을 받아 벡터 공간의 모든 좌표를 담은 행렬을 반환
xx1, xx2 = np.meshgrid(np.arange(x1_min, x1_max, resolution),
np.arange(x2_min, x2_max, resolution))
# ravel : 배열을 1차원 배열로 펼치기
# T : transpose
# 2개 배열을 펼치고 각 행으로 붙이고 transpose 하여 2개의 열로 변환, 이후 class label 예측
z = classifier.predict(np.array([xx1.ravel(), xx2.ravel()]).T)
# xx1, xx2 와 같은 차원의 그리드로 크기 변경
z = z.reshape(xx1.shape)
# contourf : 등고선 그래프 그리기
plt.contourf(xx1, xx2, z, alpha=0.3, cmap=cmap)
# x, y축 limit 설정
plt.xlim(xx1.min(), xx1.max())
plt.ylim(xx2.min(), xx2.max())
for idx, cl in enumerate(np.unique(y)):
plt.scatter(x=x[y == cl, 0], y=x[y == cl, 1],
alpha=0.8, c=colors[idx],
marker=markers[idx], label=cl,
edgecolor='black')
if test_idx:
x_test, y_test = x[test_idx, :], y[test_idx]
plt.scatter(x_test[:, 0], x_test[:, 1],
facecolors='none', edgecolor='black', alpha=1.0,
linewidth=1, marker='o',
s=100, label='test_set')