M3T: three-dimensional Medical image classifier using Multi-plane and Multi-slice Transformer
Paper review
자세한 내용은 논문을 참고하세요.
M3T: three-dimensional Medical image classifier using Multi-plane and Multi-slice Transformer: https://openaccess.thecvf.com/content/CVPR2022/papers/Jang_M3T_Three-Dimensional_Medical_Image_Classifier_Using_Multi-Plane_and_Multi-Slice_Transformer_CVPR_2022_paper.pdf
연구실 내에서 세미나를 진행하며 준비한 자료를 바탕으로 정리하는 글입니다. 궁금한 점이나 문제가 있는 부분은 댓글로 작성해주세요!
1. Introduction
-
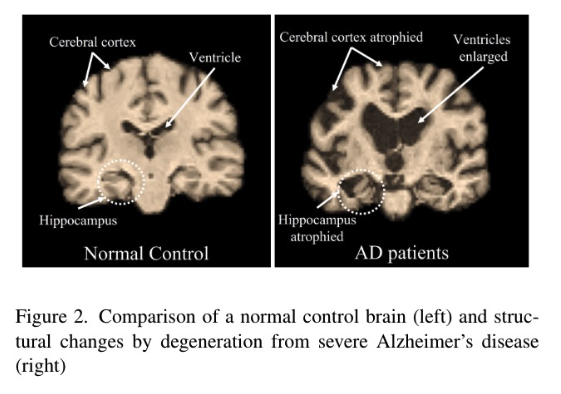
3D MRI 영상은 AD 관련 이상을 분석하는 데 널리 사용되어 왔지만, manual로 MRI 영상을 분석/추출하는 것은 어려운 문제이며, 검사자 간 또는 검사자 내의 변동성(inter- or intra-operator variablility) 문제로 인해 시간이 많이 소요되고 오진에 취약함.
-
2D/3D representation 학습에는 시간과 표현력 사이의 trade-off가 존재하며, 환경을 고려하여 둘 중 하나의 image를 선택하게 됨.
-
CNN과 transformer 사이에도 상충관계가 있는데, CNN은 inductive bias와 locality를 가지고 있어 데이터가 적더라도 높은 성능을 달성할 수 있음. 그러나 이러한 편항은 receptive field가 좁아 고차원 데이터를 다루는 데 한계가 있음.
-
하지만 transformer는 inductive bias가 없어 적은 데이터셋에서는 학습이 되지 않지만, 넓은 receptive field를 통해 이미지 내의 특징을 포괄적으로 학습할 수 있음.
-
본 논문에서는 2D CNN, 3D CNN, Transformer를 함께 사용하여 3D 의료 영상을 처리하고자 하였음.
2. Overview
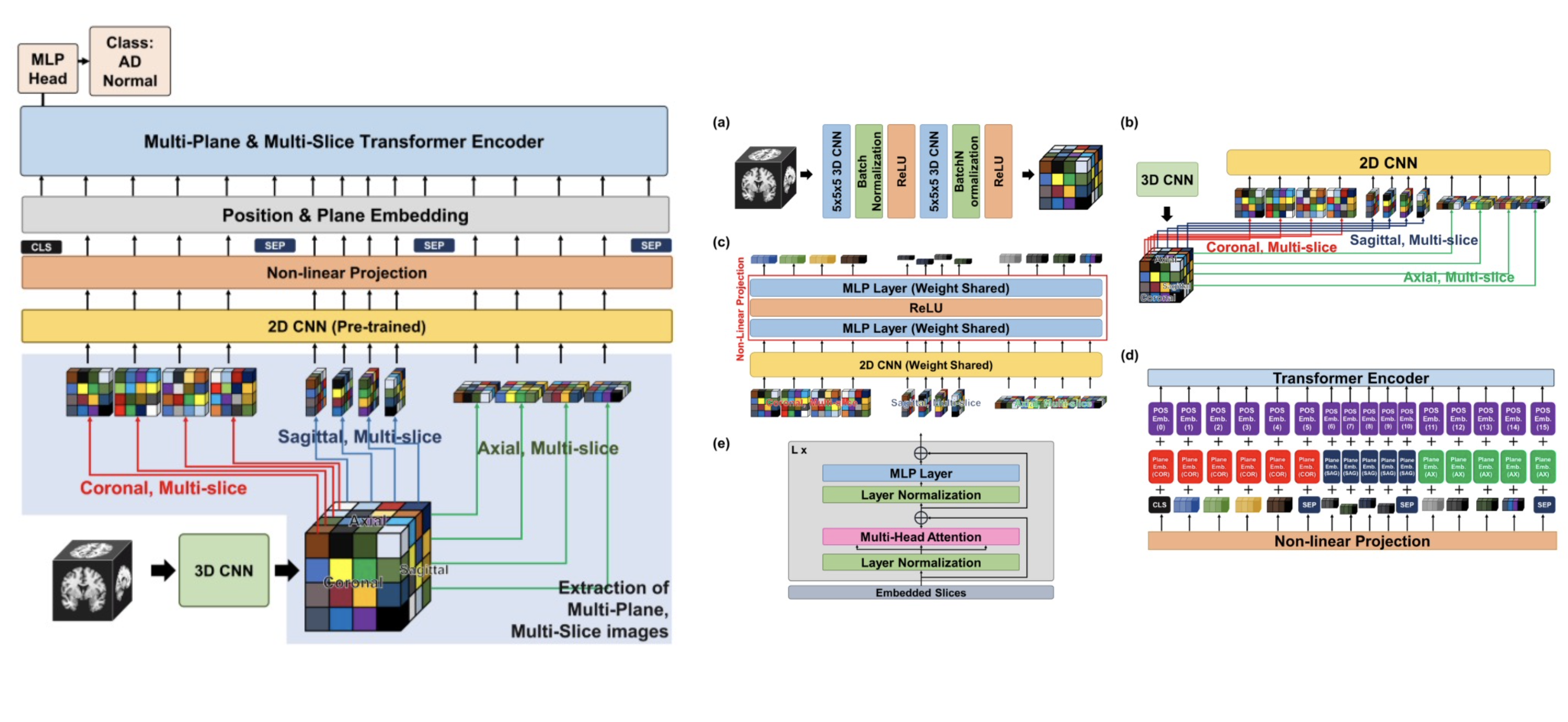
-
input으로 3D 영상을 입력을 받아 CNN을 통해 feature를 추출하게 됨. 3D 영상의 특징으로 coronal/sagittal/axial 축으로 2D CNN을 거친 뒤 transformer를 이용하여 특징을 추출함.
-
자세한 설명은 이후 파트에서 설명
3. Architecture
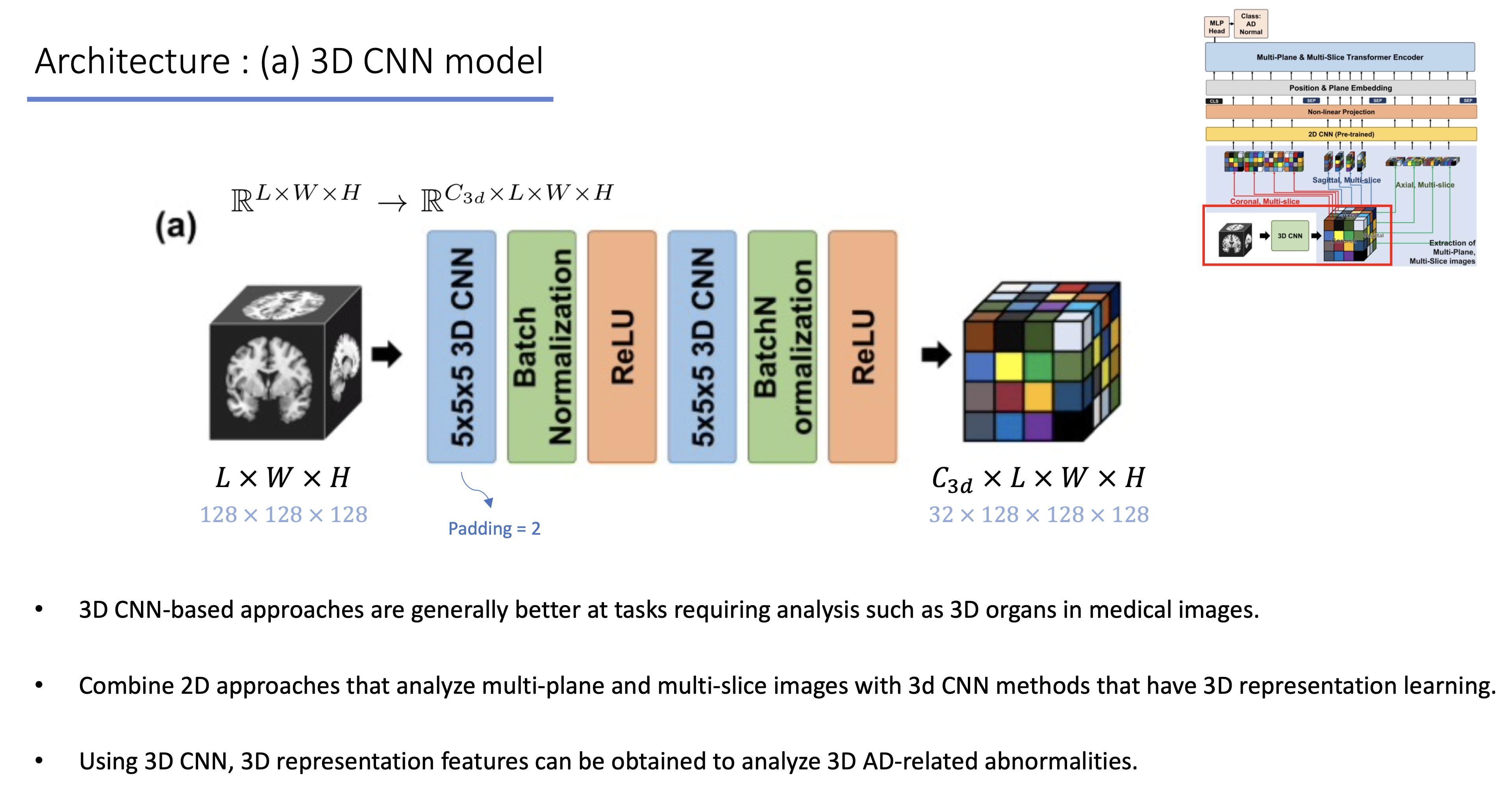
(a) 3D CNN model
-
3D 영상이 input으로 들어오게 되면 2-layer 3D CNN을 거치게 됨.
-
3D input과 동일한 size를 가지며 32채널의 representation vector로 변하게 됨.
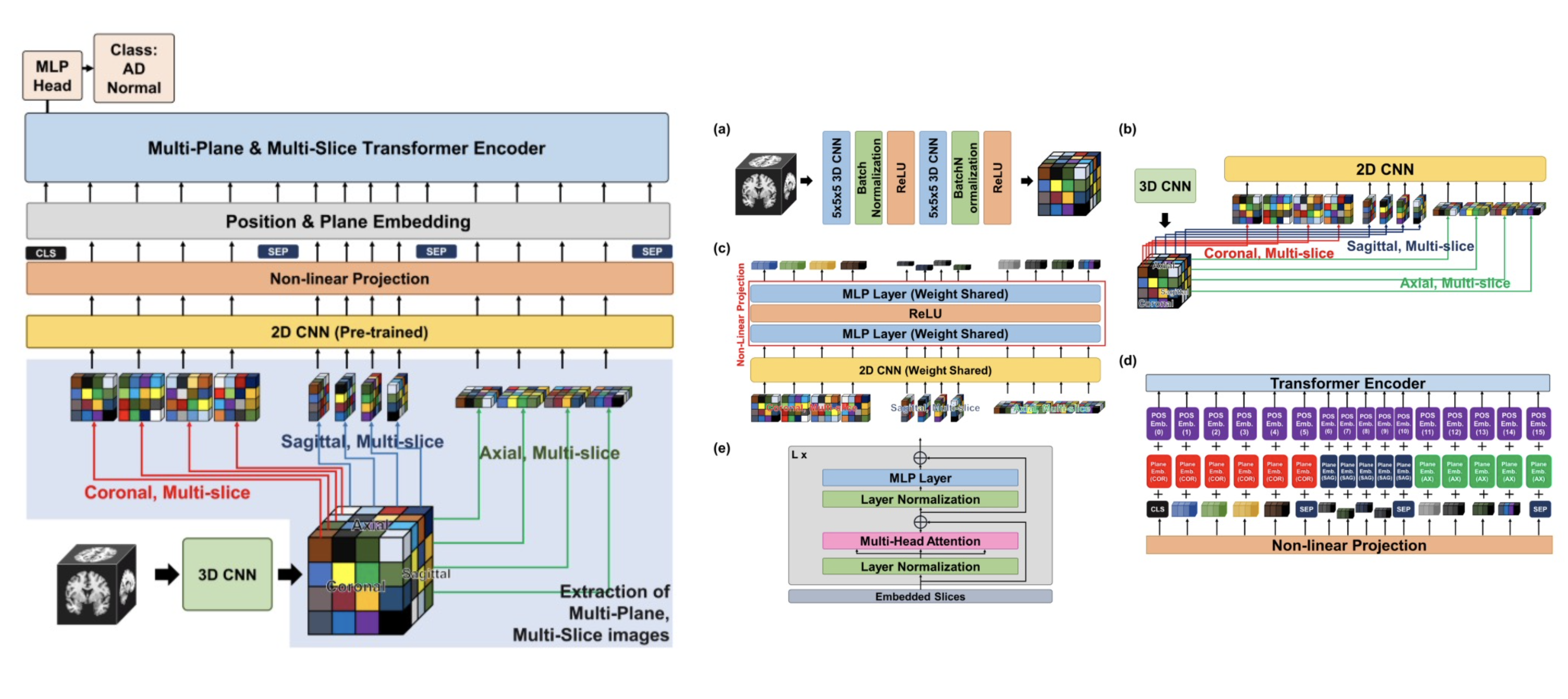
(b) Extraction of Multi-plane and Multi-slice images
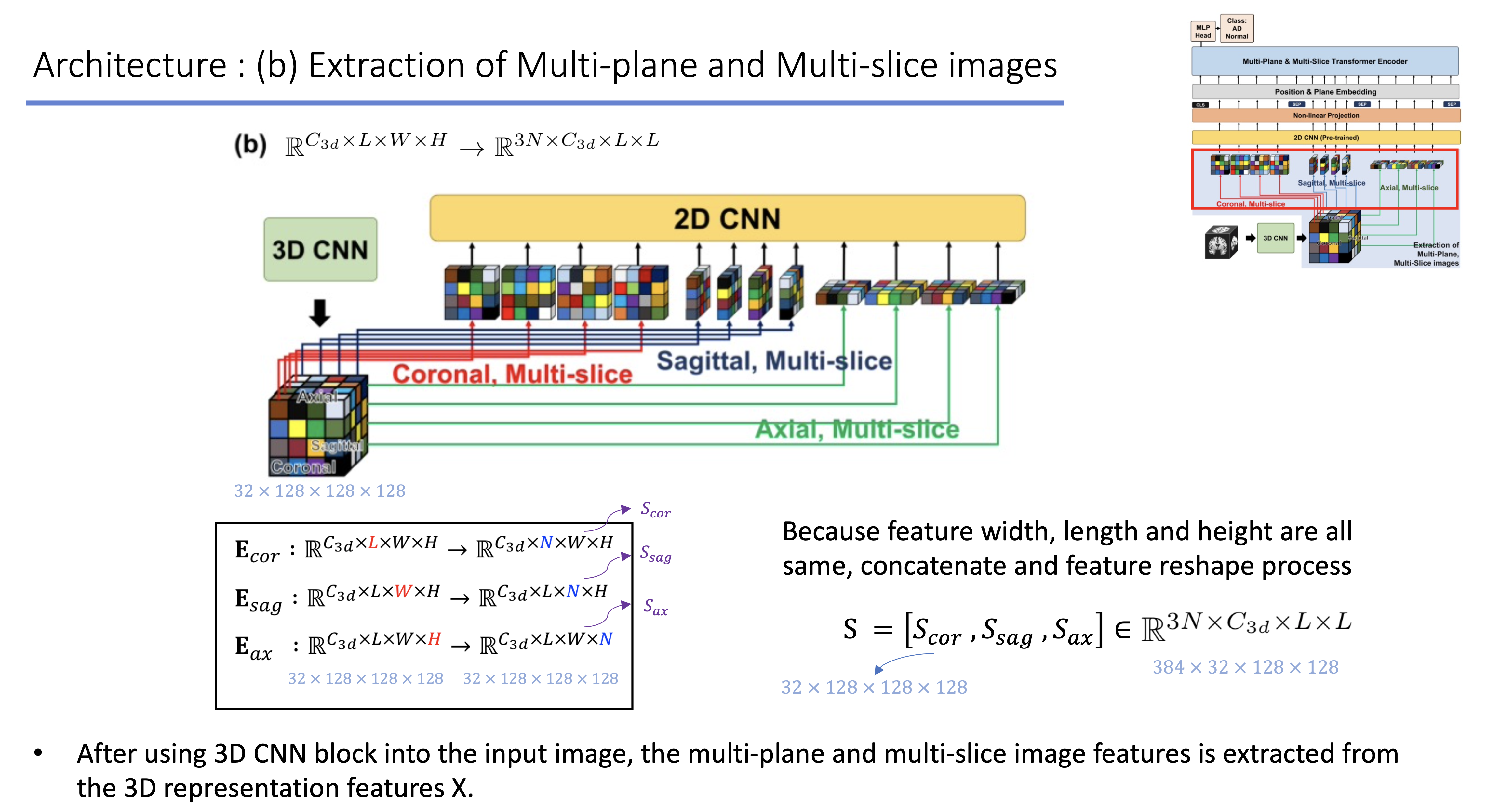
-
(a)의 output인 3d representation vector를 input으로 사용하며, 3D 영상의 특징을 각 축(coronal, sagittal, axial) 방면으로 slice하여 2D CNN을 거치게 됨.
-
예시로, coronal 축을 기준으로 하나의 slice의 shape은 (32, 1, 128, 128)일 것이고 128개의 slice가 존재를 하며, 기존 32채널로 표현되어있는 representation vector를 2D CNN을 거쳐 32차원의 representation vector로 표현함. 즉, 2D CNN을 거친 이전 slice의 shape은 (32, 128, 128, 128)이고, 각 multi-plane을 기준으로 reshape을 한 뒤 concat을 진행함.
-
이후, size를 줄이며 channel을 늘리는 2D CNN & Global Average Pooling을 진행함.
(c) non-linear projection

- (b)에서 진행한 2D CNN + GAP와 함께 MLP layer를 거치며 384개의 multi-plane의 관점에서 256개의 feature를 만들어 (384, 256)의 feature를 생성하게 됨.
(d) Position & Plane embedding
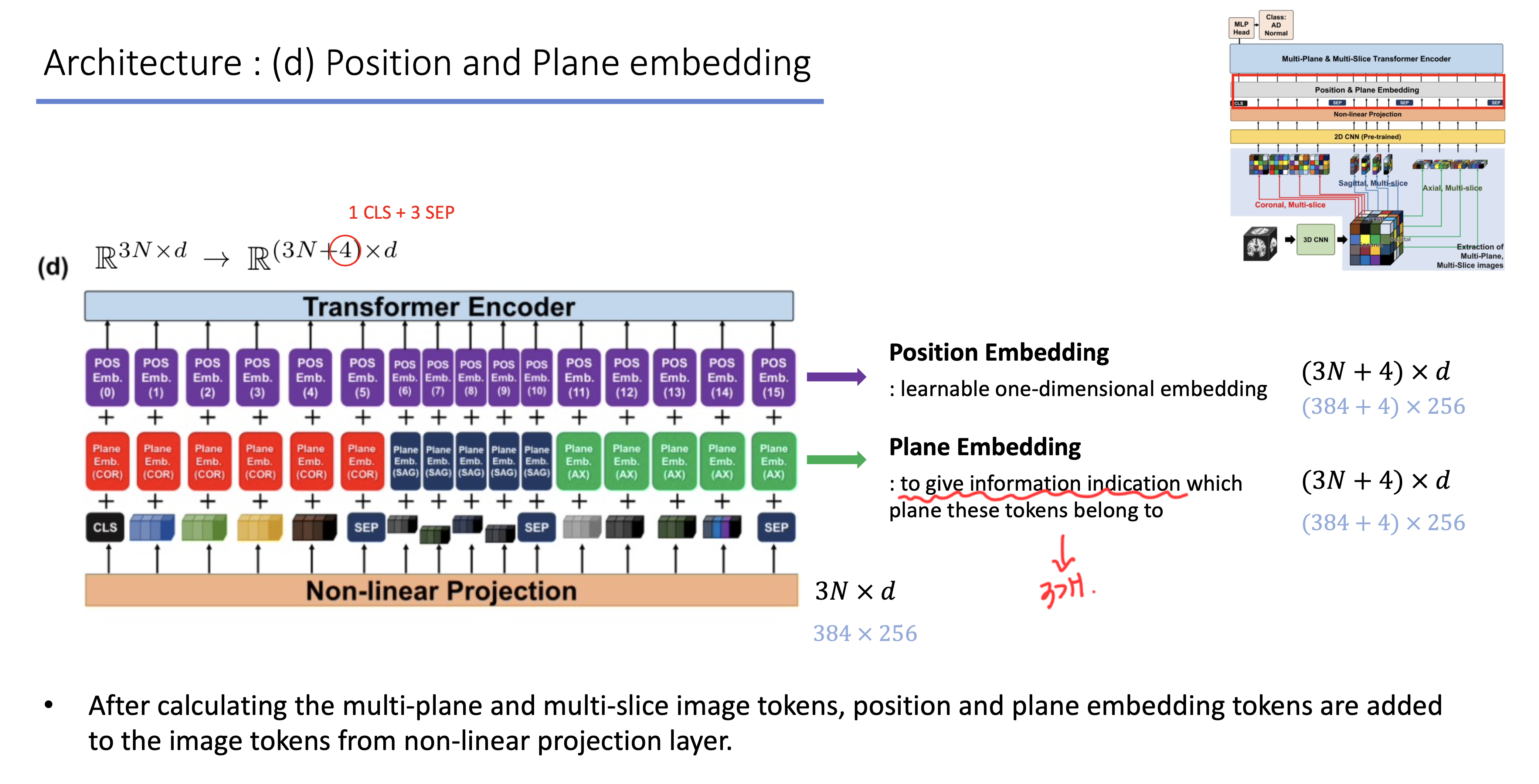
-
시작할 때 추가되는 CLS token, 각 plane을 구분해줄 수 있는 2개의 SEP token, 마지막을 알려주는 SEP token, 총 4개의 special token을 추가해줌.
-
multi-plane 관점에서의 각 128개의 slice를 기준으로 서로 다른 plane별로 plane embedding을 더해줌. 이 때 각 plane별로 같은 plane끼리는 같은 값을 더해줌.
-
positional embedding은 transformer에서 사용하는 embedding을 사용하여 각 token에 더해주며, learnable한 파라미터임.
(e) Tranformer encoder
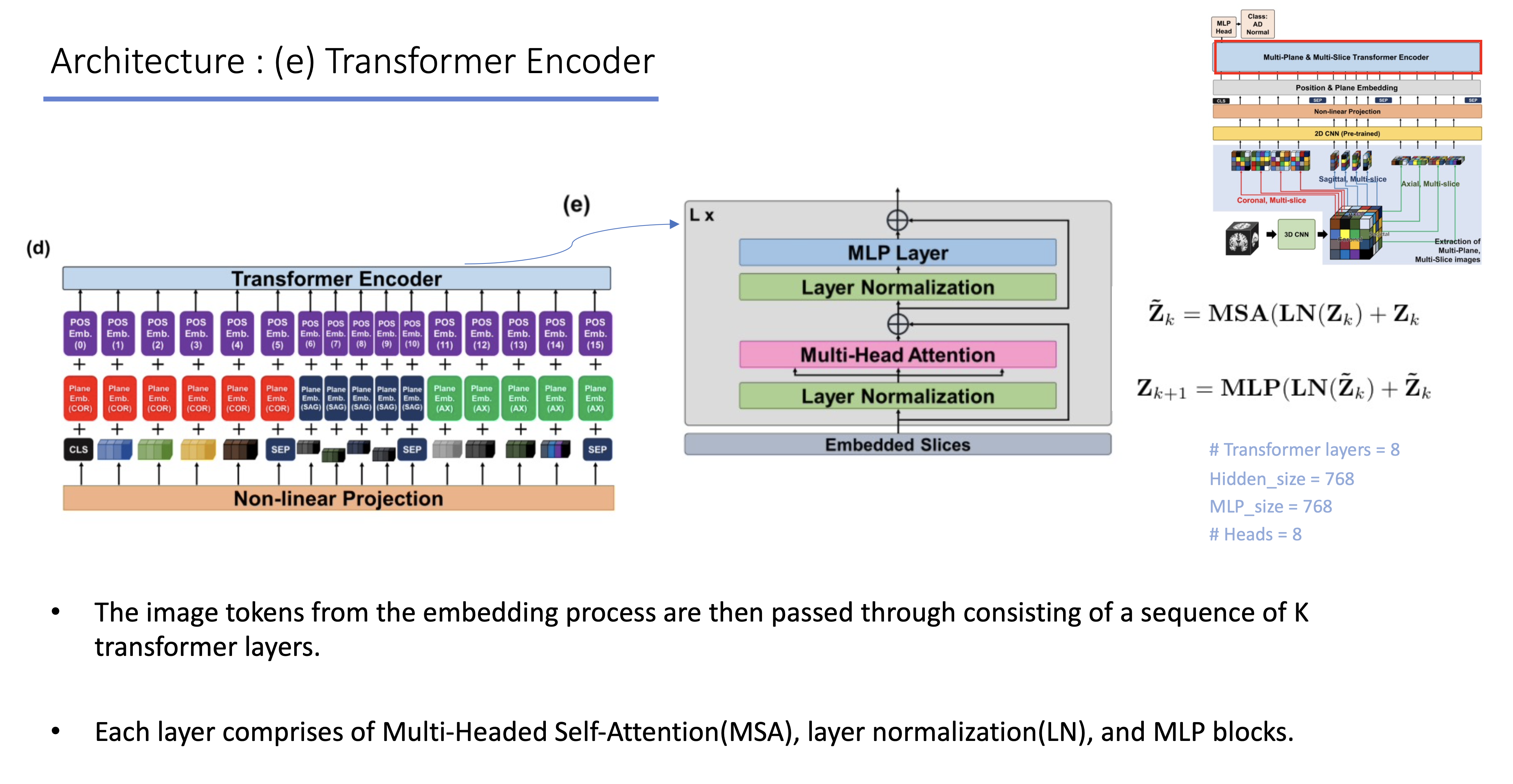
- (d)에서 embedding을 마친 representation vector는 transformer layer를 거치게 됨. 최종적으로 cls token을 이용하여 classification을 진행하거나 feature vector를 이용한 downstream task를 진행함.
4. Result & Ablation study
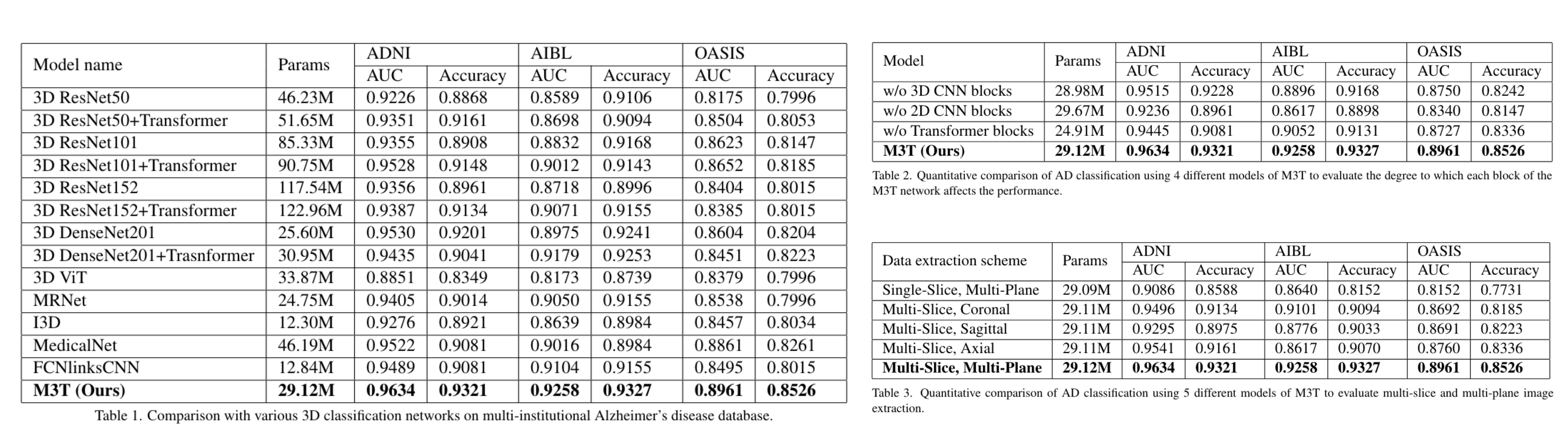
- 비교한 model로는 일반적인 3D ViT, 3D CNN과 함께 3D CNN + Transformer도 진행을 하였음. 3D 관점에서만 모델을 학습하는 것 보다 2D 관점, 즉 plane 관점에서의 feature 또한 함께 고려하는 것이 성능 향상이 가장 많이 되는 것을 확인할 수 있음.
