0. imbalanced data의 문제를 해결하기 위한 resampling
- 데이터의 각각의 class들의 비율이 균일하지 않게 분포하는 경우
- 모델 성능의 왜곡이 발생
- 높은 정확도를 보이는 것 처럼 보이지만, 비율이 적은 class는 제대로 분류하지 못하는 문제가 발생
-> 이를 해결하기 위해서 resampling을 통해 데이터의 불균형을 해결
🔴 출처
김성범[ 고려대 / 산업경영공학부 ] 교수님의 youtube 채널에서 보고 공부한 내용을 정리하였습니다.
채널명 : 김성범[ 교수 / 산업경영공학부 ]
채널주소 : https://www.youtube.com/@user-yu5qs4ct2b
✅ 영상 주소
[핵심 머신러닝] 불균형 데이터 분석을 위한 샘플링 기법
https://www.youtube.com/watch?v=Vhwz228VrIk&list=PLpIPLT0Pf7IoTxTCi2MEQ94MZnHaxrP0j&index=18
1. Oversampling
- 적은 클래스의 데이터 수를 증가 시키는 방법
- 정보 손실이 없다.
- undersampling보다 높은 정확도
- overfitting 발생우려
1) random oversampling - 적은 클래스의 데이터를 임의로 복제
-> 적은 클래스 분류시 overfitting 발생
2) SMOTE(Synthetic Minority Over-sampling Technique)
- 소수의 클래스와 유사한 가상의 데이터를 생성
- KNN 방식을 통해서 개별 데이터(소수 데이터)의 거리 계산
- X와 X(NN)를 잇는 가상의 라인의 임의의 위치에 가상의 데이터를 생성
3) Borderline SMOTE
- 경계부분만 oversampling
- 소수 클래스의 k개의 주변을 탐색하고, k개의 클래스의 수를 확인
- k개가 모두 다수 클래스가 아니면서 절반 이상은 다수 클래스인 경우 경계선에 위치한다고 판단
- 경계선에 해당하는 관측치만 SMOTE, borderline에만 새로운 데이터 생성
2. Undersampling
- 많은 수의 클래스의 데이터수를 감소 시키는 방법
- 데이터 제거로 진한 정보 손실
1) random undersampling - 많은 클래스의 데이터를 임의로 샘플링해서, 적은 클래스와 유사한 비율
-> 데이터 유실, 샘플로 인해 잘못된 bias 발생 가능성
2) tomek links -> 두 범주 사이를 탐지하고, 정리하여 부정확한 분류경계선 방지
- 두 범주에서 하나씩 포인트를 추출하여, tomek link 형성(수학적 계산을 통해서 경계선에 위치한 클래스가 다른 샘플 선택)
- tomek link를 형성한 샘플중에서 다수 클래스를 제거
- 분류 경계를 확실히 하여, 정보손실의 가능성은 감소
- 제거되는 샘플이 한정적
3) CNN(Condensed Nearest neighbor)
- 다수 범주에서 하나의 샘플을 무작위로 선택, 적은 클래스는 모든 샘플 선택하여 sub-data를 구성
- sub-data를 기준으로 1-NN classification
- 원래는 다수 범주이지만 위 과정을 통해서 적은 클래스로 분류된 샘플들 만 남겨둔다.
- 이 과정을 통해서 경계선에서 먼 데이터들이 삭제
4) One-Sided Selection (OSS)
- tomek link + CNN
- tomek link로 경계선 데이터를 제거하고, CNN에서 경계선에서 멀리 떨어진 데이터 제거
3. 대표적인 oversampling 방법인 SMOTE 연습
✅ dataset - abalone
UC Irvine Machine Learning Repository
https://archive.ics.uci.edu/dataset/1/abalone
1) 준비
# data
raw_file = pd.read_table('./data/abalone.txt', sep=',', header=None)
# feature_names
abal_col = []
with open('./data/abalone_attributes.txt') as f:
for line in f:
abal_col.append(line.strip())
raw_file.columns = abal_col
abal = raw_file
X = abal.iloc[:, 1:]
y = abal['Sex']
np.unique(y, return_counts=True)
(array(['F', 'I', 'M'], dtype=object), array([1307, 1342, 1528], dtype=int64))
- F -> 1307
- I -> 1342
- M -> 1528
2) 간단한 resampling - (random)oversampling / (random)undersampling
a. oversampling
from imblearn.over_sampling import RandomOverSampler
ros = RandomOverSampler()
oversampled_data, oversampled_label = ros.fit_resample(X, y)
oversampled_data = pd.DataFrame(oversampled_data, columns=X.columns)
print(f'원본 데이터의 클래스 비율 {pd.get_dummies(y).sum())}')
print(f'oversampling의 클래스 비율 {pd.get_dummies(oversampled_label).sum())}')
-
원본 데이터의 클래스 비율
F 1307
I 1342
M 1528
dtype: int64 -
oversampling 샘플링 결과
F 1528
I 1528
M 1528
from imblearn.under_sampling import RandomUnderSampler
rus = RandomUnderSampler()
undersampled_data, undersampled_label = rus.fit_resample(X, y)
undersampled_data = pd.DataFrame(undersampled_data, columns=X.columns)
print(f'원본 데이터의 클래스 비율 {pd.get_dummies(y).sum())}')
print(f'undersampling의 클래스 비율 {pd.get_dummies(undersampled_label).sum())}')
Random Under 샘플링 결과
F 1307
I 1307
M 1307
3. SMOTE (oversampling)
✅
abalone 데이터는 class 분류가 균등하게 되어있다.
SOMTE를 연습하기 위해서 abalone dataset을 기반으로 임의의 데이터를 생성
from sklearn.datasets import make_classification
# 1000개의 데이터 샘플
# 5 : 15 : 80 비율
# 2차원 데이터
data, label = make_classification(n_samples=1000, n_features=2, n_informative=2, n_redundant=0, n_repeated=0, n_classes=3,
n_clusters_per_class=1,
weights=[0.05, 0.15, 0.8],
class_sep=0.8, random_state=2019)
a) SMOTE 이전
import matplotlib.pyplot as plt
fig = plt.Figure(figsize=(12,6))
plt.scatter(data[:, 0], data[:, 1], c=label, linewidth=1, edgecolor='black')
plt.show()
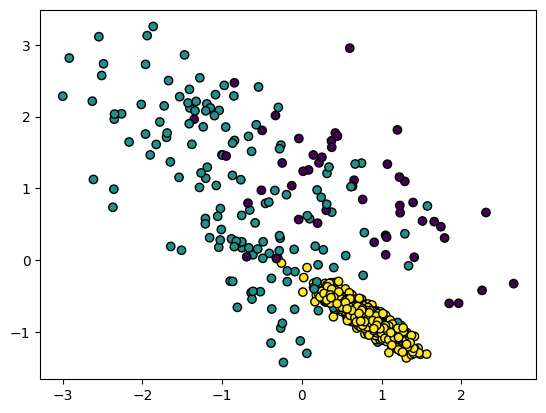
b) SMOTE
from imblearn.over_sampling import SMOTE
## k_neighbors - 측정 k의 갯수
smote = SMOTE(k_neighbors=5)
smoted_data, smoted_label = smote.fit_resample(data, label)
print(f'원본 데이터의 클래스 비율 {pd.get_dummies(label).sum())}')
print(f'undersampling의 클래스 비율 {pd.get_dummies(smote_label).sum())}')
원본 데이터의 클래스 비율
0 53
1 154
2 793
dtype: int64
SMOTE 결과
0 793
1 793
2 793
dtype: int64
✅ 가장 많은 클래스의 수를 기준으로 oversampling
smote 결과
fig = plt.Figure(figsize=(12,6))
plt.scatter(smoted_data[:, 0], smoted_data[:, 1], c=smoted_label, linewidth=1, edgecolor='black')
plt.show()
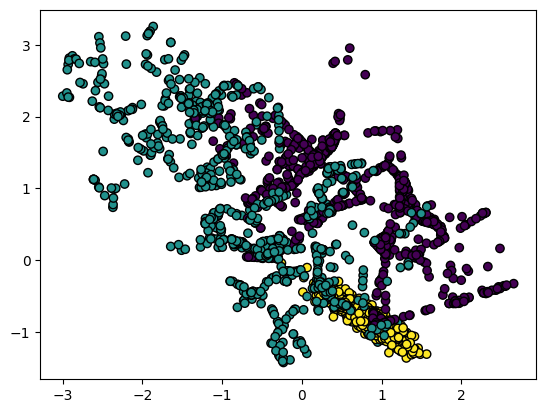
✅ KNN 방식으로 구하고, 그 선 사이위에서 샘플을 추가하는 경향
✅ 이에 따라 추가된 샘플이 선형을 나타내기도 한다.
