
HAR using PCA
HAR data
HAR 데이터 읽기
GitHub에 있는 rawdata를 읽어온다.
- 그리고 column의 이름을 설정한다.
- 라벨값(y) 설정도 진행 및
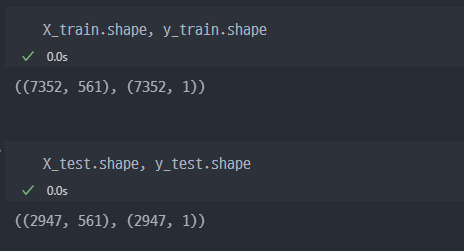
shape확인
PCA 진행
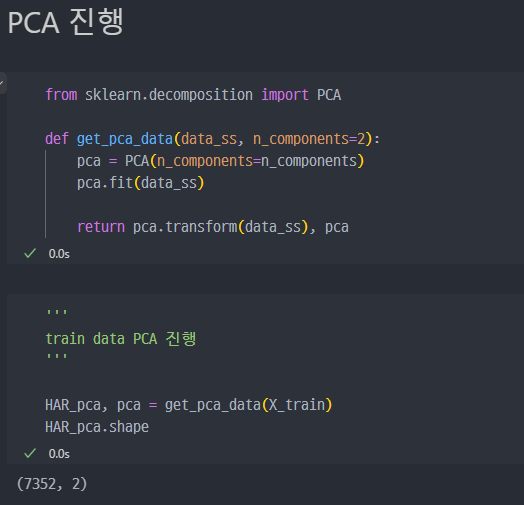
n_components를 2로 설정했으므로pca진행 된 컬럼개수는 2개 (벡터가 2개)
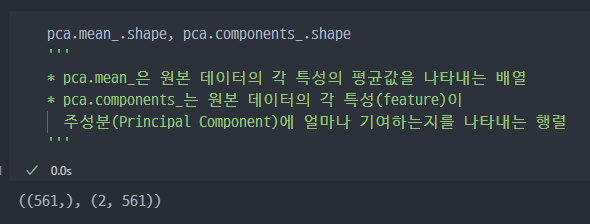
PCA to DataFrame
Pairplot
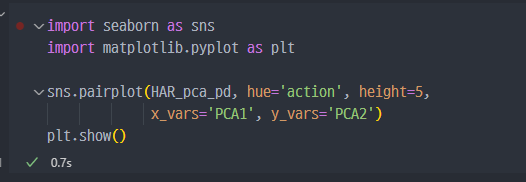
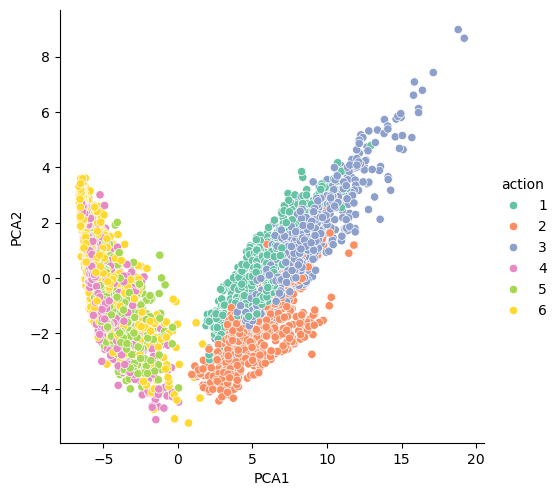
- 그래프로 보았을때는... 생각보다 구분이 되지 않는다..
- 크게 보았을때... 1, 2, 3 데이터와 4, 5, 6 이렇게 두 가지 그룹으로 구분은 되지만 각 그룹에 속한 데이터들끼리의 분리나 구분은 잘 되지 않는다.
- 당초
feature가 561개인데, 이러한datasets의feature를 2개로 줄이니... 구분이 쉽지 않은것 같다.
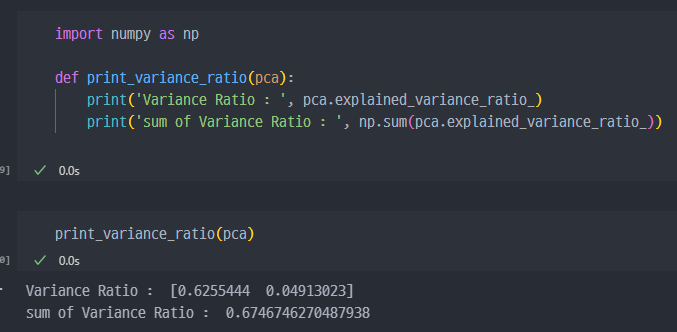
- n_components=2 일때의 variance_ratio
- n_components=3 일때의 variance_ratio

n_components가 2일때는 67%정도였는데, 3개일 때는 71%까지 상승한다.
만약 n_components가 10이면?..
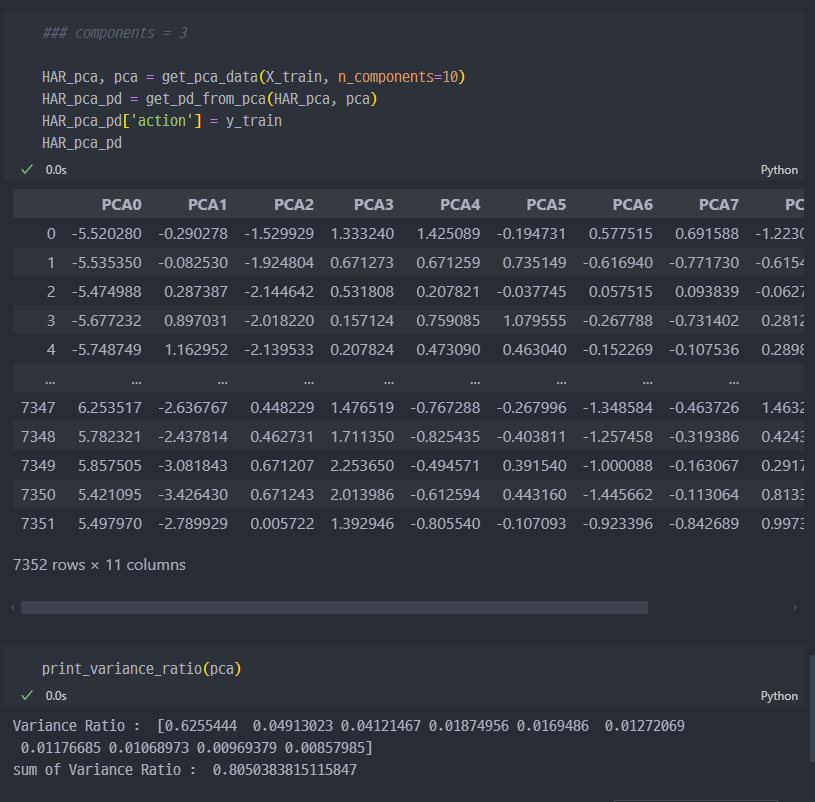
80%까지 상승한다.
다른 방법과 비교
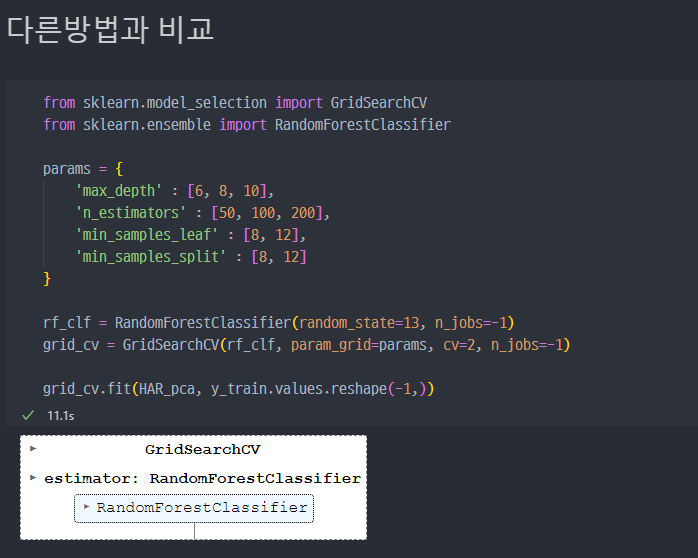
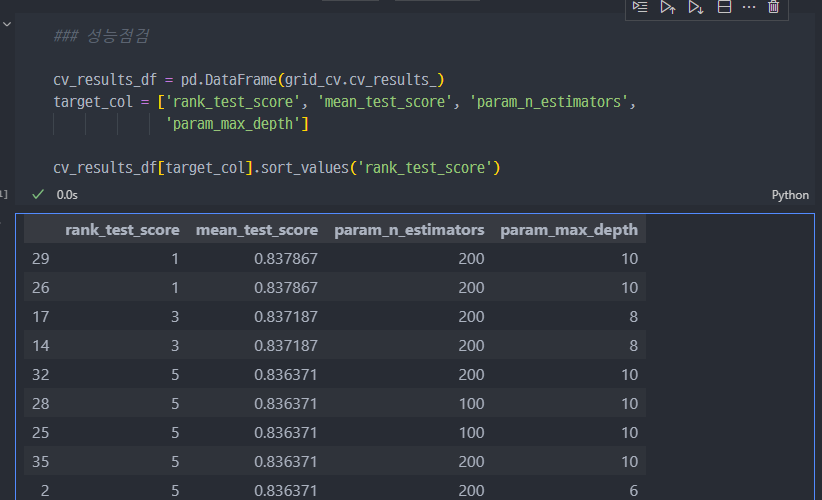
n_estimators를 200으로하고,max_depth를 10으로 했을 때 가장 좋은 성능이며, 약83%정도이다.PCA에서n_components를 10으로하고 계산한 결과와 유사하다. 다만 시간은 훨씬 더 걸린다.
test 데이터에 적용
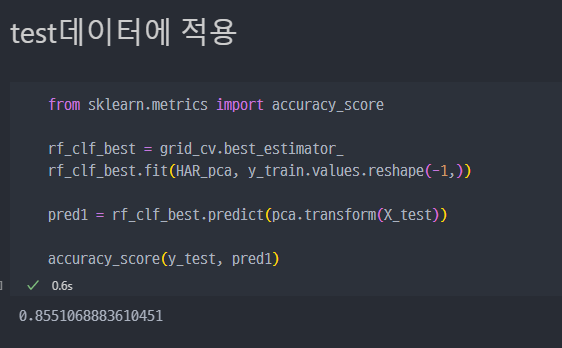
중요!! :
X_test데이터를 사용할 때 별도로fit_transform()을 하거나pca.fit()을 진행하면 안된다.X_train데이터를 이용해fit()즉 학습된pca객체를 그대로 이용해야 한다. 그렇기에X_test데이터는fit()과정이 없고 바로pca.transform()과정만 진행된다.
xgboost로도 결과비교
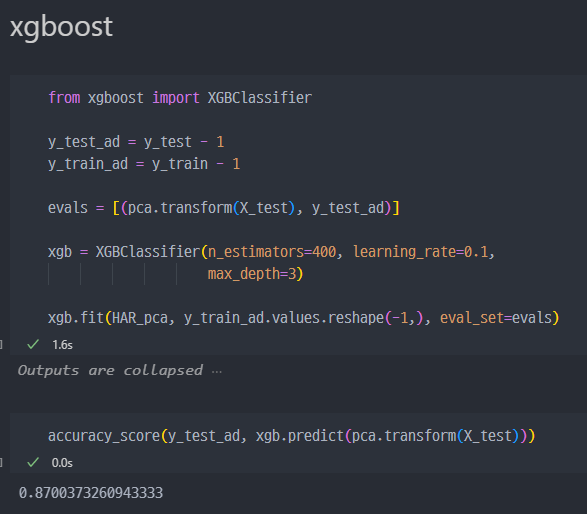
- xgb 진행 시, y_test 및 y_train에서 1씩 빼줘야 오류가 안난다.
XGBoost 모델이 y_train 및 y_test에 포함된 클래스 레이블을 예상과 다른 값으로 인식했기 때문에 발생합니다. XGBClassifier는 클래스 레이블을 0부터 시작하는 정수로 예상합니다. 하지만 현재 y_train 및 y_test에 있는 레이블은 1에서 6까지로 되어 있어 오류가 발생하고 있습니다.
이 문제를 해결하려면 클래스 레이블을 0부터 시작하도록 변경해야 합니다. 즉, 현재 레이블에서 1을 빼주는 방식으로 조정할 수 있습니다.
PCA의 variance_ratio 추가설명
만약 원본 데이터의 전체 변동성을 100%라고 가정해봅시다. PCA를 통해 차원 축소를 하면서 이 변동성의 일부를 각 주성분이 설명하게 됩니다.
- 첫 번째 주성분이 데이터 변동성의 60%를 설명한다면,
explained_variance_ratio_값은 0.6이 됩니다. - 두 번째 주성분이 데이터 변동성의 30%를 설명한다면,
explained_variance_ratio_값은 0.3이 됩니다. - 나머지 주성분들이 합쳐서 나머지 10%를 설명합니다.
이 값들이 합쳐지면 100%가 되며, 이는 전체 데이터 변동성의 비율을 설명합니다.